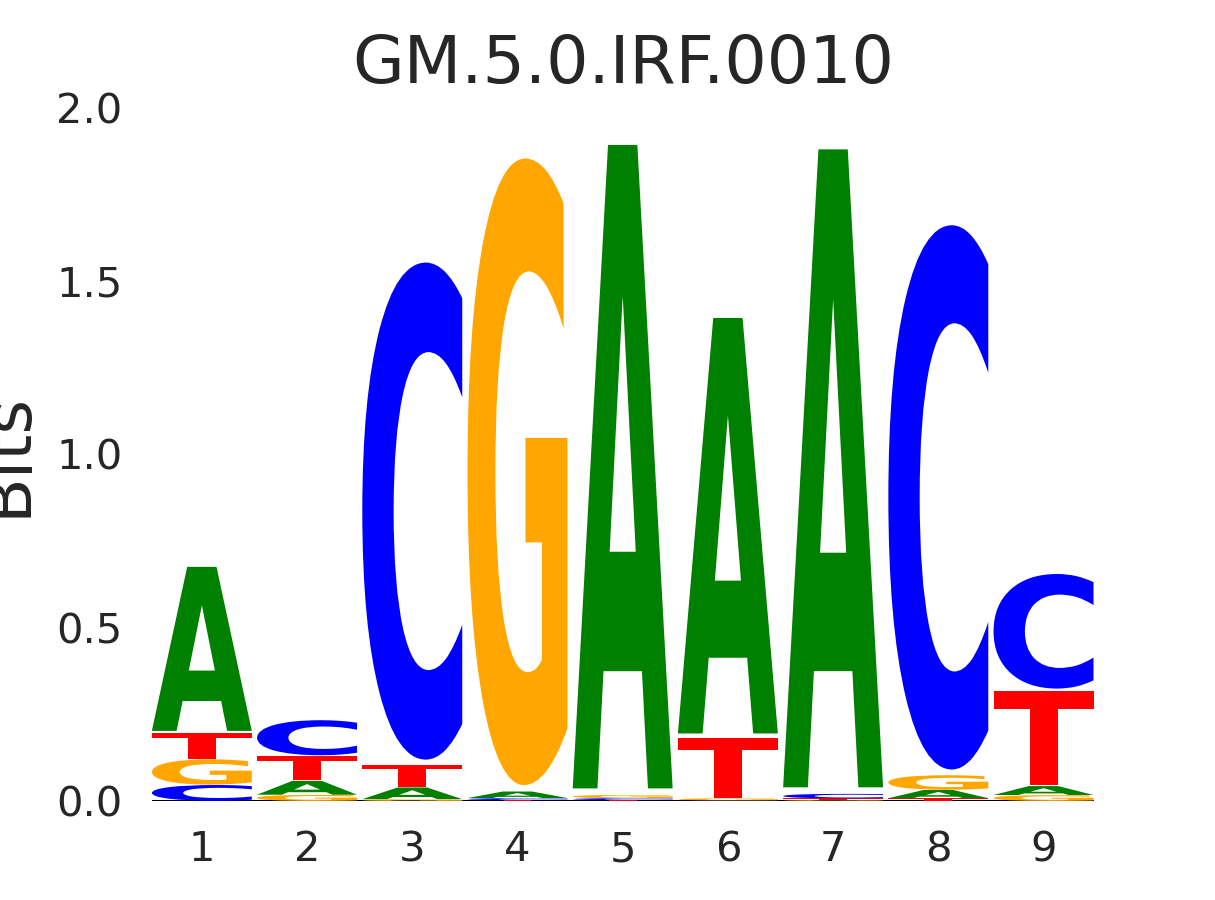
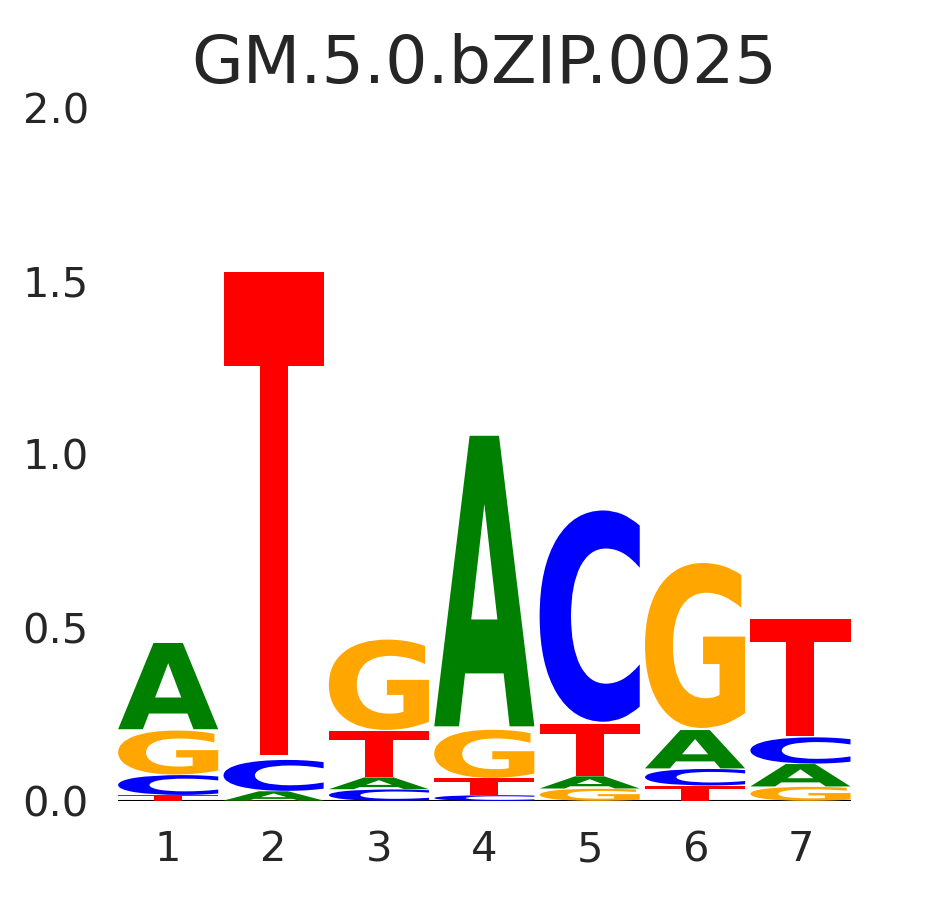
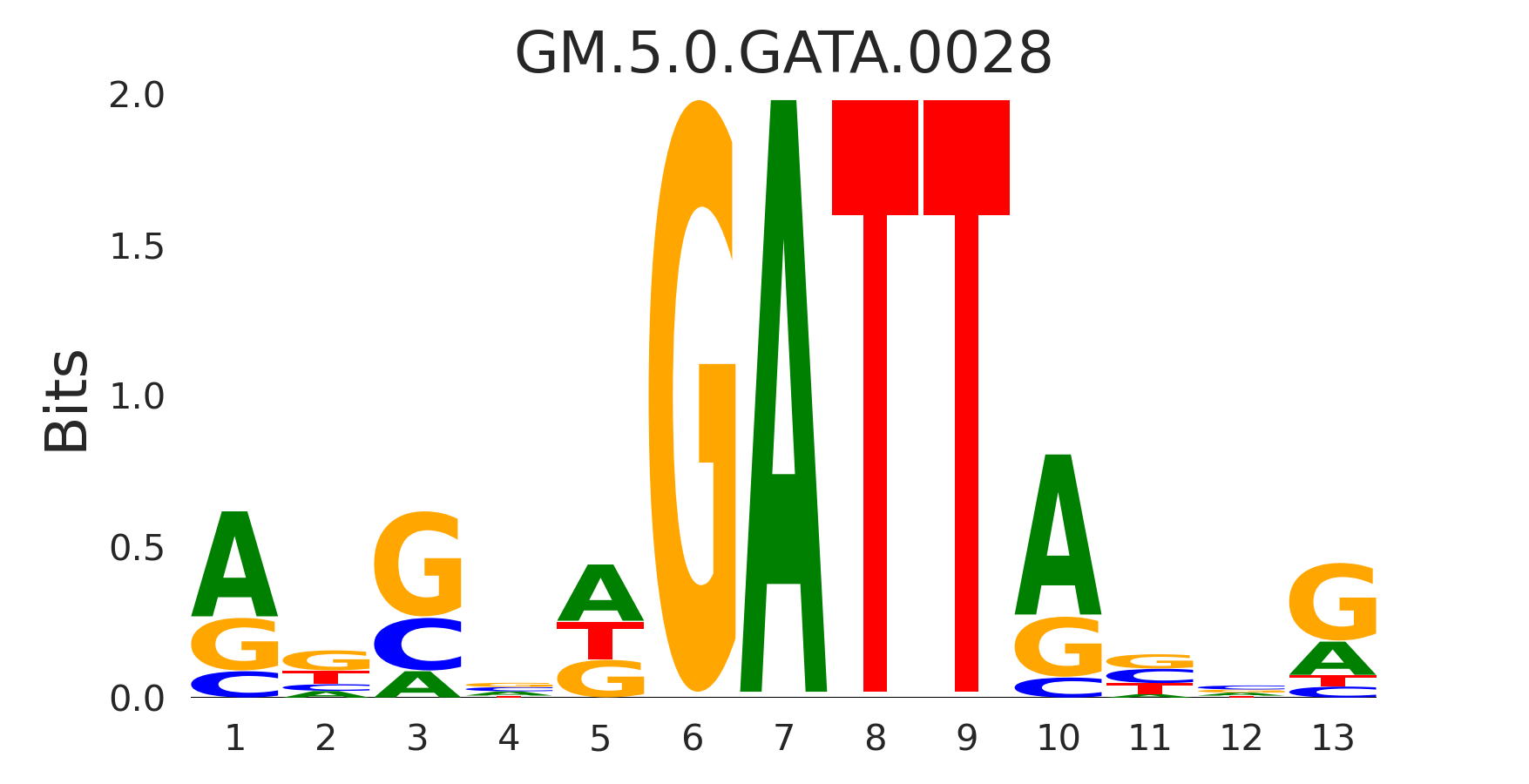
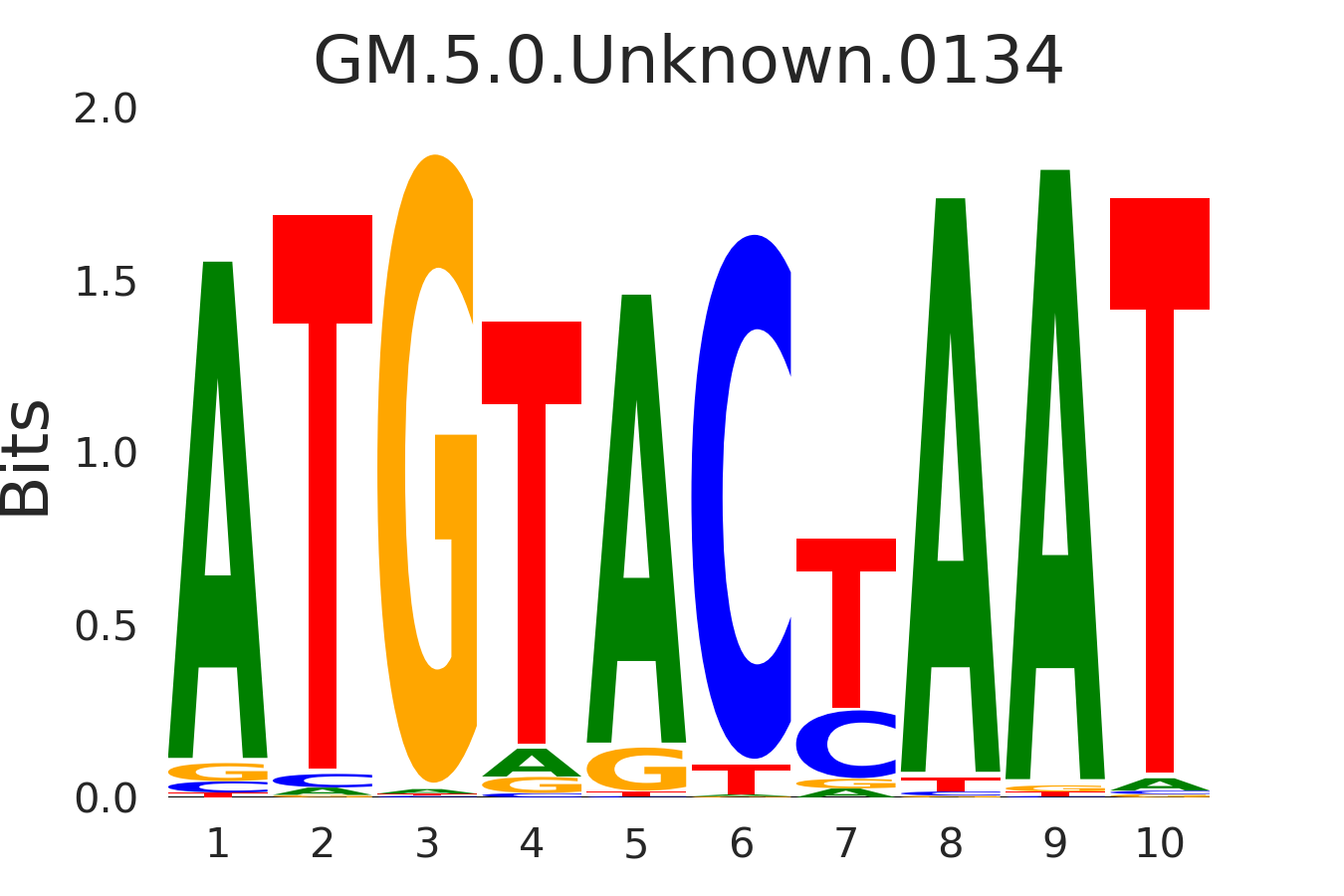
| GM.5.0.Forkhead.0043 |
FOXE3,FOXL2,FOXI1,FOXB2,FOXF2, (...) |
|
-1.51 |
-1.61 |
1.83 |
2.42 |
2.50 |
2.16 |
2.24 |
1.26 |
-1.75 |
-0.84 |
-0.19 |
-0.84 |
-0.18 |
-0.73 |
-1.90 |
0.63 |
1.37 |
0.24 |
1.03 |
-1.40 |
0.85 |
-2.76 |
-3.51 |
-1.56 |
-1.72 |
98 |
0.01 |
0.01 |
0.02 |
0.03 |
0.02 |
0.02 |
0.02 |
0.01 |
0.00 |
-0.00 |
-0.00 |
-0.00 |
-0.00 |
-0.01 |
-0.01 |
-0.01 |
-0.00 |
-0.02 |
-0.02 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.01 |
-0.01 |
| GM.5.0.Pipsqueak.0001 |
CDX4,CDX1,CDX2,LCOR |
|
0.99 |
2.21 |
4.04 |
2.94 |
4.25 |
3.20 |
2.81 |
0.86 |
1.05 |
-2.83 |
-3.52 |
-2.45 |
-1.59 |
-4.84 |
-3.93 |
-3.28 |
-1.87 |
-1.94 |
-2.29 |
-3.16 |
-1.91 |
-3.09 |
-1.39 |
0.08 |
0.55 |
7 |
0.03 |
0.04 |
0.07 |
0.09 |
0.08 |
0.07 |
0.04 |
0.02 |
0.01 |
-0.04 |
-0.05 |
-0.03 |
-0.03 |
-0.05 |
-0.05 |
-0.03 |
-0.01 |
-0.04 |
-0.05 |
-0.05 |
-0.03 |
-0.03 |
-0.02 |
-0.03 |
-0.02 |
| GM.5.0.bZIP.0083 |
CEBPA,CEBPZ |
|
1.85 |
1.08 |
0.12 |
1.61 |
-0.99 |
0.92 |
1.14 |
1.98 |
2.21 |
1.08 |
0.31 |
0.23 |
0.95 |
0.45 |
0.42 |
-1.63 |
-3.13 |
-0.37 |
-1.34 |
-1.36 |
-1.52 |
-1.52 |
-2.97 |
-0.89 |
-1.30 |
2 |
0.06 |
0.04 |
0.03 |
0.05 |
0.03 |
0.04 |
0.02 |
0.03 |
0.03 |
0.02 |
0.01 |
0.02 |
0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.04 |
-0.04 |
-0.04 |
-0.05 |
-0.05 |
-0.05 |
-0.05 |
-0.04 |
-0.04 |
| GM.5.0.Grainyhead.0007 |
UBP1,TFCP2,TFCP2L1 |
|
-1.03 |
0.27 |
-0.25 |
0.82 |
0.16 |
-2.07 |
-2.62 |
-2.97 |
-2.93 |
-3.02 |
-3.24 |
-1.87 |
-1.89 |
-2.88 |
-2.26 |
0.68 |
2.85 |
0.53 |
0.49 |
1.61 |
2.45 |
4.33 |
3.03 |
3.28 |
3.16 |
2 |
-0.05 |
-0.03 |
-0.00 |
0.01 |
0.01 |
-0.02 |
-0.03 |
-0.04 |
-0.05 |
-0.06 |
-0.07 |
-0.04 |
-0.03 |
-0.02 |
-0.01 |
0.03 |
0.03 |
0.03 |
0.03 |
0.06 |
0.07 |
0.06 |
0.04 |
0.05 |
0.03 |
| GM.5.0.bHLH.0075 |
TCF12,HAND1,TCF3,TCF4,HAND2 |
|
1.31 |
2.52 |
2.01 |
2.40 |
2.52 |
2.26 |
1.65 |
0.47 |
0.59 |
-0.06 |
-0.10 |
-2.15 |
-3.07 |
-2.26 |
-2.99 |
-0.23 |
-0.49 |
-0.75 |
-1.63 |
-0.01 |
0.11 |
-0.01 |
0.40 |
-0.78 |
0.33 |
1 |
-0.00 |
0.00 |
0.03 |
0.04 |
0.04 |
0.02 |
-0.00 |
-0.01 |
-0.02 |
-0.04 |
-0.05 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
0.00 |
0.01 |
-0.01 |
-0.02 |
0.01 |
0.03 |
0.01 |
0.02 |
0.01 |
0.00 |
| GM.5.0.C2H2_ZF.0075 |
NO ORTHOLOGS FOUND |
|
3.37 |
3.73 |
2.60 |
3.24 |
3.15 |
2.81 |
3.75 |
-2.51 |
-1.95 |
-2.20 |
-3.82 |
-0.99 |
-0.39 |
-3.49 |
-3.19 |
-2.92 |
-3.03 |
-2.22 |
-3.18 |
-2.04 |
-0.49 |
-0.88 |
1.32 |
-3.33 |
-1.23 |
<1 |
0.05 |
0.05 |
0.05 |
0.07 |
0.06 |
0.05 |
0.03 |
0.01 |
0.00 |
-0.03 |
-0.04 |
-0.02 |
-0.02 |
-0.05 |
-0.04 |
-0.03 |
-0.02 |
-0.04 |
-0.04 |
-0.05 |
-0.03 |
-0.03 |
-0.01 |
-0.04 |
-0.02 |
| GM.5.0.Nuclear_receptor.0117 |
NR5A2,NKX2-4,NR5A1,NKX2-1 |
|
1.19 |
2.73 |
3.05 |
3.36 |
3.24 |
2.51 |
3.52 |
0.28 |
0.08 |
-1.27 |
-2.47 |
1.27 |
-0.34 |
-2.63 |
-2.56 |
-1.89 |
-2.10 |
-2.57 |
-2.51 |
-2.39 |
-2.59 |
-1.41 |
-1.11 |
-2.27 |
-0.84 |
<1 |
-0.01 |
0.01 |
0.04 |
0.06 |
0.05 |
0.03 |
0.01 |
0.00 |
-0.01 |
-0.04 |
-0.05 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.01 |
-0.00 |
-0.02 |
-0.02 |
-0.01 |
0.01 |
-0.00 |
0.00 |
-0.00 |
-0.00 |
| GM.5.0.AT_hook.0002 |
HMGA2 |
|
1.68 |
3.59 |
2.58 |
4.09 |
4.22 |
1.53 |
0.79 |
1.80 |
1.54 |
-2.04 |
-1.87 |
-2.03 |
-2.18 |
-2.67 |
-2.50 |
-2.53 |
0.24 |
-2.46 |
-2.73 |
-3.13 |
-1.85 |
-2.32 |
-0.78 |
-2.76 |
-2.13 |
<1 |
0.02 |
0.03 |
0.06 |
0.08 |
0.07 |
0.05 |
0.02 |
0.04 |
0.03 |
-0.02 |
-0.03 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.03 |
-0.01 |
-0.04 |
-0.04 |
-0.05 |
-0.03 |
-0.04 |
-0.02 |
-0.04 |
-0.04 |
| GM.5.0.bZIP.0075 |
FOS |
|
-1.91 |
-0.69 |
-1.66 |
-0.51 |
0.38 |
-0.76 |
-1.31 |
-1.44 |
-1.19 |
-1.24 |
-1.33 |
0.92 |
0.25 |
0.23 |
0.55 |
1.58 |
0.25 |
2.15 |
1.84 |
3.04 |
2.33 |
0.49 |
1.41 |
1.11 |
0.60 |
1 |
-0.04 |
-0.03 |
-0.00 |
0.01 |
0.00 |
-0.01 |
-0.02 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
0.01 |
0.01 |
0.01 |
0.01 |
0.02 |
0.01 |
0.03 |
0.02 |
0.03 |
0.02 |
0.01 |
-0.00 |
0.01 |
-0.00 |
| GM.5.0.IRF.0012 |
NO ORTHOLOGS FOUND,IRF7 |
|
-2.01 |
-2.27 |
-2.60 |
-0.80 |
0.12 |
0.53 |
-1.77 |
-1.55 |
-2.32 |
-1.93 |
-2.94 |
-3.07 |
-2.71 |
-1.22 |
0.40 |
2.52 |
3.04 |
1.29 |
1.74 |
1.30 |
2.60 |
2.67 |
2.40 |
2.43 |
-0.01 |
<1 |
-0.02 |
-0.02 |
0.00 |
0.04 |
0.03 |
0.03 |
0.01 |
0.01 |
-0.01 |
-0.03 |
-0.05 |
-0.02 |
-0.02 |
-0.01 |
-0.01 |
0.02 |
0.02 |
0.01 |
-0.00 |
0.01 |
0.02 |
0.01 |
0.00 |
0.00 |
-0.01 |
| GM.5.0.C2H2_ZF.0159 |
NO ORTHOLOGS FOUND |
|
0.29 |
1.24 |
0.61 |
1.98 |
1.51 |
3.21 |
2.01 |
0.06 |
-2.04 |
-4.38 |
-3.84 |
-3.58 |
-3.80 |
-1.03 |
-0.57 |
-0.93 |
2.26 |
1.03 |
2.59 |
0.43 |
1.39 |
-0.58 |
2.46 |
0.22 |
0.50 |
1 |
0.00 |
0.00 |
0.04 |
0.05 |
0.05 |
0.04 |
0.01 |
0.00 |
-0.01 |
-0.05 |
-0.06 |
-0.03 |
-0.03 |
-0.02 |
-0.02 |
-0.01 |
0.01 |
-0.00 |
-0.00 |
-0.01 |
0.01 |
-0.01 |
-0.00 |
-0.01 |
-0.01 |
| GM.5.0.Nuclear_receptor.0090 |
NR2E3,NO ORTHOLOGS FOUND |
|
-3.18 |
-2.20 |
0.78 |
-1.57 |
-0.35 |
-0.83 |
-0.84 |
-1.89 |
-2.00 |
-0.18 |
0.83 |
-0.84 |
-0.06 |
0.09 |
-0.63 |
0.44 |
3.00 |
2.80 |
2.85 |
2.94 |
1.95 |
2.41 |
0.72 |
1.79 |
0.35 |
6 |
-0.04 |
-0.03 |
0.00 |
0.00 |
0.00 |
0.00 |
-0.00 |
-0.01 |
-0.02 |
-0.01 |
-0.02 |
-0.01 |
-0.01 |
0.00 |
-0.00 |
0.01 |
0.01 |
0.02 |
0.02 |
0.03 |
0.03 |
0.02 |
0.01 |
0.02 |
0.01 |
| GM.5.0.C2H2_ZF.0079 |
SMC3,ZBTB3,RAD21,CTCF |
|
-0.79 |
-1.15 |
0.67 |
1.47 |
1.84 |
-0.63 |
-0.14 |
-1.79 |
-1.62 |
-2.81 |
-3.14 |
-2.93 |
-2.95 |
-2.03 |
-1.63 |
0.76 |
1.49 |
2.04 |
0.98 |
-0.24 |
1.55 |
2.48 |
3.59 |
0.84 |
0.91 |
3 |
0.01 |
0.01 |
0.03 |
0.04 |
0.04 |
0.02 |
0.00 |
-0.01 |
-0.01 |
-0.03 |
-0.04 |
-0.03 |
-0.03 |
-0.03 |
-0.02 |
-0.01 |
-0.00 |
-0.01 |
-0.01 |
-0.01 |
0.00 |
0.00 |
0.01 |
-0.00 |
0.00 |
| GM.5.0.MADS_box.0017 |
RPLP0 |
|
2.18 |
2.25 |
2.63 |
2.13 |
1.51 |
1.36 |
1.53 |
-0.96 |
-0.48 |
-2.13 |
-0.48 |
-1.07 |
-1.43 |
-1.53 |
-1.87 |
-3.00 |
-0.62 |
-1.23 |
-0.83 |
-2.39 |
-2.52 |
-0.60 |
0.77 |
0.30 |
0.24 |
1 |
0.04 |
0.04 |
0.05 |
0.06 |
0.04 |
0.04 |
0.02 |
0.01 |
0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.03 |
-0.04 |
-0.03 |
-0.01 |
-0.04 |
-0.04 |
-0.05 |
-0.04 |
-0.03 |
-0.01 |
-0.03 |
-0.02 |
| GM.5.0.Mixed.0020 |
BATF3,IRF8,IRF10,MYT1L,BATF, (...) |
|
1.07 |
1.58 |
0.48 |
2.93 |
3.49 |
2.64 |
1.72 |
1.20 |
0.61 |
-2.72 |
-1.91 |
-2.64 |
-1.58 |
-1.42 |
-0.41 |
-2.23 |
0.39 |
-2.26 |
-2.06 |
-2.57 |
-1.00 |
-3.08 |
0.40 |
-0.34 |
0.24 |
1 |
0.01 |
0.01 |
0.02 |
0.05 |
0.04 |
0.04 |
0.01 |
0.00 |
-0.00 |
-0.04 |
-0.05 |
-0.03 |
-0.03 |
-0.03 |
-0.02 |
-0.01 |
0.00 |
-0.02 |
-0.02 |
-0.00 |
0.01 |
-0.01 |
0.00 |
-0.00 |
0.00 |
| GM.5.0.C2H2_ZF.0239 |
IKZF1,IKZF3,IKZF2 |
|
-2.97 |
-1.38 |
-1.06 |
-1.49 |
-0.31 |
-0.31 |
-0.73 |
-3.82 |
-2.98 |
-3.62 |
-2.59 |
-1.83 |
-1.57 |
0.86 |
2.19 |
2.11 |
3.08 |
1.93 |
2.25 |
3.37 |
3.10 |
3.21 |
2.28 |
2.92 |
3.25 |
<1 |
-0.04 |
-0.03 |
-0.01 |
0.00 |
0.00 |
-0.00 |
-0.01 |
-0.03 |
-0.03 |
-0.05 |
-0.05 |
-0.03 |
-0.03 |
-0.01 |
0.00 |
0.02 |
0.02 |
0.02 |
0.02 |
0.05 |
0.05 |
0.03 |
0.02 |
0.04 |
0.03 |
| GM.5.0.Unknown.0004 |
NFIX,NFIC,NFIA,FOXA2,NFIB, (...) |
|
1.06 |
3.31 |
1.02 |
1.49 |
1.40 |
1.37 |
0.80 |
-0.74 |
-0.72 |
-2.85 |
-0.28 |
-2.41 |
-0.73 |
-1.39 |
-0.39 |
-0.78 |
-1.16 |
-0.96 |
-1.66 |
-0.98 |
0.72 |
-0.53 |
1.14 |
0.32 |
0.09 |
<1 |
0.00 |
0.02 |
0.04 |
0.06 |
0.04 |
0.03 |
0.01 |
-0.01 |
-0.01 |
-0.04 |
-0.04 |
-0.04 |
-0.03 |
-0.03 |
-0.03 |
-0.01 |
-0.00 |
-0.02 |
-0.02 |
-0.01 |
0.01 |
-0.00 |
0.01 |
-0.00 |
0.00 |
| GM.5.0.Mixed.0015 |
TAF1,TCF12,GABPA,TCF3,TCF4, (...) |
|
-0.10 |
-0.37 |
-1.32 |
-0.33 |
0.50 |
1.37 |
1.92 |
-0.30 |
-0.31 |
-2.04 |
-1.20 |
-3.68 |
-2.47 |
0.23 |
-0.66 |
0.46 |
0.52 |
1.03 |
0.41 |
1.69 |
1.11 |
1.89 |
1.10 |
0.67 |
0.89 |
1 |
-0.02 |
-0.02 |
0.00 |
0.03 |
0.02 |
0.03 |
0.02 |
0.00 |
-0.01 |
-0.05 |
-0.06 |
-0.04 |
-0.03 |
-0.01 |
-0.01 |
0.01 |
0.02 |
0.01 |
0.00 |
0.02 |
0.03 |
0.02 |
0.02 |
0.01 |
-0.01 |
| GM.5.0.C2H2_ZF.0317 |
FEZF2,FEZF1 |
|
0.58 |
1.59 |
1.24 |
0.92 |
1.81 |
2.12 |
3.20 |
-0.08 |
-1.35 |
2.46 |
1.43 |
-0.24 |
-0.67 |
-2.91 |
-1.95 |
-1.41 |
-0.92 |
-0.26 |
-1.22 |
-1.80 |
-1.65 |
-1.60 |
-0.86 |
-2.69 |
-2.12 |
2 |
0.01 |
0.01 |
0.02 |
0.05 |
0.03 |
0.03 |
0.01 |
0.02 |
0.01 |
0.02 |
0.00 |
0.01 |
0.01 |
-0.01 |
-0.02 |
-0.01 |
-0.02 |
-0.01 |
-0.02 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
| GM.5.0.C2H2_ZF_Homeodomain.0003 |
ZEB1,ZEB2,NO ORTHOLOGS FOUND |
|
-3.60 |
-3.33 |
-2.00 |
-2.33 |
-2.06 |
-1.68 |
-2.32 |
-0.92 |
0.05 |
1.71 |
1.98 |
2.44 |
2.24 |
0.67 |
-0.01 |
2.66 |
1.15 |
3.42 |
3.21 |
2.80 |
-0.32 |
2.03 |
-1.46 |
1.21 |
1.09 |
16 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.03 |
-0.01 |
-0.01 |
0.00 |
0.01 |
0.02 |
0.03 |
0.03 |
0.02 |
0.02 |
0.01 |
0.02 |
0.00 |
0.03 |
0.03 |
0.02 |
-0.00 |
0.01 |
-0.01 |
0.00 |
-0.01 |
| GM.5.0.bHLH.0100 |
MLXIPL |
|
0.93 |
2.38 |
1.37 |
2.23 |
2.23 |
2.87 |
2.91 |
0.14 |
0.65 |
-0.16 |
-1.57 |
-2.15 |
-2.03 |
-1.99 |
-0.54 |
-3.09 |
0.35 |
-2.74 |
-3.43 |
-2.53 |
-2.35 |
-2.00 |
-0.34 |
-3.10 |
-0.24 |
2 |
0.03 |
0.03 |
0.04 |
0.06 |
0.05 |
0.04 |
0.02 |
0.02 |
0.02 |
-0.01 |
-0.02 |
-0.01 |
-0.01 |
-0.03 |
-0.03 |
-0.03 |
-0.01 |
-0.04 |
-0.04 |
-0.05 |
-0.03 |
-0.03 |
-0.02 |
-0.03 |
-0.02 |
| GM.5.0.TEA.0002 |
TEAD1,TEAD4 |
|
-0.52 |
0.93 |
-0.01 |
-0.29 |
-1.12 |
-0.65 |
-0.39 |
-2.43 |
-2.93 |
-3.04 |
-3.96 |
-1.60 |
-1.96 |
-1.28 |
0.38 |
1.34 |
3.41 |
1.48 |
1.50 |
2.39 |
2.14 |
3.09 |
3.09 |
0.94 |
0.91 |
<1 |
-0.02 |
-0.01 |
0.02 |
0.03 |
0.03 |
0.01 |
-0.00 |
-0.03 |
-0.04 |
-0.06 |
-0.08 |
-0.04 |
-0.04 |
-0.02 |
-0.02 |
0.01 |
0.02 |
0.02 |
0.01 |
0.04 |
0.05 |
0.02 |
0.03 |
0.02 |
0.01 |
| GM.5.0.C2H2_ZF.0161 |
NO ORTHOLOGS FOUND |
|
-0.08 |
1.46 |
-1.05 |
-0.60 |
-0.03 |
-1.72 |
-3.03 |
-0.49 |
-1.01 |
1.50 |
0.47 |
0.20 |
1.02 |
0.02 |
-0.41 |
0.18 |
-0.64 |
-1.33 |
-1.03 |
-0.93 |
-1.77 |
0.16 |
1.08 |
1.18 |
1.20 |
1 |
-0.03 |
-0.02 |
-0.00 |
0.01 |
0.01 |
-0.01 |
-0.02 |
0.01 |
0.01 |
0.02 |
0.01 |
0.02 |
0.02 |
0.02 |
0.01 |
0.01 |
-0.00 |
0.01 |
0.01 |
-0.00 |
-0.00 |
0.00 |
-0.01 |
0.00 |
-0.01 |
| GM.5.0.HSF.0010 |
HSF1 |
|
-0.52 |
-0.13 |
1.07 |
1.06 |
0.22 |
0.00 |
-1.16 |
-3.12 |
-3.08 |
-2.99 |
-3.28 |
-1.28 |
-1.31 |
-1.18 |
-0.43 |
-0.23 |
-0.09 |
-0.13 |
-0.63 |
1.42 |
2.71 |
2.29 |
1.77 |
1.91 |
1.55 |
<1 |
-0.04 |
-0.02 |
0.02 |
0.03 |
0.02 |
0.01 |
-0.01 |
-0.03 |
-0.04 |
-0.05 |
-0.07 |
-0.03 |
-0.02 |
-0.01 |
-0.01 |
0.01 |
0.02 |
0.02 |
0.01 |
0.04 |
0.05 |
0.03 |
0.02 |
0.03 |
0.01 |
| GM.5.0.C2H2_ZF.0171 |
NO ORTHOLOGS FOUND |
|
0.72 |
2.16 |
3.52 |
2.79 |
2.98 |
2.19 |
2.06 |
-0.02 |
-0.91 |
-1.56 |
0.62 |
-2.34 |
-1.34 |
-2.72 |
-2.51 |
-1.73 |
-1.14 |
-0.19 |
0.29 |
-1.89 |
-0.97 |
-1.69 |
-0.72 |
0.33 |
-0.10 |
<1 |
0.03 |
0.04 |
0.04 |
0.04 |
0.04 |
0.03 |
0.03 |
0.00 |
-0.00 |
-0.02 |
-0.01 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.03 |
-0.01 |
-0.02 |
-0.02 |
-0.03 |
-0.02 |
-0.02 |
-0.01 |
-0.01 |
-0.01 |
| GM.5.0.HSF.0005 |
HSF1,HSF2,HSF4 |
|
-2.38 |
-1.77 |
-0.92 |
0.10 |
-0.14 |
-1.00 |
-3.65 |
-3.33 |
-2.62 |
-2.27 |
-2.66 |
-2.04 |
-1.08 |
0.69 |
-0.67 |
1.71 |
3.24 |
2.17 |
2.39 |
1.21 |
2.48 |
1.47 |
2.71 |
1.86 |
3.90 |
<1 |
-0.04 |
-0.03 |
0.01 |
0.02 |
0.02 |
-0.00 |
-0.02 |
-0.03 |
-0.03 |
-0.05 |
-0.06 |
-0.04 |
-0.03 |
-0.02 |
-0.01 |
0.02 |
0.02 |
0.03 |
0.02 |
0.04 |
0.05 |
0.03 |
0.03 |
0.04 |
0.03 |
| GM.5.0.C2H2_ZF.0086 |
NO ORTHOLOGS FOUND |
|
2.89 |
2.54 |
1.95 |
1.44 |
1.02 |
4.11 |
3.97 |
1.73 |
2.06 |
-1.43 |
-0.20 |
-3.79 |
-3.70 |
-2.24 |
-3.05 |
-2.49 |
-2.35 |
-2.34 |
-2.32 |
-2.98 |
-2.27 |
-2.33 |
-0.94 |
-1.49 |
0.97 |
1 |
0.06 |
0.04 |
0.03 |
0.05 |
0.04 |
0.05 |
0.04 |
0.02 |
0.01 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.04 |
-0.04 |
-0.03 |
-0.01 |
-0.04 |
-0.04 |
-0.04 |
-0.02 |
-0.02 |
-0.01 |
-0.02 |
-0.01 |
| GM.5.0.T-box.0025 |
TBXT.2,TBXT |
|
2.96 |
2.03 |
0.45 |
2.55 |
0.52 |
1.62 |
2.53 |
3.42 |
2.67 |
2.91 |
3.16 |
1.03 |
1.78 |
-0.22 |
-1.98 |
-2.50 |
-3.33 |
-3.51 |
-2.20 |
-4.10 |
-3.49 |
-3.07 |
-2.91 |
-3.25 |
-2.01 |
2 |
0.07 |
0.05 |
0.03 |
0.05 |
0.04 |
0.04 |
0.03 |
0.05 |
0.04 |
0.03 |
0.03 |
0.02 |
0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.04 |
-0.06 |
-0.05 |
-0.07 |
-0.06 |
-0.06 |
-0.05 |
-0.06 |
-0.04 |
| GM.5.0.C2H2_ZF.0056 |
HIC1,HIC2,THAP1,LOC100493491,THAP2 |
|
4.13 |
4.96 |
4.96 |
4.59 |
5.49 |
4.35 |
4.35 |
1.76 |
1.89 |
-2.11 |
-2.01 |
-3.94 |
-3.11 |
-4.63 |
-4.90 |
-4.45 |
-3.44 |
-4.20 |
-3.57 |
-3.59 |
-2.79 |
-4.56 |
-2.68 |
-4.20 |
-1.47 |
1 |
0.07 |
0.08 |
0.09 |
0.11 |
0.10 |
0.06 |
0.05 |
0.03 |
0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.03 |
-0.06 |
-0.06 |
-0.06 |
-0.03 |
-0.08 |
-0.07 |
-0.09 |
-0.06 |
-0.06 |
-0.03 |
-0.05 |
-0.03 |
| GM.5.0.C2H2_ZF.0036 |
SP1,JUND,SP2 |
|
-1.07 |
-1.55 |
-0.50 |
-1.08 |
-1.11 |
-0.65 |
-0.85 |
-0.43 |
-2.23 |
-2.23 |
-2.70 |
-1.97 |
-2.90 |
-0.23 |
1.40 |
2.34 |
3.27 |
2.14 |
1.38 |
3.13 |
2.46 |
2.44 |
3.25 |
2.83 |
0.82 |
1 |
-0.04 |
-0.03 |
-0.00 |
0.01 |
0.01 |
0.00 |
-0.01 |
-0.01 |
-0.02 |
-0.05 |
-0.06 |
-0.03 |
-0.03 |
-0.01 |
-0.00 |
0.03 |
0.03 |
0.02 |
0.01 |
0.04 |
0.05 |
0.03 |
0.03 |
0.03 |
0.01 |
| GM.5.0.Unknown.0141 |
ITGB2 |
|
0.41 |
0.53 |
0.59 |
1.33 |
0.65 |
0.90 |
-0.72 |
1.57 |
2.00 |
1.43 |
0.84 |
1.90 |
0.50 |
1.41 |
1.18 |
0.90 |
-0.64 |
-0.22 |
-0.48 |
-1.56 |
-1.13 |
-0.11 |
-0.02 |
-1.73 |
-3.14 |
<1 |
-0.01 |
-0.00 |
0.03 |
0.04 |
0.03 |
0.02 |
0.00 |
0.02 |
0.02 |
0.02 |
0.01 |
0.02 |
0.01 |
0.00 |
-0.01 |
-0.00 |
-0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.03 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
| GM.5.0.C2H2_ZF.0216 |
NO ORTHOLOGS FOUND |
|
0.66 |
1.65 |
2.59 |
1.23 |
3.39 |
2.21 |
0.40 |
-1.54 |
-2.04 |
-2.74 |
-3.14 |
-3.32 |
-3.24 |
-2.84 |
-1.28 |
-0.31 |
1.40 |
-1.73 |
-0.71 |
-0.50 |
1.04 |
1.99 |
2.25 |
1.84 |
2.16 |
<1 |
-0.03 |
-0.01 |
0.01 |
0.02 |
0.03 |
0.01 |
-0.01 |
-0.03 |
-0.04 |
-0.06 |
-0.07 |
-0.05 |
-0.05 |
-0.03 |
-0.02 |
0.01 |
0.03 |
0.01 |
0.01 |
0.03 |
0.05 |
0.03 |
0.04 |
0.03 |
0.02 |
| GM.5.0.Unknown.0138 |
BHLHE41,PTEN |
|
3.33 |
3.02 |
4.26 |
2.79 |
3.73 |
2.53 |
1.99 |
1.50 |
0.43 |
0.28 |
-0.14 |
-1.02 |
-2.58 |
-3.55 |
-3.78 |
-3.11 |
-1.35 |
-4.25 |
-3.80 |
-3.13 |
-3.25 |
-1.74 |
-1.58 |
-1.70 |
-1.09 |
1 |
0.05 |
0.04 |
0.05 |
0.06 |
0.05 |
0.04 |
0.02 |
0.02 |
0.01 |
-0.01 |
-0.02 |
-0.01 |
-0.01 |
-0.03 |
-0.03 |
-0.03 |
-0.02 |
-0.05 |
-0.05 |
-0.05 |
-0.03 |
-0.03 |
-0.02 |
-0.03 |
-0.02 |
| GM.5.0.Nuclear_receptor.0146 |
ESR1,ESR2,NO ORTHOLOGS FOUND |
|
-3.05 |
-2.91 |
-3.58 |
-3.17 |
-1.93 |
-3.37 |
-2.51 |
-2.78 |
-2.52 |
-4.07 |
-3.22 |
-2.71 |
-3.49 |
0.61 |
2.30 |
1.71 |
2.56 |
2.79 |
2.50 |
4.37 |
3.23 |
3.76 |
4.03 |
4.11 |
4.53 |
<1 |
-0.06 |
-0.04 |
-0.02 |
-0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.02 |
-0.02 |
-0.03 |
-0.04 |
-0.02 |
-0.01 |
0.01 |
0.01 |
0.02 |
0.02 |
0.04 |
0.03 |
0.06 |
0.05 |
0.04 |
0.02 |
0.04 |
0.03 |
| GM.5.0.Grainyhead.0008 |
UBP1,TFCP2,TFCP2L1 |
|
-1.61 |
0.78 |
2.74 |
-0.05 |
1.94 |
2.14 |
1.35 |
-3.29 |
-2.34 |
-2.16 |
-2.61 |
-2.87 |
-2.67 |
-1.37 |
-2.23 |
-2.43 |
0.03 |
-2.15 |
-1.63 |
1.34 |
1.34 |
2.80 |
2.56 |
1.56 |
2.29 |
1 |
-0.01 |
-0.00 |
0.02 |
0.02 |
0.02 |
0.02 |
0.01 |
-0.02 |
-0.03 |
-0.05 |
-0.05 |
-0.04 |
-0.04 |
-0.03 |
-0.02 |
-0.00 |
0.01 |
-0.00 |
-0.00 |
0.03 |
0.04 |
0.03 |
0.03 |
0.02 |
0.01 |
| GM.5.0.GATA.0016 |
GATA1,GATA2,GATA3 |
|
-3.42 |
-3.64 |
0.89 |
0.14 |
-0.10 |
0.23 |
-0.86 |
2.19 |
1.22 |
1.57 |
0.41 |
0.88 |
1.67 |
2.79 |
2.34 |
1.13 |
0.39 |
1.78 |
1.62 |
2.48 |
1.32 |
0.93 |
-0.77 |
-0.43 |
0.84 |
2 |
-0.10 |
-0.08 |
-0.03 |
-0.02 |
-0.02 |
-0.02 |
-0.03 |
-0.00 |
-0.00 |
0.01 |
0.01 |
0.03 |
0.03 |
0.05 |
0.04 |
0.04 |
0.02 |
0.04 |
0.03 |
0.05 |
0.04 |
0.02 |
0.01 |
0.02 |
0.00 |
| GM.5.0.Unknown.0145 |
SRF,NO ORTHOLOGS FOUND |
|
0.54 |
2.45 |
2.93 |
2.41 |
2.30 |
2.07 |
1.08 |
1.71 |
0.70 |
0.60 |
-0.22 |
-0.96 |
-0.72 |
-1.50 |
-1.23 |
-3.52 |
-0.69 |
-2.36 |
-1.54 |
-0.82 |
-1.11 |
-1.67 |
-0.84 |
-3.43 |
-2.83 |
<1 |
0.02 |
0.02 |
0.06 |
0.07 |
0.06 |
0.05 |
0.02 |
0.03 |
0.02 |
-0.01 |
-0.02 |
-0.01 |
-0.01 |
-0.03 |
-0.03 |
-0.03 |
-0.01 |
-0.04 |
-0.04 |
-0.04 |
-0.03 |
-0.03 |
-0.02 |
-0.04 |
-0.03 |
| GM.5.0.Grainyhead.0005 |
UBP1,TFCP2,TFCP2L1 |
|
2.21 |
2.91 |
1.63 |
1.79 |
1.80 |
1.68 |
0.72 |
-2.79 |
-1.59 |
-3.32 |
-2.54 |
-3.56 |
-3.61 |
-1.58 |
-2.56 |
-0.18 |
1.08 |
-0.33 |
-0.21 |
-0.79 |
0.91 |
1.17 |
1.07 |
1.48 |
2.14 |
19 |
-0.02 |
-0.00 |
0.03 |
0.03 |
0.03 |
0.01 |
-0.00 |
-0.02 |
-0.02 |
-0.04 |
-0.04 |
-0.03 |
-0.03 |
-0.01 |
-0.01 |
0.00 |
0.01 |
-0.00 |
-0.00 |
0.01 |
0.02 |
0.01 |
0.01 |
0.01 |
0.01 |
| GM.5.0.STAT.0013 |
STAT1,STAT3.2,STAT2,STAT3.1,STAT4 |
|
-0.84 |
-1.90 |
-1.91 |
-2.02 |
-1.65 |
-1.30 |
-0.60 |
-1.94 |
0.43 |
2.39 |
1.98 |
1.18 |
3.02 |
0.82 |
1.13 |
1.35 |
-2.43 |
0.59 |
1.59 |
1.06 |
0.26 |
1.46 |
0.63 |
0.80 |
-0.15 |
1 |
-0.05 |
-0.04 |
-0.02 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
0.01 |
0.01 |
0.02 |
0.02 |
0.03 |
0.04 |
0.03 |
0.02 |
0.02 |
-0.01 |
0.02 |
0.02 |
0.01 |
0.00 |
0.01 |
-0.01 |
0.00 |
-0.01 |
| GM.5.0.Unknown.0176 |
MBD3,MBD2,MECP2,NO ORTHOLOGS FOUND |
|
1.21 |
0.92 |
1.86 |
1.55 |
0.97 |
1.57 |
1.56 |
2.12 |
1.39 |
-1.26 |
0.77 |
0.64 |
1.66 |
-1.98 |
-2.93 |
-2.10 |
-3.25 |
-1.49 |
-2.20 |
-1.83 |
-2.00 |
0.10 |
0.63 |
-1.04 |
-1.43 |
23 |
-0.02 |
-0.02 |
0.01 |
0.03 |
0.02 |
0.03 |
0.01 |
0.03 |
0.02 |
-0.01 |
-0.02 |
0.01 |
0.01 |
-0.01 |
-0.01 |
0.01 |
-0.00 |
0.01 |
0.00 |
0.00 |
0.00 |
-0.00 |
-0.01 |
-0.01 |
-0.03 |
| GM.5.0.Homeodomain.0085 |
SIX1,SIX4,SIX2 |
|
-2.05 |
-0.70 |
-2.31 |
-2.43 |
-2.18 |
-1.94 |
-3.15 |
-2.87 |
-2.11 |
-1.27 |
-1.49 |
-1.21 |
-1.06 |
-0.04 |
0.99 |
1.93 |
0.50 |
1.81 |
1.85 |
2.85 |
2.89 |
3.86 |
3.30 |
2.66 |
1.95 |
<1 |
-0.06 |
-0.05 |
-0.02 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.01 |
-0.01 |
-0.02 |
-0.04 |
-0.01 |
-0.00 |
0.01 |
0.01 |
0.04 |
0.02 |
0.04 |
0.03 |
0.05 |
0.05 |
0.04 |
0.02 |
0.02 |
0.01 |
| GM.5.0.C2H2_ZF.0154 |
NO ORTHOLOGS FOUND |
|
0.43 |
0.85 |
1.61 |
0.15 |
1.43 |
0.99 |
0.81 |
-1.48 |
-2.49 |
-0.46 |
-0.35 |
-0.96 |
0.13 |
-0.31 |
-1.35 |
-3.30 |
-1.29 |
0.32 |
-0.86 |
-1.57 |
-1.07 |
0.85 |
-1.16 |
-0.47 |
0.92 |
<1 |
-0.01 |
0.00 |
0.02 |
0.02 |
0.02 |
0.02 |
0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.00 |
-0.00 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
0.00 |
0.00 |
-0.00 |
-0.00 |
-0.00 |
| GM.5.0.Mixed.0064 |
NKX2-5,NKX2-3,NKX2-6,NKX3-1,POU3F2, (...) |
|
-1.18 |
-0.48 |
-1.00 |
-1.22 |
-1.09 |
-0.60 |
0.32 |
-1.23 |
0.64 |
-0.05 |
0.56 |
0.35 |
1.89 |
-2.30 |
0.91 |
1.88 |
4.04 |
2.11 |
1.33 |
0.44 |
0.70 |
0.65 |
-0.09 |
0.61 |
0.10 |
1 |
-0.01 |
-0.01 |
0.02 |
0.03 |
0.02 |
0.02 |
0.01 |
0.03 |
0.04 |
0.02 |
0.01 |
0.02 |
0.02 |
-0.00 |
-0.01 |
0.00 |
0.00 |
-0.01 |
-0.01 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
-0.04 |
-0.04 |
| GM.5.0.C2H2_ZF.0284 |
RAD21,CTCF,REST,ESRRG,ESRRA, (...) |
|
-0.80 |
-1.65 |
-0.65 |
-1.21 |
-1.53 |
-0.82 |
-2.09 |
-2.12 |
-1.32 |
1.33 |
1.16 |
1.20 |
0.58 |
1.20 |
1.51 |
3.13 |
1.78 |
1.89 |
2.33 |
0.68 |
-1.59 |
2.01 |
1.91 |
2.05 |
1.75 |
2 |
-0.05 |
-0.05 |
0.00 |
0.01 |
0.01 |
0.01 |
-0.01 |
0.02 |
0.02 |
0.02 |
0.01 |
0.03 |
0.03 |
0.03 |
0.03 |
0.03 |
0.00 |
0.02 |
0.02 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.03 |
| GM.5.0.Unknown.0171 |
PAXIP1 |
|
-0.37 |
0.59 |
-0.42 |
-1.83 |
-1.36 |
-1.47 |
-0.28 |
-2.45 |
-1.94 |
0.96 |
-0.02 |
-1.50 |
-0.76 |
0.81 |
1.16 |
1.18 |
1.73 |
0.20 |
0.47 |
2.03 |
2.63 |
3.33 |
2.38 |
0.04 |
0.11 |
1 |
-0.02 |
-0.01 |
0.00 |
0.00 |
0.00 |
-0.00 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.03 |
-0.02 |
-0.02 |
-0.00 |
0.00 |
0.02 |
0.01 |
0.02 |
0.01 |
0.03 |
0.03 |
0.02 |
0.02 |
0.01 |
0.00 |
| GM.5.0.C2H2_ZF.0197 |
NO ORTHOLOGS FOUND |
|
0.68 |
0.80 |
0.48 |
0.75 |
-1.41 |
1.99 |
1.62 |
2.23 |
1.55 |
1.93 |
1.30 |
1.58 |
2.04 |
-2.68 |
-1.92 |
-1.83 |
-1.88 |
-0.42 |
-0.92 |
-0.45 |
-0.80 |
-1.46 |
-1.45 |
-1.93 |
-3.27 |
<1 |
0.03 |
0.02 |
0.02 |
0.03 |
0.02 |
0.03 |
0.03 |
0.04 |
0.04 |
0.03 |
0.02 |
0.02 |
0.02 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.03 |
-0.02 |
-0.04 |
-0.04 |
-0.04 |
-0.04 |
-0.04 |
-0.04 |
| GM.5.0.bZIP.0066 |
FOSB,MAFF,MAFG,FOS,FOSL2, (...) |
|
-3.00 |
-2.57 |
-2.09 |
-2.62 |
-2.83 |
-3.32 |
-2.09 |
-0.71 |
0.53 |
-0.22 |
0.56 |
0.24 |
0.37 |
2.56 |
3.13 |
1.28 |
3.33 |
2.85 |
1.72 |
3.35 |
2.52 |
3.37 |
3.12 |
-0.11 |
-0.38 |
<1 |
-0.05 |
-0.06 |
-0.03 |
-0.02 |
-0.02 |
-0.02 |
-0.03 |
-0.00 |
-0.00 |
-0.00 |
-0.01 |
0.01 |
0.01 |
0.02 |
0.02 |
0.03 |
0.02 |
0.04 |
0.04 |
0.05 |
0.04 |
0.03 |
0.01 |
0.02 |
0.00 |
| GM.5.0.bHLH.0054 |
TCF3,TCF4,TCF12 |
|
-0.55 |
0.46 |
1.17 |
-0.14 |
-0.58 |
-0.32 |
0.19 |
-0.57 |
0.36 |
-2.42 |
-2.35 |
-1.54 |
-2.32 |
-1.55 |
-0.57 |
-0.26 |
1.66 |
-0.32 |
-0.16 |
0.70 |
0.74 |
1.95 |
3.54 |
1.14 |
1.02 |
<1 |
-0.02 |
-0.01 |
0.02 |
0.02 |
0.01 |
-0.00 |
-0.01 |
-0.02 |
-0.03 |
-0.06 |
-0.06 |
-0.04 |
-0.04 |
-0.03 |
-0.02 |
0.01 |
0.02 |
0.01 |
0.01 |
0.04 |
0.05 |
0.03 |
0.04 |
0.03 |
0.02 |
| GM.5.0.Paired_box.0013 |
PAX1,PAX8,PAX9,PAX5 |
|
-1.31 |
-2.10 |
-2.51 |
-2.68 |
-1.72 |
-3.11 |
-3.54 |
-1.99 |
-0.40 |
-1.52 |
-1.19 |
-0.31 |
-0.02 |
1.43 |
0.55 |
2.12 |
2.62 |
2.57 |
3.93 |
2.30 |
1.96 |
1.67 |
0.00 |
2.49 |
0.81 |
<1 |
-0.05 |
-0.04 |
-0.01 |
0.00 |
-0.00 |
-0.02 |
-0.03 |
-0.02 |
-0.02 |
-0.02 |
-0.04 |
-0.01 |
-0.01 |
0.01 |
0.01 |
0.03 |
0.02 |
0.04 |
0.03 |
0.05 |
0.05 |
0.03 |
0.01 |
0.03 |
0.01 |
| GM.5.0.Homeodomain.0092 |
ONECUT3,OTX1,CRX,LHX6,ONECUT2, (...) |
|
1.78 |
2.12 |
0.15 |
0.35 |
-1.28 |
0.88 |
-0.41 |
3.45 |
3.61 |
3.42 |
3.38 |
2.60 |
3.59 |
0.44 |
1.41 |
-0.13 |
-1.96 |
-1.49 |
-2.38 |
-3.54 |
-3.03 |
-2.69 |
-2.91 |
-1.80 |
-2.79 |
1 |
-0.02 |
-0.02 |
-0.01 |
-0.00 |
-0.01 |
0.00 |
-0.01 |
0.05 |
0.06 |
0.06 |
0.05 |
0.05 |
0.05 |
0.03 |
0.02 |
0.00 |
-0.01 |
-0.01 |
-0.01 |
-0.03 |
-0.03 |
-0.03 |
-0.04 |
-0.03 |
-0.04 |
| GM.5.0.C2H2_ZF.0229 |
NO ORTHOLOGS FOUND |
|
-0.46 |
-0.13 |
0.70 |
1.22 |
1.94 |
0.99 |
1.55 |
-1.19 |
-0.97 |
-1.32 |
-1.19 |
-2.86 |
-3.30 |
-2.16 |
-1.17 |
-1.14 |
1.95 |
-1.49 |
-1.60 |
0.42 |
1.58 |
0.63 |
2.37 |
-0.59 |
-0.18 |
1 |
-0.01 |
-0.00 |
0.02 |
0.04 |
0.03 |
0.02 |
0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
-0.02 |
-0.02 |
-0.00 |
0.01 |
-0.00 |
-0.00 |
0.01 |
0.01 |
0.01 |
0.01 |
0.00 |
-0.00 |
| GM.5.0.C2H2_ZF.0218 |
SP1,CHD1,E2F1,CHD2 |
|
3.43 |
2.67 |
2.41 |
0.61 |
-0.10 |
0.30 |
0.03 |
1.77 |
0.05 |
0.33 |
-0.58 |
2.47 |
0.69 |
-1.65 |
-1.02 |
-2.27 |
-0.36 |
-1.97 |
-2.11 |
-1.91 |
-1.77 |
-2.30 |
-1.67 |
-3.06 |
-1.45 |
<1 |
0.02 |
0.02 |
0.03 |
0.04 |
0.02 |
0.03 |
0.01 |
0.03 |
0.02 |
0.01 |
-0.00 |
0.02 |
0.02 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
| GM.5.0.bZIP.0102 |
PAX1,PAX9,FOSL1,PAX8,PAX5 |
|
1.66 |
2.71 |
2.54 |
3.68 |
3.01 |
1.16 |
1.26 |
0.12 |
0.80 |
-1.97 |
-2.49 |
-2.18 |
-1.77 |
-3.94 |
-3.48 |
-3.09 |
-1.27 |
-1.85 |
-1.52 |
-1.32 |
-1.16 |
-0.22 |
-0.08 |
-0.77 |
0.02 |
<1 |
0.04 |
0.04 |
0.04 |
0.06 |
0.05 |
0.03 |
0.02 |
0.01 |
0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.04 |
-0.04 |
-0.04 |
-0.02 |
-0.04 |
-0.04 |
-0.04 |
-0.03 |
-0.03 |
-0.01 |
-0.02 |
-0.01 |
| GM.5.0.C2H2_ZF.0224 |
MBTPS2,YY1 |
|
0.72 |
2.00 |
0.55 |
1.17 |
0.51 |
0.75 |
0.32 |
-1.62 |
-1.77 |
-0.37 |
-0.16 |
-1.21 |
-1.38 |
-3.33 |
-2.09 |
-0.64 |
-0.34 |
-1.59 |
-2.47 |
-0.57 |
-0.46 |
-0.29 |
1.36 |
1.36 |
0.92 |
<1 |
0.01 |
0.01 |
0.03 |
0.05 |
0.04 |
0.03 |
0.01 |
-0.00 |
-0.01 |
-0.02 |
-0.03 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.01 |
0.00 |
-0.01 |
0.00 |
-0.00 |
-0.01 |
| GM.5.0.C2H2_ZF.0260 |
YY1,THAP1,LOC100493491,ELF1,GABPA, (...) |
|
2.41 |
3.26 |
1.41 |
1.06 |
1.55 |
1.13 |
1.18 |
0.82 |
1.00 |
-0.15 |
-0.14 |
-1.36 |
-0.55 |
-2.59 |
-2.20 |
-3.19 |
-1.41 |
-1.18 |
-1.21 |
-1.17 |
-0.74 |
-0.52 |
0.45 |
-0.24 |
0.74 |
1 |
0.03 |
0.02 |
0.02 |
0.03 |
0.03 |
0.03 |
0.01 |
0.00 |
0.00 |
-0.02 |
-0.02 |
-0.02 |
-0.01 |
-0.03 |
-0.03 |
-0.02 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.01 |
-0.00 |
-0.01 |
-0.01 |
| GM.5.0.bHLH.0059 |
SREBF1,SREBF2,SCAP |
|
1.36 |
2.32 |
0.52 |
1.83 |
1.65 |
0.24 |
0.54 |
0.97 |
-0.54 |
-0.35 |
1.10 |
-0.60 |
-1.58 |
-2.28 |
-3.27 |
-0.84 |
-1.59 |
-0.86 |
-0.72 |
-1.25 |
-0.84 |
0.34 |
0.49 |
-0.04 |
0.06 |
1 |
-0.00 |
0.00 |
0.01 |
0.03 |
0.02 |
0.01 |
0.00 |
0.02 |
0.01 |
0.01 |
0.01 |
0.01 |
0.01 |
-0.00 |
-0.01 |
-0.00 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
| GM.5.0.Homeodomain.0146 |
DRGX,PRRX1 |
|
-3.93 |
-3.82 |
-2.82 |
-4.21 |
-3.05 |
-3.89 |
-3.67 |
2.39 |
2.45 |
1.85 |
1.48 |
3.23 |
2.26 |
3.77 |
2.73 |
3.05 |
2.11 |
1.80 |
2.94 |
2.61 |
1.47 |
1.14 |
-1.41 |
1.57 |
1.35 |
4 |
-0.06 |
-0.05 |
-0.03 |
-0.03 |
-0.02 |
-0.02 |
-0.02 |
0.02 |
0.02 |
0.02 |
0.01 |
0.03 |
0.02 |
0.03 |
0.03 |
0.03 |
0.02 |
0.03 |
0.02 |
0.03 |
0.02 |
0.01 |
0.00 |
0.00 |
-0.01 |
| GM.5.0.C2H2_ZF.0132 |
GZF1 |
|
2.94 |
2.77 |
4.03 |
3.78 |
3.66 |
3.47 |
2.67 |
2.44 |
0.32 |
-1.03 |
-0.48 |
-1.17 |
-1.67 |
-1.23 |
-1.34 |
-3.46 |
-2.84 |
-2.16 |
-3.01 |
-0.88 |
-2.27 |
-3.57 |
-1.98 |
-3.60 |
-4.34 |
1 |
0.06 |
0.04 |
0.06 |
0.07 |
0.06 |
0.05 |
0.04 |
0.03 |
0.02 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.03 |
-0.03 |
-0.04 |
-0.03 |
-0.05 |
-0.04 |
-0.05 |
-0.05 |
-0.05 |
-0.03 |
-0.05 |
-0.04 |
| GM.5.0.Homeodomain_POU.0025 |
POU3F2,POU3F3,POU3F4,POU3F1,POU1F1 |
|
2.55 |
3.39 |
1.12 |
1.72 |
2.61 |
3.53 |
3.04 |
1.22 |
1.08 |
-0.09 |
-1.43 |
0.25 |
-0.23 |
-1.16 |
-2.07 |
-2.52 |
-0.37 |
-2.10 |
-2.41 |
-2.60 |
-2.79 |
-3.01 |
-2.43 |
-2.73 |
-2.35 |
<1 |
0.07 |
0.06 |
0.04 |
0.06 |
0.05 |
0.05 |
0.04 |
0.02 |
0.02 |
-0.01 |
-0.03 |
-0.02 |
-0.01 |
-0.04 |
-0.04 |
-0.03 |
-0.02 |
-0.05 |
-0.06 |
-0.06 |
-0.04 |
-0.04 |
-0.03 |
-0.04 |
-0.03 |
| GM.5.0.Mixed.0050 |
PRDM1,IRF1 |
|
0.28 |
2.07 |
1.38 |
1.22 |
0.60 |
0.58 |
0.37 |
-1.37 |
-0.57 |
-2.53 |
-2.13 |
-1.06 |
-1.74 |
-3.10 |
-0.62 |
-1.05 |
-0.58 |
-0.41 |
0.78 |
-0.50 |
-1.22 |
-0.38 |
-1.01 |
-0.12 |
1.23 |
<1 |
0.01 |
0.01 |
0.04 |
0.05 |
0.05 |
0.03 |
0.02 |
0.01 |
-0.00 |
-0.02 |
-0.03 |
-0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.01 |
-0.01 |
-0.02 |
-0.01 |
-0.02 |
-0.01 |
-0.02 |
-0.01 |
-0.01 |
-0.01 |
| GM.5.0.Unknown.0207 |
ZBTB14,ZNF852,ZNF250,EGR1 |
|
1.19 |
-0.44 |
-0.15 |
1.01 |
-0.60 |
0.51 |
0.07 |
0.12 |
1.10 |
0.95 |
1.19 |
1.49 |
2.18 |
1.32 |
0.19 |
-0.15 |
1.21 |
0.90 |
1.36 |
-0.17 |
-1.34 |
-1.02 |
-2.57 |
-1.69 |
-3.18 |
<1 |
0.04 |
0.01 |
0.03 |
0.04 |
0.03 |
0.04 |
0.02 |
0.03 |
0.02 |
0.01 |
0.01 |
0.02 |
0.02 |
-0.01 |
-0.02 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.04 |
-0.04 |
-0.04 |
-0.04 |
-0.05 |
-0.05 |
| GM.5.0.Unknown.0040 |
ZBTB7C,NO ORTHOLOGS FOUND |
|
4.79 |
4.33 |
5.16 |
5.88 |
5.05 |
4.96 |
4.53 |
2.99 |
2.82 |
-2.86 |
-2.61 |
-4.27 |
-3.51 |
-4.94 |
-4.42 |
-5.40 |
-3.74 |
-4.55 |
-4.84 |
-4.85 |
-4.55 |
-4.02 |
-1.13 |
-3.96 |
-2.94 |
1 |
0.06 |
0.08 |
0.10 |
0.13 |
0.12 |
0.09 |
0.06 |
0.05 |
0.04 |
-0.02 |
-0.03 |
-0.03 |
-0.03 |
-0.07 |
-0.07 |
-0.06 |
-0.03 |
-0.09 |
-0.09 |
-0.11 |
-0.08 |
-0.06 |
-0.03 |
-0.07 |
-0.05 |
| GM.5.0.IRF.0019 |
IRF8,IRF10,IRF4 |
|
-1.44 |
-1.28 |
-1.51 |
-2.41 |
-1.39 |
-0.72 |
0.04 |
-2.16 |
-2.48 |
-2.75 |
-1.87 |
-2.06 |
-2.03 |
1.94 |
0.37 |
1.56 |
2.72 |
3.49 |
2.15 |
1.79 |
1.50 |
1.35 |
0.12 |
1.89 |
0.71 |
2 |
-0.02 |
-0.02 |
-0.01 |
-0.01 |
-0.01 |
-0.00 |
-0.01 |
-0.00 |
-0.01 |
-0.01 |
-0.01 |
0.00 |
-0.00 |
0.01 |
0.01 |
0.02 |
0.01 |
0.02 |
0.02 |
0.02 |
0.02 |
0.01 |
-0.01 |
0.02 |
-0.00 |
| GM.5.0.Myb_SANT.0001 |
MYBL1,MYBL2 |
|
-1.32 |
-2.00 |
-2.03 |
-1.39 |
-0.43 |
-1.59 |
-1.28 |
-1.04 |
-2.48 |
1.00 |
1.05 |
1.64 |
0.88 |
1.37 |
1.32 |
0.64 |
1.55 |
1.23 |
2.45 |
1.51 |
-1.03 |
3.05 |
-0.12 |
0.22 |
0.95 |
1 |
-0.06 |
-0.05 |
-0.03 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.01 |
-0.01 |
0.01 |
-0.00 |
0.02 |
0.01 |
0.03 |
0.02 |
0.03 |
0.01 |
0.04 |
0.03 |
0.04 |
0.03 |
0.03 |
0.01 |
0.01 |
-0.00 |
| GM.5.0.Ndt80_PhoG.0001 |
MYRFL,GCM1,MYRF,GCM2 |
|
3.48 |
2.62 |
2.16 |
2.55 |
3.15 |
1.79 |
2.59 |
0.96 |
1.20 |
0.15 |
0.13 |
-0.03 |
-1.52 |
-1.98 |
-2.89 |
-2.83 |
-1.86 |
-2.15 |
-2.42 |
-2.30 |
-0.76 |
-2.10 |
-0.78 |
-1.86 |
-1.09 |
<1 |
0.02 |
0.03 |
0.05 |
0.06 |
0.06 |
0.04 |
0.03 |
0.02 |
0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.04 |
-0.04 |
-0.03 |
-0.02 |
-0.04 |
-0.03 |
-0.03 |
-0.02 |
-0.03 |
-0.01 |
-0.03 |
-0.02 |
| GM.5.0.C2H2_ZF.0236 |
MAF,MAFB,MAFA,MAFF,MAFG, (...) |
|
-2.48 |
-2.85 |
0.38 |
0.32 |
0.64 |
0.63 |
-0.91 |
-0.83 |
0.05 |
-1.23 |
-0.08 |
0.22 |
-0.02 |
0.47 |
-0.38 |
1.49 |
-1.20 |
1.47 |
1.73 |
2.57 |
1.29 |
1.99 |
1.39 |
3.09 |
2.19 |
2 |
-0.03 |
-0.02 |
0.02 |
0.03 |
0.02 |
0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.01 |
-0.01 |
-0.00 |
-0.01 |
0.01 |
-0.00 |
0.01 |
0.01 |
0.02 |
0.02 |
0.01 |
0.00 |
0.01 |
0.01 |
| GM.5.0.bHLH.0072 |
OSR2,MYOG,MYF5,MYOD1,MYF6, (...) |
|
-1.64 |
-2.40 |
-2.33 |
-2.64 |
-2.23 |
-2.33 |
-1.19 |
-0.74 |
0.54 |
2.33 |
1.87 |
1.18 |
0.68 |
3.03 |
1.72 |
2.61 |
1.18 |
2.66 |
2.57 |
-0.36 |
-0.42 |
1.46 |
-0.70 |
-0.60 |
-0.80 |
2 |
-0.05 |
-0.04 |
-0.01 |
0.00 |
-0.00 |
-0.01 |
-0.02 |
-0.01 |
-0.01 |
0.00 |
-0.00 |
0.02 |
0.01 |
0.03 |
0.02 |
0.04 |
0.01 |
0.04 |
0.04 |
0.02 |
0.01 |
0.01 |
-0.01 |
0.01 |
-0.00 |
| GM.5.0.C2H2_ZF.0195 |
BCL11B,BCL11A |
|
-2.97 |
-2.33 |
-1.26 |
-2.31 |
-2.13 |
-1.40 |
-1.66 |
-1.49 |
-2.21 |
-0.55 |
-0.64 |
-1.74 |
-0.38 |
2.01 |
1.98 |
2.24 |
3.37 |
2.79 |
1.97 |
1.61 |
2.41 |
2.57 |
2.86 |
2.61 |
0.29 |
1 |
-0.04 |
-0.03 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.03 |
-0.04 |
-0.04 |
-0.03 |
-0.02 |
0.01 |
0.01 |
0.03 |
0.03 |
0.03 |
0.02 |
0.05 |
0.05 |
0.04 |
0.03 |
0.04 |
0.02 |
| GM.5.0.Nuclear_receptor.0145 |
HNF4B,HNF4G,HNF4A,DBP |
|
-2.89 |
-3.63 |
-3.77 |
-2.08 |
-2.06 |
-1.94 |
-3.63 |
-1.83 |
0.88 |
2.93 |
2.98 |
1.41 |
1.64 |
3.82 |
3.73 |
3.13 |
1.28 |
4.17 |
3.34 |
2.73 |
0.81 |
1.03 |
-0.73 |
1.11 |
0.16 |
2 |
-0.05 |
-0.05 |
-0.02 |
-0.01 |
-0.02 |
-0.01 |
-0.03 |
-0.01 |
-0.00 |
0.02 |
0.03 |
0.02 |
0.03 |
0.04 |
0.02 |
0.03 |
0.00 |
0.04 |
0.04 |
0.03 |
0.01 |
0.01 |
-0.01 |
0.00 |
-0.01 |
| GM.5.0.Paired_box.0016 |
PAX5,PAX8,PAX7,PAX1,PAX9, (...) |
|
-1.61 |
-0.57 |
-1.12 |
-0.54 |
-0.51 |
-1.24 |
-3.34 |
-0.69 |
-0.04 |
-2.04 |
-0.46 |
-1.62 |
-0.20 |
-1.00 |
-0.85 |
0.62 |
1.20 |
1.52 |
1.95 |
0.63 |
0.92 |
0.14 |
0.24 |
1.15 |
0.61 |
1 |
-0.01 |
-0.02 |
-0.00 |
0.01 |
0.01 |
0.00 |
-0.02 |
-0.00 |
-0.01 |
-0.02 |
-0.02 |
-0.01 |
-0.00 |
-0.00 |
-0.00 |
0.01 |
0.01 |
0.02 |
0.02 |
0.02 |
0.02 |
0.01 |
-0.00 |
0.01 |
-0.00 |
| GM.5.0.TEA.0004 |
TEAD1,TEAD4 |
|
-0.92 |
-0.94 |
-1.92 |
-1.08 |
-2.17 |
-1.39 |
-0.46 |
-1.54 |
-3.68 |
-1.19 |
-1.37 |
0.33 |
-0.85 |
1.04 |
2.11 |
1.72 |
2.47 |
0.74 |
2.07 |
0.45 |
2.25 |
1.45 |
1.98 |
-0.88 |
-1.35 |
<1 |
-0.03 |
-0.02 |
0.01 |
0.02 |
0.01 |
0.01 |
0.00 |
-0.00 |
-0.02 |
-0.03 |
-0.04 |
-0.01 |
-0.01 |
0.00 |
0.01 |
0.02 |
0.01 |
0.02 |
0.01 |
0.02 |
0.03 |
0.01 |
0.01 |
-0.01 |
-0.02 |
| GM.5.0.C2H2_ZF.0124 |
LOC116411990 |
|
2.58 |
1.71 |
1.13 |
1.78 |
0.96 |
1.37 |
0.65 |
1.43 |
1.29 |
1.25 |
2.06 |
0.83 |
0.96 |
-0.68 |
-0.84 |
-3.29 |
-2.28 |
-0.69 |
-0.38 |
-2.60 |
-3.87 |
-3.26 |
-3.90 |
-1.63 |
-1.19 |
1 |
0.04 |
0.02 |
0.02 |
0.04 |
0.02 |
0.02 |
0.01 |
0.03 |
0.03 |
0.02 |
0.01 |
0.01 |
0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.05 |
-0.04 |
-0.04 |
-0.03 |
-0.03 |
-0.03 |
| GM.5.0.C2H2_ZF.0179 |
HIC1 |
|
2.77 |
4.19 |
3.58 |
2.94 |
3.81 |
1.47 |
2.08 |
1.31 |
2.02 |
-1.48 |
-2.09 |
-2.29 |
-2.21 |
-4.52 |
-4.22 |
-2.92 |
-1.88 |
-3.62 |
-3.34 |
-2.90 |
-1.32 |
-1.73 |
2.53 |
-3.15 |
-0.01 |
1 |
0.05 |
0.05 |
0.05 |
0.06 |
0.06 |
0.03 |
0.03 |
0.02 |
0.02 |
-0.02 |
-0.02 |
-0.03 |
-0.02 |
-0.04 |
-0.04 |
-0.04 |
-0.02 |
-0.05 |
-0.05 |
-0.05 |
-0.03 |
-0.03 |
-0.01 |
-0.03 |
-0.01 |
| GM.5.0.Unknown.0183 |
NO ORTHOLOGS FOUND |
|
0.20 |
-0.47 |
0.29 |
1.13 |
1.54 |
1.66 |
1.38 |
0.08 |
0.30 |
-2.04 |
-2.02 |
-1.68 |
-2.46 |
-1.90 |
-1.29 |
-0.97 |
-0.63 |
-1.17 |
-1.77 |
-0.45 |
-0.29 |
1.53 |
-0.22 |
2.39 |
3.21 |
2 |
-0.02 |
-0.01 |
0.02 |
0.03 |
0.03 |
0.02 |
0.00 |
-0.01 |
-0.01 |
-0.04 |
-0.04 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.00 |
0.00 |
0.00 |
-0.00 |
0.01 |
0.01 |
0.01 |
0.01 |
0.02 |
0.01 |
| GM.5.0.Nuclear_receptor.0073 |
RXRB,RXRG,RXRA,NR1I2,VDR, (...) |
|
-2.50 |
-1.38 |
-0.97 |
-1.24 |
-0.87 |
-1.94 |
-0.46 |
-0.38 |
-1.50 |
1.78 |
1.51 |
1.24 |
1.53 |
-0.23 |
-1.24 |
0.39 |
1.31 |
-0.74 |
0.08 |
0.32 |
2.17 |
1.86 |
3.11 |
2.79 |
2.64 |
2 |
-0.06 |
-0.04 |
-0.00 |
0.01 |
0.00 |
-0.00 |
-0.02 |
-0.01 |
-0.02 |
-0.02 |
-0.03 |
-0.01 |
-0.01 |
-0.00 |
-0.01 |
0.01 |
0.01 |
0.01 |
0.01 |
0.04 |
0.04 |
0.04 |
0.02 |
0.04 |
0.03 |
| GM.5.0.C2H2_ZF.0225 |
SMAD1,SMAD9,NO ORTHOLOGS FOUND |
|
0.01 |
-0.46 |
-0.06 |
-0.68 |
-1.04 |
0.24 |
0.70 |
-0.84 |
-0.75 |
-0.98 |
-1.42 |
0.31 |
0.56 |
-0.98 |
-1.77 |
-0.54 |
-0.01 |
-0.52 |
0.01 |
0.57 |
0.37 |
1.87 |
3.01 |
0.99 |
1.29 |
1 |
-0.03 |
-0.02 |
-0.00 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.00 |
-0.00 |
0.01 |
-0.00 |
0.01 |
0.02 |
0.01 |
0.00 |
0.01 |
-0.00 |
0.01 |
0.00 |
0.01 |
0.01 |
0.02 |
0.01 |
0.01 |
-0.00 |
| GM.5.0.Nuclear_receptor.0045 |
ESR1,NR2F2,RXRA,RXRB,RORA, (...) |
|
-3.13 |
-3.29 |
-2.81 |
-3.33 |
-0.91 |
-3.26 |
-3.90 |
-4.62 |
-4.36 |
-2.00 |
-1.10 |
-0.74 |
0.06 |
2.24 |
-0.85 |
0.55 |
-1.42 |
3.21 |
3.96 |
3.38 |
3.87 |
3.33 |
1.87 |
4.29 |
3.90 |
2 |
-0.06 |
-0.05 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.01 |
-0.00 |
0.01 |
0.00 |
0.01 |
0.01 |
0.04 |
0.03 |
0.05 |
0.05 |
0.04 |
0.02 |
0.05 |
0.03 |
| GM.5.0.Forkhead.0055 |
FOXD1,FOXE3,FOXD3,FOXL2,FOXF1, (...) |
|
0.54 |
-0.01 |
-1.19 |
-2.02 |
-1.24 |
-0.04 |
-0.44 |
0.91 |
1.72 |
2.15 |
1.79 |
2.18 |
3.12 |
2.62 |
1.70 |
-0.97 |
-1.49 |
0.79 |
2.04 |
-0.02 |
-3.22 |
-2.70 |
-2.03 |
-1.28 |
-1.57 |
2 |
0.00 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.00 |
-0.00 |
0.02 |
0.02 |
0.05 |
0.05 |
0.05 |
0.04 |
0.03 |
0.02 |
0.00 |
-0.02 |
0.01 |
0.01 |
-0.01 |
-0.04 |
-0.03 |
-0.04 |
-0.02 |
-0.03 |
| GM.5.0.C2H2_ZF.0315 |
RUNX1,MECOM,RUNX2,RUNX3,PRDM16 |
|
-2.59 |
-1.28 |
-1.42 |
-0.83 |
-2.11 |
-0.18 |
-0.96 |
-0.03 |
-0.93 |
-2.51 |
-2.08 |
0.70 |
-0.09 |
1.42 |
1.99 |
3.62 |
3.38 |
1.78 |
1.88 |
1.97 |
1.39 |
2.97 |
1.94 |
1.03 |
1.41 |
<1 |
-0.07 |
-0.05 |
-0.01 |
0.01 |
0.00 |
0.00 |
-0.01 |
0.01 |
0.00 |
-0.01 |
-0.03 |
0.01 |
0.00 |
0.02 |
0.02 |
0.04 |
0.03 |
0.03 |
0.02 |
0.02 |
0.03 |
0.02 |
0.01 |
0.00 |
-0.02 |
| GM.5.0.Unknown.0005 |
NO ORTHOLOGS FOUND |
|
2.33 |
2.62 |
1.43 |
1.86 |
1.93 |
2.51 |
1.76 |
1.56 |
0.82 |
-0.42 |
-1.03 |
-2.42 |
0.29 |
-2.70 |
-3.06 |
-1.08 |
0.58 |
-1.57 |
-2.11 |
-2.16 |
-0.60 |
0.11 |
0.16 |
-0.91 |
0.01 |
<1 |
0.01 |
0.01 |
0.04 |
0.06 |
0.05 |
0.04 |
0.02 |
0.02 |
0.01 |
-0.02 |
-0.03 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.01 |
0.00 |
-0.03 |
-0.03 |
-0.03 |
-0.01 |
-0.01 |
-0.00 |
-0.02 |
-0.02 |
| GM.5.0.MADS_box.0004 |
SRF |
|
0.39 |
0.83 |
0.54 |
-1.20 |
-2.18 |
-1.07 |
-0.91 |
-1.41 |
-0.98 |
1.34 |
3.05 |
2.07 |
1.47 |
0.66 |
0.35 |
-0.12 |
-0.46 |
0.14 |
0.12 |
0.12 |
0.53 |
-1.10 |
-0.53 |
-0.64 |
-1.95 |
2 |
-0.03 |
-0.03 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.01 |
-0.01 |
0.01 |
0.01 |
0.02 |
0.02 |
0.01 |
0.01 |
0.02 |
0.00 |
0.02 |
0.02 |
0.02 |
0.02 |
0.01 |
-0.00 |
0.00 |
-0.01 |
| GM.5.0.Myb_SANT.0011 |
SP9,SP3,TERF2 |
|
3.11 |
2.69 |
3.62 |
2.44 |
3.76 |
2.46 |
2.44 |
0.43 |
2.04 |
0.38 |
-0.30 |
-0.26 |
-1.01 |
-0.01 |
-1.95 |
-3.64 |
-2.91 |
-3.82 |
-3.94 |
-3.88 |
-3.47 |
-1.67 |
1.29 |
-2.89 |
-1.18 |
3 |
0.02 |
0.03 |
0.04 |
0.05 |
0.05 |
0.04 |
0.03 |
0.02 |
0.02 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.03 |
-0.01 |
-0.05 |
-0.05 |
-0.05 |
-0.03 |
-0.02 |
-0.01 |
-0.03 |
-0.02 |
| GM.5.0.C2H2_ZF.0199 |
SMC3,CTCF |
|
-2.84 |
-1.27 |
-1.17 |
0.43 |
0.82 |
0.27 |
-0.65 |
0.35 |
-0.58 |
-2.42 |
-3.22 |
-2.25 |
-2.07 |
-0.97 |
0.14 |
1.71 |
2.21 |
1.69 |
0.43 |
2.23 |
3.59 |
1.64 |
2.88 |
1.86 |
-0.03 |
<1 |
-0.05 |
-0.04 |
-0.01 |
0.01 |
0.01 |
0.00 |
-0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.05 |
-0.02 |
-0.02 |
0.00 |
0.01 |
0.03 |
0.03 |
0.03 |
0.02 |
0.04 |
0.05 |
0.02 |
0.02 |
0.01 |
0.00 |
| GM.5.0.Unknown.0026 |
TLX2,TLX3,DBX2,DBX1,CDX4, (...) |
|
-2.42 |
-1.51 |
0.32 |
1.70 |
2.52 |
-1.08 |
-2.61 |
-2.07 |
-1.82 |
-2.83 |
-3.88 |
-3.29 |
-2.68 |
-2.98 |
-0.97 |
0.01 |
2.86 |
0.34 |
0.21 |
0.43 |
0.76 |
2.27 |
2.71 |
2.44 |
2.78 |
<1 |
-0.03 |
-0.01 |
0.05 |
0.07 |
0.07 |
0.04 |
0.01 |
0.02 |
0.01 |
-0.03 |
-0.05 |
-0.02 |
-0.02 |
-0.03 |
-0.02 |
0.00 |
0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.03 |
| GM.5.0.Nuclear_receptor.0067 |
AR,PGR,NR3C1,NR3C2,HSF2 |
|
-2.55 |
-1.27 |
-1.49 |
-2.25 |
-0.57 |
-1.70 |
-2.47 |
-1.16 |
-0.25 |
-0.96 |
0.68 |
1.44 |
0.58 |
-0.72 |
-0.58 |
0.83 |
2.12 |
3.01 |
3.60 |
1.33 |
-0.35 |
0.30 |
-0.79 |
2.53 |
2.30 |
16 |
-0.04 |
-0.03 |
0.00 |
0.02 |
0.02 |
0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
0.01 |
0.01 |
0.02 |
0.02 |
0.02 |
0.02 |
0.01 |
0.00 |
0.01 |
0.01 |
| GM.5.0.HSF.0001 |
GTF3C2,HSF1,HSF4,POLR3G,POLR3GL, (...) |
|
2.45 |
0.70 |
2.08 |
2.14 |
3.13 |
2.17 |
1.91 |
2.96 |
2.75 |
0.91 |
1.18 |
-0.76 |
-0.12 |
-1.78 |
-0.55 |
-2.37 |
-2.07 |
-2.70 |
-2.31 |
-3.15 |
-2.27 |
-2.44 |
-2.18 |
-2.59 |
-2.80 |
1 |
0.01 |
0.01 |
0.04 |
0.05 |
0.05 |
0.04 |
0.02 |
0.03 |
0.02 |
0.00 |
-0.01 |
-0.01 |
-0.00 |
-0.02 |
-0.02 |
-0.02 |
-0.01 |
-0.03 |
-0.03 |
-0.03 |
-0.02 |
-0.03 |
-0.02 |
-0.03 |
-0.03 |
| GM.5.0.Homeodomain.0155 |
LOC116410824,LOC100485279,ZNF84-LIKE,LOC116407953,MEOX2, (...) |
|
1.93 |
2.23 |
1.39 |
0.92 |
3.31 |
0.79 |
0.68 |
0.21 |
-0.87 |
-0.14 |
-1.42 |
-1.14 |
-0.96 |
-2.60 |
-1.74 |
-2.85 |
0.17 |
-2.36 |
-1.95 |
-0.68 |
1.51 |
-1.10 |
0.01 |
-0.30 |
0.51 |
1 |
0.03 |
0.02 |
0.03 |
0.04 |
0.04 |
0.03 |
0.02 |
0.04 |
0.03 |
0.03 |
0.01 |
0.02 |
0.02 |
-0.02 |
-0.02 |
-0.01 |
-0.02 |
-0.04 |
-0.04 |
-0.05 |
-0.03 |
-0.05 |
-0.03 |
-0.05 |
-0.04 |
| GM.5.0.bHLH.0051 |
AHR,ARNT,AHRR |
|
-1.38 |
-1.92 |
-3.73 |
-4.31 |
-4.05 |
-2.34 |
-2.16 |
-1.30 |
-0.83 |
-0.03 |
-0.93 |
1.98 |
1.41 |
3.27 |
1.41 |
2.79 |
1.88 |
3.31 |
2.87 |
2.79 |
1.35 |
2.88 |
1.93 |
2.86 |
3.09 |
1 |
-0.03 |
-0.04 |
-0.03 |
-0.03 |
-0.03 |
-0.02 |
-0.02 |
-0.00 |
-0.00 |
0.01 |
0.01 |
0.02 |
0.02 |
0.02 |
0.01 |
0.02 |
0.00 |
0.03 |
0.03 |
0.03 |
0.02 |
0.02 |
-0.00 |
0.02 |
0.00 |
| GM.5.0.Nuclear_receptor.0135 |
RXRG,RXRA,RXRB |
|
-1.50 |
-2.74 |
-1.54 |
-1.23 |
-1.83 |
-0.08 |
-0.05 |
0.05 |
-0.14 |
1.64 |
0.30 |
1.63 |
1.08 |
3.20 |
3.35 |
2.35 |
1.92 |
0.38 |
-1.74 |
1.63 |
1.54 |
1.79 |
-2.06 |
-0.31 |
-2.34 |
1 |
-0.04 |
-0.03 |
-0.01 |
-0.00 |
-0.01 |
-0.00 |
-0.01 |
0.01 |
0.01 |
0.02 |
0.01 |
0.03 |
0.02 |
0.03 |
0.03 |
0.02 |
0.00 |
0.01 |
-0.00 |
0.01 |
0.01 |
-0.00 |
-0.02 |
-0.00 |
-0.01 |
| GM.5.0.C2H2_ZF.0134 |
NO ORTHOLOGS FOUND |
|
-2.01 |
-2.39 |
-1.15 |
-0.34 |
0.65 |
-2.00 |
-0.10 |
-2.86 |
-3.16 |
-0.34 |
-0.90 |
-1.53 |
-1.45 |
0.67 |
1.03 |
1.27 |
1.88 |
1.05 |
1.56 |
1.86 |
2.39 |
1.78 |
1.31 |
1.90 |
2.08 |
1 |
-0.04 |
-0.03 |
-0.00 |
0.01 |
0.00 |
-0.01 |
-0.02 |
-0.02 |
-0.01 |
-0.00 |
-0.01 |
0.00 |
0.00 |
0.01 |
0.01 |
0.02 |
0.02 |
0.02 |
0.02 |
0.03 |
0.02 |
0.01 |
0.01 |
0.01 |
0.01 |
| GM.5.0.C2H2_ZF.0137 |
STAT3.2,STAT4,STAT2,STAT3.1,STAT1, (...) |
|
-3.14 |
-2.22 |
-1.93 |
-1.64 |
-0.61 |
-0.86 |
-0.37 |
-0.40 |
-0.43 |
-1.45 |
-0.84 |
-0.89 |
-1.72 |
0.89 |
0.56 |
2.63 |
2.17 |
2.38 |
2.13 |
1.86 |
1.85 |
1.75 |
1.97 |
2.17 |
0.99 |
1 |
-0.04 |
-0.03 |
-0.00 |
0.01 |
0.01 |
0.00 |
-0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.05 |
-0.02 |
-0.02 |
-0.00 |
-0.00 |
0.03 |
0.02 |
0.03 |
0.02 |
0.04 |
0.04 |
0.02 |
0.02 |
0.02 |
0.01 |
| GM.5.0.Mixed.0041 |
ZFHX3,LHX5,PAX7,LHX4,LHX3, (...) |
|
0.64 |
0.54 |
-0.36 |
0.54 |
-0.32 |
-0.71 |
-2.06 |
3.75 |
3.64 |
2.88 |
1.56 |
2.67 |
1.83 |
0.52 |
0.38 |
-1.60 |
-2.47 |
-1.77 |
-1.93 |
-2.81 |
-1.73 |
-1.02 |
-0.39 |
-2.33 |
-2.23 |
1 |
-0.02 |
-0.02 |
-0.00 |
0.02 |
0.01 |
0.01 |
-0.01 |
0.05 |
0.05 |
0.04 |
0.03 |
0.04 |
0.04 |
0.02 |
0.01 |
0.00 |
-0.01 |
-0.00 |
-0.01 |
-0.02 |
-0.03 |
-0.02 |
-0.03 |
-0.03 |
-0.05 |
| GM.5.0.C2H2_ZF.0287 |
ZIC4,LOC100493456,ZIC5,ZIC2,GLI1, (...) |
|
1.29 |
1.90 |
1.40 |
2.18 |
2.56 |
1.90 |
1.95 |
1.48 |
2.03 |
2.47 |
3.31 |
1.36 |
1.47 |
1.11 |
1.25 |
-0.87 |
-3.18 |
-1.16 |
-1.69 |
-3.38 |
-2.82 |
-2.33 |
-2.65 |
-2.27 |
-2.47 |
2 |
0.02 |
0.01 |
0.02 |
0.04 |
0.03 |
0.04 |
0.04 |
0.05 |
0.05 |
0.04 |
0.03 |
0.03 |
0.03 |
0.02 |
0.01 |
-0.01 |
-0.02 |
-0.04 |
-0.04 |
-0.06 |
-0.06 |
-0.05 |
-0.05 |
-0.06 |
-0.06 |
| GM.5.0.C2H2_ZF.0233 |
NO ORTHOLOGS FOUND |
|
0.18 |
-1.60 |
-1.51 |
-2.23 |
-1.41 |
-3.16 |
-0.33 |
-1.82 |
-0.12 |
0.52 |
-0.57 |
-0.28 |
-0.71 |
-0.05 |
0.78 |
-0.12 |
1.41 |
0.66 |
0.60 |
1.13 |
1.20 |
0.09 |
-0.01 |
1.19 |
2.04 |
1 |
-0.04 |
-0.03 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.00 |
0.00 |
0.00 |
-0.01 |
0.01 |
0.01 |
0.02 |
0.02 |
0.02 |
0.01 |
0.02 |
0.02 |
0.02 |
0.02 |
0.01 |
-0.00 |
0.01 |
0.00 |
| GM.5.0.Homeodomain_Paired_box.0002 |
PAX1,PAX9,PAX8,PAX5,PAX6 |
|
-0.24 |
-0.94 |
-0.39 |
-0.19 |
-0.82 |
0.19 |
1.87 |
-1.86 |
-1.55 |
-2.19 |
-3.24 |
-0.89 |
-1.69 |
-0.22 |
0.44 |
0.48 |
1.51 |
1.41 |
0.54 |
0.86 |
0.54 |
0.65 |
0.99 |
0.70 |
0.62 |
<1 |
0.02 |
0.01 |
0.02 |
0.03 |
0.02 |
0.02 |
0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.04 |
-0.01 |
-0.01 |
-0.02 |
-0.01 |
-0.00 |
0.00 |
-0.00 |
-0.01 |
-0.00 |
0.00 |
-0.00 |
-0.00 |
0.00 |
-0.00 |
| GM.5.0.IRF.0010 |
IRF6,IRF5,IRF6.2,IRF2,IRF10, (...) |
|
4.52 |
4.33 |
1.56 |
3.02 |
1.32 |
3.51 |
4.69 |
2.08 |
1.08 |
-0.65 |
0.33 |
-3.19 |
-4.28 |
-3.70 |
-3.84 |
-3.00 |
-0.18 |
-3.35 |
-4.05 |
-2.62 |
-3.23 |
-2.15 |
-2.38 |
-3.00 |
-1.43 |
<1 |
0.04 |
0.04 |
0.02 |
0.04 |
0.03 |
0.04 |
0.03 |
0.02 |
0.00 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
-0.01 |
-0.00 |
-0.03 |
-0.03 |
-0.02 |
-0.01 |
-0.02 |
-0.01 |
-0.02 |
-0.02 |
| GM.5.0.bZIP.0012 |
ATF3,SREBF2,ATF6,ATF6B,SREBF1, (...) |
|
0.36 |
-2.09 |
-2.22 |
-0.98 |
-3.09 |
-2.14 |
-2.39 |
-0.86 |
-0.88 |
1.84 |
0.31 |
2.43 |
2.16 |
-1.14 |
-1.02 |
0.99 |
1.51 |
2.48 |
2.67 |
2.42 |
1.13 |
1.77 |
0.45 |
1.87 |
2.14 |
<1 |
-0.02 |
-0.04 |
-0.02 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
0.00 |
-0.00 |
-0.01 |
-0.01 |
0.01 |
0.02 |
0.01 |
0.00 |
0.02 |
0.01 |
0.03 |
0.02 |
0.03 |
0.02 |
0.01 |
0.00 |
0.02 |
0.00 |
| GM.5.0.Homeodomain.0201 |
TLX3,TLX1,TLX2 |
|
2.64 |
1.46 |
3.02 |
2.26 |
1.21 |
0.80 |
1.87 |
1.58 |
2.56 |
-0.11 |
0.69 |
-0.81 |
-2.14 |
-1.79 |
-2.03 |
-2.42 |
-2.00 |
-1.86 |
-2.10 |
-1.73 |
-1.58 |
-2.27 |
-2.29 |
-2.48 |
-1.17 |
1 |
0.02 |
0.02 |
0.03 |
0.04 |
0.03 |
0.03 |
0.02 |
0.03 |
0.03 |
0.01 |
0.01 |
0.00 |
-0.00 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
-0.02 |
| GM.5.0.Homeodomain.0028 |
CUX2,CUX1,ONECUT1,ONECUT3,ONECUT2, (...) |
|
2.76 |
3.19 |
0.88 |
1.74 |
1.93 |
1.11 |
0.17 |
1.02 |
1.74 |
-0.31 |
1.16 |
-0.87 |
0.49 |
-1.96 |
-1.85 |
-1.88 |
-2.08 |
-1.54 |
-2.35 |
-1.72 |
-1.60 |
-1.80 |
0.49 |
-2.04 |
-1.55 |
1 |
0.03 |
0.02 |
0.03 |
0.04 |
0.03 |
0.02 |
0.01 |
0.03 |
0.03 |
0.01 |
0.00 |
0.01 |
0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.04 |
-0.03 |
-0.04 |
-0.03 |
-0.04 |
-0.04 |
| GM.5.0.C2H2_ZF.0238 |
NO ORTHOLOGS FOUND |
|
1.86 |
1.48 |
-0.48 |
0.63 |
0.70 |
2.09 |
2.95 |
1.11 |
0.82 |
-1.24 |
-1.25 |
-1.57 |
-3.13 |
-2.65 |
-3.29 |
-2.40 |
0.22 |
-2.74 |
-2.33 |
-0.54 |
0.04 |
-1.33 |
-0.51 |
0.98 |
1.16 |
1 |
0.02 |
0.01 |
0.01 |
0.02 |
0.01 |
0.02 |
0.02 |
0.02 |
0.01 |
0.01 |
0.00 |
-0.00 |
-0.01 |
-0.01 |
-0.02 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.01 |
-0.01 |
| GM.5.0.C2H2_ZF.0194 |
ZBTB7B,ZIC4,INSM1,LOC100493456,ZBTB7C, (...) |
|
1.27 |
1.57 |
0.52 |
1.18 |
1.24 |
0.50 |
1.35 |
0.33 |
-0.18 |
-0.85 |
-0.30 |
-0.20 |
-0.26 |
-0.98 |
-0.92 |
-1.47 |
-0.70 |
-1.62 |
-0.89 |
-1.12 |
-0.29 |
-0.82 |
-3.05 |
-0.01 |
0.84 |
<1 |
0.00 |
0.01 |
0.03 |
0.04 |
0.03 |
0.02 |
0.02 |
0.02 |
0.02 |
-0.01 |
-0.01 |
-0.00 |
-0.00 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.03 |
-0.03 |
-0.03 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
| GM.5.0.Nuclear_receptor.0047 |
ESRRA,ESRRG,NR5A1,NR5A2,RARB, (...) |
|
-1.34 |
-0.57 |
-2.08 |
-1.09 |
-2.41 |
-2.62 |
-1.46 |
-3.19 |
-2.97 |
-0.46 |
0.13 |
-1.31 |
-1.04 |
-1.60 |
-0.80 |
0.00 |
-0.03 |
0.81 |
1.61 |
1.41 |
2.02 |
2.05 |
1.25 |
3.93 |
4.11 |
1 |
-0.05 |
-0.04 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.01 |
-0.01 |
0.00 |
0.00 |
0.01 |
0.00 |
0.03 |
0.02 |
0.05 |
0.04 |
0.04 |
0.02 |
0.05 |
0.03 |
| GM.5.0.C2H2_ZF.0005 |
ZEB1,ZEB2,TCF12,SNAI2,TCF3, (...) |
|
1.91 |
2.45 |
2.54 |
4.71 |
4.43 |
3.21 |
2.71 |
0.19 |
-1.70 |
-2.56 |
-3.65 |
-4.72 |
-4.62 |
-4.16 |
-3.44 |
-0.76 |
0.55 |
-2.02 |
-2.11 |
-0.24 |
1.51 |
-1.72 |
1.31 |
-0.10 |
-0.31 |
3 |
-0.01 |
-0.00 |
0.02 |
0.04 |
0.04 |
0.03 |
0.02 |
-0.00 |
-0.02 |
-0.04 |
-0.05 |
-0.03 |
-0.03 |
-0.03 |
-0.02 |
0.01 |
0.01 |
-0.01 |
-0.01 |
0.01 |
0.02 |
0.00 |
0.01 |
-0.00 |
-0.01 |
| GM.5.0.GATA.0023 |
GATA1,GATA2,GATA3 |
|
-2.26 |
-1.09 |
-2.65 |
-3.05 |
-2.79 |
-2.22 |
-1.72 |
0.69 |
-0.60 |
2.68 |
1.38 |
2.55 |
2.65 |
3.94 |
3.52 |
2.40 |
0.50 |
1.88 |
2.28 |
1.56 |
-0.30 |
0.24 |
-0.10 |
-0.03 |
-1.84 |
2 |
-0.06 |
-0.05 |
-0.03 |
-0.03 |
-0.03 |
-0.02 |
-0.02 |
0.01 |
0.01 |
0.04 |
0.03 |
0.05 |
0.05 |
0.05 |
0.04 |
0.03 |
0.01 |
0.03 |
0.02 |
0.02 |
0.00 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
| GM.5.0.Homeodomain.0125 |
NO ORTHOLOGS FOUND |
|
0.80 |
1.19 |
1.86 |
1.24 |
1.20 |
1.20 |
1.26 |
2.74 |
2.46 |
1.12 |
0.71 |
1.21 |
1.44 |
0.04 |
-0.85 |
-0.83 |
-0.75 |
-2.06 |
-2.12 |
-2.03 |
-1.96 |
-3.49 |
-2.83 |
-2.29 |
-1.83 |
<1 |
0.03 |
0.02 |
0.03 |
0.05 |
0.04 |
0.03 |
0.01 |
0.04 |
0.03 |
0.02 |
0.01 |
0.02 |
0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.03 |
-0.04 |
-0.04 |
-0.04 |
-0.05 |
-0.04 |
-0.04 |
-0.03 |
| GM.5.0.Unknown.0014 |
NO ORTHOLOGS FOUND |
|
0.42 |
1.08 |
3.06 |
1.66 |
2.63 |
0.96 |
-0.20 |
-1.91 |
-1.52 |
-1.23 |
-3.04 |
-2.41 |
-3.03 |
-1.93 |
-1.66 |
-1.32 |
-1.05 |
0.02 |
0.17 |
-0.53 |
-0.46 |
-0.85 |
-1.11 |
1.42 |
0.64 |
<1 |
-0.01 |
-0.00 |
0.03 |
0.03 |
0.03 |
0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.04 |
-0.04 |
-0.04 |
-0.03 |
-0.02 |
-0.02 |
0.00 |
0.01 |
0.01 |
0.01 |
0.02 |
0.02 |
0.01 |
0.01 |
0.02 |
0.01 |
| GM.5.0.Mixed.0060 |
FOXA2,STAT3.2,STAT4,FOXA1,STAT2, (...) |
|
-1.98 |
-1.12 |
-0.97 |
-0.71 |
0.25 |
-1.14 |
-1.20 |
-1.87 |
-2.69 |
-1.91 |
-2.38 |
-1.18 |
-0.45 |
0.10 |
-0.07 |
2.80 |
2.53 |
1.52 |
1.55 |
1.60 |
2.55 |
1.17 |
0.76 |
3.37 |
3.19 |
2 |
-0.03 |
-0.02 |
0.01 |
0.03 |
0.03 |
0.01 |
-0.00 |
-0.01 |
-0.02 |
-0.05 |
-0.07 |
-0.03 |
-0.03 |
-0.02 |
-0.01 |
0.02 |
0.02 |
0.01 |
0.00 |
0.03 |
0.04 |
0.02 |
0.01 |
0.02 |
0.01 |
| GM.5.0.GCM.0001 |
GCM2,GCM1 |
|
2.50 |
2.32 |
3.92 |
3.19 |
3.38 |
2.76 |
3.78 |
1.83 |
0.76 |
-0.94 |
-1.56 |
-1.63 |
-1.68 |
-2.36 |
-1.68 |
-2.44 |
-1.35 |
-3.07 |
-3.70 |
-3.20 |
-2.47 |
-0.45 |
-0.74 |
-3.61 |
-0.72 |
1 |
0.03 |
0.03 |
0.06 |
0.07 |
0.06 |
0.05 |
0.04 |
0.03 |
0.01 |
-0.02 |
-0.03 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.02 |
-0.01 |
-0.05 |
-0.05 |
-0.05 |
-0.03 |
-0.03 |
-0.01 |
-0.04 |
-0.03 |
| GM.5.0.Nuclear_receptor.0103 |
GATA1,POU2F2,GATA3,NR2C2,NR2C1, (...) |
|
0.15 |
-1.20 |
-0.71 |
-1.12 |
-1.62 |
0.14 |
0.71 |
-0.39 |
0.12 |
1.07 |
0.18 |
0.67 |
0.23 |
0.86 |
0.31 |
0.47 |
-0.54 |
1.06 |
0.59 |
-0.24 |
-1.07 |
0.28 |
-0.67 |
-1.96 |
-3.08 |
<1 |
0.05 |
0.03 |
0.02 |
0.02 |
0.01 |
0.02 |
0.02 |
0.02 |
0.01 |
0.01 |
0.02 |
0.01 |
0.01 |
-0.01 |
-0.02 |
-0.01 |
-0.03 |
-0.01 |
-0.02 |
-0.04 |
-0.04 |
-0.03 |
-0.03 |
-0.04 |
-0.04 |
| GM.5.0.C2H2_ZF.0281 |
NO ORTHOLOGS FOUND |
|
1.65 |
-0.05 |
3.59 |
1.95 |
2.72 |
1.15 |
1.90 |
2.77 |
2.51 |
-0.36 |
-0.34 |
-0.71 |
-1.09 |
-1.05 |
-1.21 |
-2.22 |
-1.80 |
-3.17 |
-2.38 |
-2.62 |
-1.24 |
-1.81 |
0.27 |
-2.40 |
-1.50 |
1 |
-0.00 |
0.00 |
0.03 |
0.04 |
0.03 |
0.02 |
0.02 |
0.02 |
0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.00 |
-0.03 |
-0.03 |
-0.01 |
-0.00 |
-0.01 |
0.00 |
-0.02 |
-0.02 |
| GM.5.0.Grainyhead.0011 |
UBP1,TFCP2,TFCP2L1 |
|
-0.59 |
-1.40 |
-2.01 |
-1.25 |
0.08 |
-2.15 |
-1.98 |
0.34 |
-0.23 |
-1.36 |
-0.17 |
-1.29 |
-1.08 |
0.23 |
1.04 |
1.30 |
3.39 |
0.10 |
-0.50 |
1.60 |
1.99 |
0.98 |
1.97 |
3.49 |
2.67 |
1 |
-0.06 |
-0.05 |
-0.03 |
-0.01 |
0.00 |
-0.01 |
-0.02 |
-0.02 |
-0.04 |
-0.06 |
-0.07 |
-0.05 |
-0.04 |
-0.01 |
0.00 |
0.04 |
0.05 |
0.03 |
0.02 |
0.07 |
0.08 |
0.06 |
0.05 |
0.06 |
0.03 |
| GM.5.0.Homeodomain.0052 |
PKNOX1,PBX2,PBX1,PBX3 |
|
-2.41 |
-3.32 |
-2.81 |
-3.03 |
-2.19 |
-3.18 |
-2.51 |
-1.81 |
-1.56 |
0.32 |
0.60 |
1.20 |
0.71 |
1.58 |
1.49 |
3.31 |
0.16 |
3.33 |
3.36 |
3.07 |
1.93 |
2.86 |
0.68 |
2.84 |
1.90 |
2 |
-0.05 |
-0.05 |
-0.02 |
-0.02 |
-0.02 |
-0.03 |
-0.04 |
-0.01 |
-0.02 |
-0.00 |
-0.01 |
0.01 |
0.01 |
0.03 |
0.02 |
0.04 |
0.01 |
0.04 |
0.04 |
0.05 |
0.03 |
0.02 |
0.01 |
0.03 |
0.01 |
| GM.5.0.C2H2_ZF.0175 |
STAT3.2,STAT4,STAT2,STAT3.1,STAT1, (...) |
|
-1.29 |
-0.80 |
-1.59 |
-2.06 |
-1.48 |
-0.89 |
-1.99 |
-0.76 |
-1.00 |
-0.36 |
-3.43 |
-1.20 |
-1.18 |
1.15 |
1.30 |
1.85 |
1.36 |
1.38 |
0.91 |
4.13 |
3.65 |
3.11 |
3.07 |
2.62 |
2.30 |
1 |
-0.05 |
-0.03 |
-0.00 |
0.01 |
0.00 |
0.00 |
-0.01 |
-0.01 |
-0.02 |
-0.04 |
-0.06 |
-0.03 |
-0.02 |
-0.01 |
-0.00 |
0.02 |
0.02 |
0.02 |
0.01 |
0.05 |
0.06 |
0.04 |
0.03 |
0.03 |
0.01 |
| GM.5.0.GATA.0006 |
GATA1,GATA2,GATA3 |
|
-3.31 |
-2.17 |
-3.50 |
-2.69 |
-2.50 |
-3.69 |
-2.53 |
0.57 |
0.97 |
1.29 |
1.41 |
2.22 |
2.32 |
4.16 |
3.77 |
3.60 |
3.03 |
1.90 |
1.96 |
1.64 |
-0.12 |
0.96 |
1.10 |
-0.22 |
-1.73 |
2 |
-0.05 |
-0.04 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
0.01 |
0.01 |
0.03 |
0.02 |
0.04 |
0.04 |
0.05 |
0.04 |
0.03 |
0.01 |
0.02 |
0.01 |
0.02 |
0.01 |
0.00 |
-0.01 |
-0.01 |
-0.02 |
| GM.5.0.GATA.0030 |
GATA1,GATA2,GATA3 |
|
0.66 |
1.00 |
0.70 |
0.23 |
0.26 |
-1.50 |
-0.71 |
2.44 |
3.12 |
1.45 |
0.54 |
-0.13 |
0.04 |
1.20 |
0.97 |
0.64 |
0.05 |
-0.98 |
-1.85 |
-0.66 |
-0.48 |
-0.85 |
0.79 |
0.75 |
0.98 |
2 |
-0.00 |
-0.00 |
-0.00 |
0.00 |
0.00 |
-0.01 |
-0.01 |
0.01 |
0.02 |
0.01 |
0.01 |
0.01 |
0.00 |
0.01 |
0.00 |
0.00 |
-0.00 |
-0.01 |
-0.01 |
-0.00 |
-0.00 |
-0.01 |
-0.01 |
-0.00 |
-0.00 |
| GM.5.0.C2H2_ZF.0310 |
NO ORTHOLOGS FOUND |
|
-1.78 |
-1.01 |
1.49 |
-0.52 |
0.33 |
0.26 |
-2.28 |
-2.18 |
-2.83 |
-1.61 |
-1.99 |
-0.92 |
-1.08 |
-0.69 |
-1.47 |
1.68 |
1.71 |
2.58 |
1.97 |
2.66 |
3.01 |
3.62 |
2.06 |
1.50 |
2.52 |
1 |
-0.03 |
-0.02 |
-0.00 |
0.00 |
0.00 |
-0.00 |
-0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.04 |
-0.01 |
-0.02 |
-0.00 |
-0.00 |
0.02 |
0.02 |
0.02 |
0.02 |
0.03 |
0.04 |
0.02 |
0.01 |
0.02 |
0.01 |
| GM.5.0.Homeodomain.0023 |
IRX4,IRX1,FOXO6,IRX5,FOXO4, (...) |
|
3.78 |
3.77 |
3.23 |
1.17 |
1.41 |
1.27 |
1.93 |
0.04 |
-1.55 |
-2.93 |
-3.15 |
-2.70 |
-2.06 |
-3.81 |
-2.13 |
-0.75 |
1.36 |
-3.57 |
-3.83 |
-1.82 |
-0.66 |
-1.45 |
1.34 |
-1.64 |
-0.63 |
2 |
0.05 |
0.04 |
0.03 |
0.05 |
0.04 |
0.04 |
0.03 |
0.01 |
-0.01 |
-0.04 |
-0.05 |
-0.04 |
-0.03 |
-0.04 |
-0.03 |
-0.01 |
0.01 |
-0.04 |
-0.04 |
-0.02 |
0.00 |
-0.01 |
0.00 |
-0.02 |
-0.02 |
| GM.5.0.bZIP.0025 |
XBP1,ATF1,ATF2,E4F1,CREB5, (...) |
|
-2.26 |
-3.08 |
-1.89 |
-0.13 |
-0.23 |
-2.51 |
-3.18 |
-1.78 |
-0.75 |
-2.23 |
-0.20 |
-0.04 |
1.63 |
-1.18 |
2.25 |
2.41 |
2.17 |
2.26 |
3.07 |
2.01 |
1.99 |
1.49 |
2.90 |
2.37 |
3.71 |
2 |
-0.03 |
-0.04 |
-0.01 |
0.00 |
-0.00 |
-0.00 |
-0.02 |
0.00 |
0.00 |
-0.01 |
-0.02 |
0.01 |
0.01 |
0.00 |
0.00 |
0.02 |
0.01 |
0.02 |
0.02 |
0.03 |
0.02 |
0.01 |
0.00 |
0.01 |
-0.00 |
| GM.5.0.Homeodomain.0139 |
PBX2,PBX3,PBX1,HOXA2,HOXD1, (...) |
|
-0.48 |
-2.21 |
-2.14 |
-3.39 |
-2.16 |
-3.34 |
-3.43 |
-2.10 |
-1.05 |
1.55 |
0.29 |
0.43 |
1.91 |
2.86 |
2.74 |
3.18 |
2.74 |
3.01 |
3.16 |
2.66 |
2.41 |
1.01 |
0.05 |
0.57 |
-0.02 |
2 |
-0.04 |
-0.04 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.00 |
0.00 |
0.01 |
0.00 |
0.02 |
0.03 |
0.03 |
0.02 |
0.03 |
0.01 |
0.03 |
0.03 |
0.02 |
0.01 |
0.00 |
-0.01 |
-0.00 |
-0.01 |
| GM.5.0.Forkhead.0050 |
FOXE3,FOXL2,FOXI1,FOXB2,FOXF2, (...) |
|
-1.69 |
-2.25 |
-2.57 |
-2.70 |
-2.79 |
-2.57 |
-2.98 |
-1.19 |
-0.76 |
1.10 |
1.21 |
2.83 |
3.14 |
3.24 |
2.36 |
3.42 |
-0.04 |
3.87 |
3.65 |
2.01 |
1.24 |
-0.08 |
-1.42 |
0.86 |
-1.10 |
2 |
-0.03 |
-0.04 |
-0.03 |
-0.03 |
-0.03 |
-0.02 |
-0.02 |
0.00 |
0.01 |
0.03 |
0.03 |
0.05 |
0.05 |
0.04 |
0.03 |
0.03 |
-0.00 |
0.04 |
0.03 |
0.02 |
0.00 |
-0.00 |
-0.02 |
-0.01 |
-0.03 |
| GM.5.0.Homeodomain.0186 |
HNF1A,HNF1B |
|
-2.73 |
-3.86 |
-2.00 |
-0.05 |
-0.84 |
-1.16 |
-3.05 |
-2.57 |
-1.03 |
1.16 |
2.74 |
2.01 |
1.96 |
1.58 |
-0.11 |
1.49 |
0.02 |
2.18 |
3.88 |
2.12 |
0.15 |
1.59 |
-1.69 |
2.12 |
2.40 |
1 |
-0.04 |
-0.04 |
-0.01 |
0.00 |
0.01 |
-0.01 |
-0.01 |
0.02 |
0.03 |
0.03 |
0.03 |
0.04 |
0.03 |
0.02 |
0.01 |
0.02 |
-0.01 |
0.03 |
0.03 |
0.00 |
-0.01 |
-0.01 |
-0.03 |
-0.01 |
-0.03 |
| GM.5.0.bZIP.0073 |
CREBZF |
|
0.92 |
0.50 |
0.75 |
-0.48 |
0.83 |
0.86 |
2.08 |
0.13 |
1.62 |
1.84 |
1.68 |
-0.76 |
-1.08 |
0.82 |
0.32 |
-1.87 |
-3.76 |
-1.13 |
-1.06 |
-1.67 |
-1.46 |
-1.04 |
0.53 |
-0.31 |
-0.90 |
1 |
0.03 |
0.01 |
0.02 |
0.04 |
0.03 |
0.03 |
0.02 |
0.02 |
0.01 |
0.01 |
0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.03 |
-0.01 |
-0.02 |
-0.02 |
| GM.5.0.GATA.0028 |
GATA1,GATA2,GFI1,GATA3 |
|
0.12 |
-1.84 |
-2.52 |
-3.56 |
-3.31 |
-1.48 |
0.19 |
1.47 |
1.03 |
2.85 |
2.46 |
3.59 |
2.58 |
2.01 |
2.19 |
2.11 |
0.81 |
0.74 |
-0.96 |
-0.80 |
-1.24 |
1.24 |
0.51 |
-0.77 |
-0.87 |
1 |
-0.01 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.02 |
-0.01 |
0.01 |
0.02 |
0.04 |
0.04 |
0.04 |
0.04 |
0.03 |
0.02 |
0.02 |
0.00 |
0.00 |
0.00 |
-0.01 |
-0.01 |
-0.00 |
-0.01 |
-0.02 |
-0.02 |
| GM.5.0.ARID_BRIGHT.0004 |
ARID5B |
|
3.67 |
4.35 |
2.82 |
2.98 |
0.99 |
0.58 |
2.59 |
1.10 |
2.37 |
-0.60 |
0.29 |
-0.69 |
-0.60 |
-1.70 |
-1.60 |
-3.74 |
-3.58 |
-2.95 |
-2.79 |
-3.65 |
-2.97 |
-2.69 |
-1.38 |
-2.20 |
-0.94 |
1 |
0.08 |
0.07 |
0.05 |
0.06 |
0.04 |
0.04 |
0.03 |
0.03 |
0.02 |
-0.00 |
0.00 |
-0.01 |
-0.01 |
-0.03 |
-0.04 |
-0.05 |
-0.04 |
-0.06 |
-0.05 |
-0.07 |
-0.06 |
-0.05 |
-0.03 |
-0.04 |
-0.03 |
| GM.5.0.Nuclear_receptor.0076 |
NR4A1,NR4A3,NR2F2,NR4A2,RXRA, (...) |
|
-2.21 |
-0.88 |
1.87 |
-0.02 |
1.31 |
-2.34 |
-1.24 |
-4.13 |
-2.84 |
-2.83 |
-2.09 |
-0.96 |
-1.40 |
1.69 |
1.02 |
-0.27 |
-0.26 |
2.42 |
1.60 |
3.72 |
4.08 |
2.25 |
2.22 |
2.44 |
2.37 |
2 |
-0.05 |
-0.03 |
0.00 |
0.01 |
0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.01 |
-0.01 |
0.00 |
0.00 |
0.01 |
-0.00 |
0.02 |
0.01 |
0.04 |
0.04 |
0.03 |
0.01 |
0.04 |
0.02 |
| GM.5.0.C2H2_ZF.0219 |
NO ORTHOLOGS FOUND |
|
2.08 |
3.27 |
1.62 |
2.50 |
2.17 |
1.94 |
2.77 |
1.98 |
-0.32 |
-1.27 |
-2.49 |
-0.96 |
-0.40 |
-2.71 |
-2.86 |
-2.86 |
-1.35 |
-1.77 |
-2.16 |
-1.97 |
-2.39 |
-1.25 |
0.18 |
-1.22 |
-0.68 |
1 |
0.02 |
0.02 |
0.02 |
0.03 |
0.02 |
0.03 |
0.02 |
0.02 |
0.01 |
0.01 |
-0.00 |
0.01 |
0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.03 |
-0.02 |
-0.03 |
-0.02 |
| GM.5.0.ARID_BRIGHT.0002 |
FOXE3,FOXL2,FOXI1,FOXB2,FOXF2, (...) |
|
0.99 |
1.64 |
3.66 |
4.04 |
3.76 |
2.81 |
1.73 |
0.42 |
1.25 |
-3.11 |
-3.33 |
-3.22 |
-4.60 |
-3.95 |
-3.76 |
-4.14 |
-1.26 |
-2.45 |
-2.19 |
-1.63 |
0.20 |
0.12 |
1.75 |
-0.18 |
1.95 |
6 |
0.00 |
0.01 |
0.06 |
0.08 |
0.08 |
0.05 |
0.02 |
0.01 |
0.01 |
-0.04 |
-0.05 |
-0.04 |
-0.03 |
-0.04 |
-0.04 |
-0.03 |
-0.01 |
-0.03 |
-0.03 |
-0.03 |
-0.01 |
-0.02 |
-0.00 |
-0.02 |
-0.01 |
| GM.5.0.Mixed.0051 |
FEV,ETS2,ERF,ELK1,GABPA, (...) |
|
0.10 |
-2.82 |
-1.27 |
-1.98 |
-2.26 |
-1.32 |
-0.43 |
2.06 |
2.07 |
1.83 |
1.05 |
1.33 |
3.36 |
1.92 |
1.43 |
2.81 |
-0.54 |
1.16 |
0.17 |
1.07 |
-0.46 |
1.97 |
-0.91 |
-1.18 |
-2.33 |
1 |
-0.05 |
-0.05 |
-0.02 |
-0.00 |
-0.02 |
-0.01 |
-0.02 |
0.00 |
-0.00 |
-0.01 |
-0.02 |
0.01 |
0.01 |
0.02 |
0.01 |
0.04 |
0.02 |
0.03 |
0.02 |
0.04 |
0.04 |
0.03 |
0.01 |
0.01 |
-0.01 |
| GM.5.0.C2H2_ZF.0178 |
PRDM1 |
|
-1.90 |
-2.26 |
0.02 |
-1.70 |
-1.08 |
-1.04 |
-0.97 |
-0.53 |
-1.21 |
-0.45 |
-0.33 |
1.88 |
0.30 |
2.87 |
1.78 |
3.20 |
1.44 |
2.47 |
1.64 |
1.08 |
1.18 |
0.79 |
1.09 |
0.11 |
0.06 |
<1 |
-0.03 |
-0.03 |
-0.01 |
-0.00 |
-0.01 |
-0.01 |
-0.01 |
0.01 |
0.01 |
0.00 |
-0.00 |
0.02 |
0.02 |
0.02 |
0.02 |
0.02 |
0.00 |
0.02 |
0.01 |
0.01 |
0.01 |
0.00 |
-0.00 |
-0.00 |
-0.01 |
| GM.5.0.Homeodomain.0086 |
VENTX2.2,MSX1,BARX1,NKX6-1,MSX2, (...) |
|
-0.36 |
-0.24 |
1.31 |
0.15 |
-0.45 |
1.01 |
-1.46 |
1.75 |
1.24 |
1.64 |
1.74 |
0.55 |
1.54 |
0.62 |
-0.99 |
-0.89 |
-1.36 |
-0.01 |
-1.30 |
0.16 |
-1.61 |
-1.09 |
-2.19 |
-1.49 |
-3.35 |
<1 |
0.00 |
-0.00 |
0.02 |
0.03 |
0.02 |
0.03 |
0.00 |
0.05 |
0.05 |
0.04 |
0.03 |
0.03 |
0.03 |
0.00 |
-0.00 |
-0.00 |
-0.02 |
-0.01 |
-0.02 |
-0.03 |
-0.03 |
-0.04 |
-0.04 |
-0.06 |
-0.06 |
| GM.5.0.p53.0009 |
TP53 |
|
1.64 |
1.77 |
0.57 |
2.30 |
1.21 |
0.31 |
-0.13 |
-2.97 |
-3.39 |
-2.87 |
-2.91 |
-1.02 |
-0.57 |
-2.78 |
-2.72 |
0.83 |
2.80 |
-0.07 |
-0.47 |
1.19 |
1.27 |
1.54 |
3.02 |
1.03 |
0.32 |
<1 |
-0.00 |
-0.00 |
0.00 |
0.02 |
0.02 |
0.01 |
-0.01 |
-0.02 |
-0.04 |
-0.06 |
-0.06 |
-0.03 |
-0.03 |
-0.02 |
-0.02 |
0.02 |
0.02 |
0.01 |
0.00 |
0.03 |
0.04 |
0.03 |
0.03 |
0.02 |
0.01 |
| GM.5.0.Nuclear_receptor.0109 |
ESRRA,ESRRG,PPARGC1A |
|
1.52 |
2.00 |
1.27 |
0.72 |
1.25 |
1.21 |
1.24 |
1.08 |
1.42 |
1.23 |
0.46 |
1.49 |
1.02 |
-1.29 |
-0.50 |
0.36 |
0.36 |
-1.29 |
-2.96 |
-0.54 |
-1.85 |
-1.29 |
-1.42 |
-3.24 |
-3.41 |
1 |
0.01 |
0.01 |
0.03 |
0.04 |
0.03 |
0.03 |
0.01 |
0.03 |
0.02 |
0.02 |
0.01 |
0.02 |
0.01 |
-0.01 |
-0.01 |
-0.00 |
-0.01 |
-0.03 |
-0.03 |
-0.03 |
-0.04 |
-0.03 |
-0.03 |
-0.04 |
-0.03 |
| GM.5.0.C2H2_ZF.0158 |
ZNF423,ZNF521 |
|
2.04 |
3.15 |
2.10 |
2.50 |
1.42 |
2.12 |
0.86 |
1.32 |
2.24 |
0.21 |
0.36 |
-1.66 |
-0.82 |
-0.93 |
-1.67 |
-2.42 |
0.35 |
-0.95 |
-2.19 |
-2.27 |
-1.40 |
-0.44 |
-0.89 |
-1.89 |
0.10 |
1 |
0.03 |
0.04 |
0.04 |
0.05 |
0.04 |
0.03 |
0.02 |
0.03 |
0.03 |
0.01 |
0.00 |
-0.00 |
-0.00 |
-0.02 |
-0.02 |
-0.03 |
-0.02 |
-0.04 |
-0.04 |
-0.05 |
-0.04 |
-0.03 |
-0.02 |
-0.04 |
-0.03 |
| GM.5.0.bZIP.0096 |
CREM,CREB1,ATF1,ATF2,ATF7, (...) |
|
-2.66 |
-3.26 |
-2.20 |
-3.36 |
-3.65 |
-2.58 |
-0.98 |
-0.19 |
0.56 |
-1.44 |
0.15 |
-0.09 |
-0.15 |
1.87 |
2.26 |
2.02 |
3.38 |
2.83 |
3.11 |
2.68 |
1.81 |
2.69 |
-0.61 |
2.32 |
0.08 |
1 |
-0.04 |
-0.06 |
-0.02 |
-0.02 |
-0.02 |
-0.01 |
-0.02 |
0.00 |
0.00 |
0.00 |
-0.00 |
0.01 |
0.01 |
0.02 |
0.02 |
0.03 |
0.02 |
0.03 |
0.03 |
0.04 |
0.03 |
0.02 |
-0.00 |
0.01 |
-0.00 |
| GM.5.0.Nuclear_receptor.0094 |
ESR1,ESR2,NO ORTHOLOGS FOUND |
|
-2.43 |
-1.93 |
-1.64 |
-2.02 |
-1.80 |
-1.00 |
0.19 |
-2.02 |
-2.65 |
2.51 |
1.76 |
0.98 |
2.42 |
3.15 |
2.60 |
2.17 |
2.86 |
1.90 |
3.04 |
0.56 |
1.64 |
-1.09 |
-0.84 |
0.90 |
-1.11 |
1 |
-0.05 |
-0.04 |
-0.02 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.00 |
-0.01 |
0.02 |
0.01 |
0.03 |
0.03 |
0.03 |
0.03 |
0.04 |
0.01 |
0.03 |
0.02 |
0.02 |
0.01 |
0.00 |
-0.01 |
0.00 |
-0.02 |
| GM.5.0.Unknown.0134 |
NO ORTHOLOGS FOUND |
|
1.46 |
1.86 |
1.94 |
1.89 |
2.33 |
3.20 |
1.66 |
2.00 |
1.93 |
0.69 |
1.42 |
0.58 |
1.50 |
-1.41 |
-1.32 |
-1.10 |
-0.42 |
-3.03 |
-2.57 |
-2.75 |
-2.19 |
-1.89 |
-1.98 |
-3.03 |
-2.64 |
<1 |
0.06 |
0.04 |
0.05 |
0.07 |
0.06 |
0.05 |
0.03 |
0.05 |
0.04 |
0.01 |
0.01 |
0.01 |
0.01 |
-0.03 |
-0.04 |
-0.03 |
-0.02 |
-0.06 |
-0.06 |
-0.07 |
-0.06 |
-0.06 |
-0.05 |
-0.06 |
-0.05 |
| GM.5.0.bHLH.0126 |
SREBF1,SREBF2 |
|
-0.88 |
-1.04 |
0.14 |
-0.89 |
-0.27 |
-2.06 |
-3.11 |
0.50 |
-0.20 |
-0.41 |
0.09 |
-0.00 |
1.57 |
0.62 |
1.45 |
0.87 |
2.02 |
1.12 |
0.57 |
-0.47 |
-0.32 |
0.75 |
0.12 |
-0.61 |
-2.20 |
1 |
-0.03 |
-0.01 |
0.00 |
-0.00 |
0.00 |
-0.01 |
-0.02 |
0.00 |
0.00 |
0.00 |
-0.00 |
0.01 |
0.01 |
0.01 |
0.01 |
0.01 |
0.01 |
0.01 |
0.01 |
0.01 |
0.01 |
0.01 |
-0.00 |
0.00 |
-0.01 |
| GM.5.0.SAND.0001 |
NPAS3,HIF1A,SIM1,GMEB2,XBP1, (...) |
|
2.96 |
3.11 |
3.14 |
2.34 |
2.69 |
1.29 |
0.27 |
-0.54 |
-2.43 |
1.30 |
-2.29 |
-0.42 |
-1.45 |
-1.69 |
-3.40 |
-2.81 |
-2.33 |
-0.97 |
-1.61 |
-2.49 |
-3.49 |
-0.90 |
-0.98 |
-1.21 |
-1.85 |
4 |
0.03 |
0.01 |
0.02 |
0.04 |
0.03 |
0.03 |
0.01 |
0.02 |
0.00 |
0.00 |
-0.01 |
-0.00 |
-0.00 |
-0.02 |
-0.03 |
-0.01 |
-0.02 |
-0.01 |
-0.00 |
-0.03 |
-0.03 |
-0.02 |
-0.02 |
-0.02 |
-0.03 |
| GM.5.0.Homeodomain.0187 |
NO ORTHOLOGS FOUND,HOXD9,HOXA11,HOXA9,HOXB9, (...) |
|
-1.57 |
-1.73 |
-1.90 |
0.06 |
0.48 |
-0.73 |
-0.76 |
-4.02 |
-0.64 |
-1.57 |
-2.25 |
0.54 |
0.43 |
1.86 |
1.83 |
0.64 |
0.57 |
1.39 |
1.57 |
2.08 |
2.32 |
2.22 |
1.01 |
1.71 |
1.13 |
1 |
-0.02 |
-0.02 |
0.00 |
0.02 |
0.02 |
0.01 |
-0.00 |
-0.01 |
-0.00 |
-0.01 |
-0.02 |
0.00 |
0.00 |
0.00 |
0.01 |
0.00 |
-0.00 |
0.01 |
0.01 |
0.01 |
0.01 |
-0.00 |
-0.00 |
0.00 |
-0.01 |
| GM.5.0.C2H2_ZF.0213 |
KLF17,KLF8,ZNF534,KLF2,KLF4, (...) |
|
0.18 |
0.19 |
0.81 |
0.71 |
0.88 |
0.87 |
0.49 |
2.34 |
3.05 |
2.48 |
3.09 |
2.34 |
2.62 |
1.08 |
-1.46 |
-1.38 |
-1.88 |
-1.25 |
-1.95 |
-2.59 |
-2.09 |
-3.07 |
-2.56 |
-3.18 |
-1.68 |
2 |
0.04 |
0.03 |
0.03 |
0.04 |
0.03 |
0.03 |
0.02 |
0.05 |
0.05 |
0.05 |
0.04 |
0.03 |
0.03 |
0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.04 |
-0.04 |
-0.07 |
-0.07 |
-0.06 |
-0.05 |
-0.06 |
-0.05 |
| GM.5.0.C2H2_ZF.0257 |
KLF17,ZNF534,KLF2,KLF4,KLF12, (...) |
|
-0.56 |
-1.05 |
-1.03 |
1.31 |
1.51 |
1.13 |
0.20 |
0.27 |
2.36 |
2.29 |
2.82 |
0.74 |
1.07 |
0.66 |
0.48 |
-0.65 |
-1.79 |
-0.31 |
-0.25 |
-1.48 |
-3.13 |
-2.59 |
-1.28 |
-0.30 |
-0.89 |
1 |
0.03 |
0.02 |
0.02 |
0.03 |
0.03 |
0.02 |
0.01 |
0.03 |
0.04 |
0.05 |
0.05 |
0.03 |
0.02 |
0.00 |
-0.01 |
-0.02 |
-0.04 |
-0.03 |
-0.03 |
-0.06 |
-0.06 |
-0.05 |
-0.04 |
-0.04 |
-0.03 |
| GM.5.0.T-box.0021 |
TBX21,EOMES,TBX1,TBR1 |
|
0.41 |
0.56 |
-1.33 |
-2.37 |
-0.35 |
1.13 |
0.49 |
3.09 |
2.21 |
1.70 |
0.83 |
2.54 |
2.10 |
1.18 |
2.41 |
2.25 |
1.33 |
-0.48 |
-0.35 |
-1.29 |
-0.52 |
-1.88 |
-2.21 |
0.14 |
-1.24 |
1 |
0.01 |
0.00 |
-0.01 |
0.00 |
-0.00 |
0.01 |
-0.00 |
0.03 |
0.03 |
0.03 |
0.03 |
0.04 |
0.03 |
0.02 |
0.01 |
0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.03 |
-0.04 |
-0.03 |
-0.03 |
| GM.5.0.C2H2_ZF.0254 |
RAD21,CTCF |
|
-1.92 |
-1.82 |
-2.49 |
-3.03 |
-2.66 |
-1.74 |
-0.39 |
-0.25 |
0.27 |
1.46 |
1.99 |
1.27 |
0.88 |
2.02 |
1.13 |
1.03 |
2.05 |
2.48 |
1.01 |
1.13 |
0.60 |
0.22 |
0.86 |
-0.96 |
0.98 |
1 |
-0.05 |
-0.04 |
-0.02 |
-0.03 |
-0.02 |
-0.01 |
-0.01 |
-0.00 |
-0.00 |
0.01 |
0.01 |
0.01 |
0.01 |
0.02 |
0.02 |
0.02 |
0.02 |
0.02 |
0.02 |
0.03 |
0.03 |
0.01 |
0.01 |
0.01 |
0.01 |
| GM.5.0.p53.0004 |
TP63,TP73,TP53,MDM4,TPRG1L, (...) |
|
-0.68 |
-2.01 |
-3.52 |
-1.20 |
-1.74 |
-1.08 |
-2.27 |
-4.28 |
-3.57 |
-3.66 |
-3.44 |
-1.17 |
-0.82 |
1.14 |
1.27 |
1.77 |
2.55 |
1.85 |
1.59 |
4.27 |
4.30 |
3.46 |
3.87 |
3.17 |
2.32 |
1 |
-0.00 |
-0.01 |
-0.01 |
0.01 |
0.00 |
0.00 |
-0.01 |
-0.03 |
-0.04 |
-0.05 |
-0.06 |
-0.03 |
-0.02 |
-0.01 |
-0.01 |
0.01 |
0.02 |
0.01 |
0.01 |
0.04 |
0.05 |
0.04 |
0.03 |
0.03 |
0.01 |
| GM.5.0.C2H2_ZF.0193 |
HBP1,SOX8,BBX,SOX2,SOX3, (...) |
|
-0.40 |
-0.80 |
-1.99 |
-1.49 |
-2.02 |
-0.68 |
-0.65 |
2.11 |
2.11 |
1.21 |
1.05 |
1.31 |
1.97 |
1.26 |
1.39 |
0.21 |
-0.22 |
0.47 |
1.28 |
0.92 |
-0.93 |
-0.76 |
-4.20 |
0.08 |
0.76 |
2 |
0.02 |
0.00 |
0.01 |
0.02 |
0.01 |
0.01 |
0.00 |
0.02 |
0.02 |
0.01 |
0.00 |
0.01 |
0.01 |
0.00 |
-0.01 |
-0.00 |
-0.01 |
-0.00 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.04 |
-0.01 |
-0.01 |
| GM.5.0.Homeodomain.0042 |
OTP,DRGX,ALX1,VSX2,TRAF4, (...) |
|
1.68 |
0.75 |
-1.38 |
-0.13 |
-0.82 |
0.59 |
3.06 |
4.01 |
4.90 |
2.90 |
4.38 |
2.29 |
3.25 |
1.42 |
0.11 |
-2.02 |
-1.45 |
-2.97 |
-2.62 |
-3.39 |
-3.72 |
-3.85 |
-2.75 |
-3.59 |
-2.65 |
2 |
0.00 |
-0.01 |
0.00 |
0.02 |
0.01 |
0.02 |
0.01 |
0.07 |
0.07 |
0.06 |
0.05 |
0.04 |
0.03 |
0.02 |
0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.03 |
-0.05 |
-0.05 |
-0.04 |
-0.04 |
-0.06 |
-0.05 |
| GM.5.0.Sox.0004 |
SOX18,SOX17A,LOC116407953,SOX6,SOX14, (...) |
|
-0.26 |
-0.19 |
0.75 |
-0.79 |
-1.77 |
-0.53 |
1.77 |
3.76 |
3.57 |
2.92 |
3.45 |
3.21 |
2.43 |
1.80 |
0.78 |
-3.50 |
-4.44 |
-1.51 |
-1.58 |
-1.31 |
-2.03 |
-3.67 |
-4.02 |
-2.59 |
-3.75 |
5 |
0.03 |
0.01 |
0.01 |
0.02 |
0.00 |
0.01 |
0.01 |
0.05 |
0.05 |
0.07 |
0.07 |
0.05 |
0.04 |
0.01 |
-0.00 |
-0.03 |
-0.04 |
-0.03 |
-0.02 |
-0.04 |
-0.06 |
-0.05 |
-0.06 |
-0.06 |
-0.06 |
| GM.5.0.C2H2_ZF.0099 |
NO ORTHOLOGS FOUND,SP2,TCF7L2,SP8,PRDM1, (...) |
|
-1.13 |
-0.68 |
-0.66 |
-1.17 |
-0.89 |
0.35 |
1.49 |
2.64 |
2.20 |
3.27 |
2.95 |
3.44 |
3.63 |
0.27 |
0.99 |
-0.99 |
-1.40 |
-1.80 |
-1.51 |
-2.90 |
-1.78 |
-1.51 |
-1.96 |
-1.64 |
-0.92 |
<1 |
-0.02 |
-0.02 |
0.01 |
0.02 |
0.01 |
0.03 |
0.01 |
0.05 |
0.06 |
0.05 |
0.04 |
0.05 |
0.05 |
0.03 |
0.02 |
0.00 |
-0.01 |
-0.01 |
-0.02 |
-0.05 |
-0.05 |
-0.04 |
-0.05 |
-0.05 |
-0.06 |
| GM.5.0.bHLH.0135 |
NHLH1,LYL1,NHLH2,TAL1,TFAP4, (...) |
|
-3.39 |
-2.81 |
0.34 |
-1.81 |
-1.84 |
-2.06 |
-3.02 |
-3.11 |
-3.61 |
0.87 |
1.76 |
1.58 |
2.56 |
2.74 |
2.97 |
2.89 |
0.04 |
3.30 |
3.71 |
0.77 |
0.34 |
1.69 |
-0.27 |
1.34 |
-0.37 |
1 |
-0.05 |
-0.04 |
0.00 |
0.01 |
0.00 |
-0.01 |
-0.03 |
-0.02 |
-0.02 |
-0.00 |
-0.01 |
0.02 |
0.02 |
0.02 |
0.02 |
0.04 |
0.01 |
0.05 |
0.04 |
0.02 |
0.01 |
0.00 |
-0.01 |
0.00 |
-0.01 |
| GM.5.0.C2H2_ZF.0208 |
TBX21,TBX1,NO ORTHOLOGS FOUND |
|
0.60 |
0.96 |
-0.68 |
-0.88 |
-1.43 |
-0.15 |
0.70 |
2.26 |
2.63 |
3.47 |
2.99 |
3.64 |
1.99 |
2.07 |
2.39 |
-0.90 |
-2.45 |
-0.97 |
-1.72 |
-0.89 |
-2.11 |
-2.77 |
-2.50 |
-3.27 |
-2.33 |
2 |
0.03 |
0.02 |
0.01 |
0.01 |
0.00 |
0.02 |
0.01 |
0.03 |
0.03 |
0.04 |
0.04 |
0.04 |
0.03 |
0.01 |
0.00 |
-0.01 |
-0.03 |
-0.02 |
-0.03 |
-0.04 |
-0.05 |
-0.04 |
-0.04 |
-0.04 |
-0.03 |
| GM.5.0.E2F.0002 |
E2F5,E2F7,TFDP1,E2F1,E2F4, (...) |
|
-0.97 |
-3.36 |
-3.36 |
-3.54 |
-3.96 |
-1.83 |
-2.07 |
1.78 |
0.92 |
1.91 |
1.98 |
3.13 |
2.66 |
2.61 |
2.95 |
3.00 |
2.26 |
2.39 |
2.00 |
2.40 |
0.01 |
-0.30 |
0.08 |
-1.39 |
-2.41 |
<1 |
-0.04 |
-0.05 |
-0.02 |
-0.01 |
-0.02 |
-0.00 |
-0.01 |
0.02 |
0.02 |
0.03 |
0.01 |
0.04 |
0.04 |
0.03 |
0.03 |
0.04 |
0.01 |
0.03 |
0.02 |
0.01 |
0.00 |
-0.01 |
-0.02 |
-0.01 |
-0.03 |
| GM.5.0.bHLH.0032 |
TCF12,NHLH1,LYL1,TCF3,TAL1, (...) |
|
-2.62 |
-2.52 |
-2.45 |
-3.60 |
-2.93 |
-3.47 |
-3.13 |
-3.82 |
-2.48 |
-0.53 |
1.78 |
4.11 |
3.80 |
2.75 |
2.89 |
3.03 |
1.53 |
3.34 |
2.89 |
2.03 |
-0.80 |
1.88 |
-1.58 |
0.54 |
2.06 |
2 |
-0.05 |
-0.04 |
-0.02 |
-0.02 |
-0.01 |
-0.03 |
-0.04 |
-0.03 |
-0.02 |
0.00 |
0.01 |
0.04 |
0.04 |
0.03 |
0.03 |
0.04 |
0.01 |
0.05 |
0.05 |
0.03 |
0.01 |
0.01 |
-0.01 |
0.01 |
0.01 |
| GM.5.0.C2H2_ZF.0107 |
ZNF524,LOC101730373 |
|
2.14 |
0.94 |
2.70 |
2.92 |
1.96 |
2.39 |
1.65 |
0.89 |
0.12 |
-1.71 |
-1.15 |
-1.87 |
-1.43 |
-3.26 |
-3.38 |
-2.26 |
0.44 |
-1.95 |
-2.18 |
-2.07 |
-0.48 |
0.66 |
2.22 |
-1.11 |
-0.99 |
1 |
0.02 |
0.03 |
0.04 |
0.06 |
0.05 |
0.04 |
0.03 |
0.02 |
0.02 |
-0.01 |
-0.02 |
-0.01 |
-0.01 |
-0.03 |
-0.04 |
-0.02 |
-0.01 |
-0.04 |
-0.04 |
-0.04 |
-0.03 |
-0.02 |
-0.01 |
-0.03 |
-0.02 |
| GM.5.0.Homeodomain_POU.0019 |
POU3F2,POU3F3,POU3F4,POU3F1,POU1F1, (...) |
|
4.34 |
4.34 |
5.23 |
4.61 |
4.40 |
4.87 |
3.67 |
2.20 |
0.98 |
-1.42 |
-1.02 |
-2.76 |
-3.15 |
-4.72 |
-4.49 |
-3.85 |
-3.03 |
-4.54 |
-4.09 |
-4.19 |
-4.06 |
-5.14 |
-3.21 |
-3.02 |
-0.15 |
2 |
0.13 |
0.10 |
0.08 |
0.10 |
0.08 |
0.08 |
0.06 |
0.04 |
0.03 |
-0.01 |
-0.02 |
-0.03 |
-0.03 |
-0.07 |
-0.06 |
-0.06 |
-0.04 |
-0.10 |
-0.10 |
-0.10 |
-0.08 |
-0.08 |
-0.04 |
-0.06 |
-0.04 |
| GM.5.0.Homeodomain.0174 |
TGIF2,TGIF1,MEIS2,MEIS1,MEIS3, (...) |
|
-3.44 |
-3.71 |
-3.46 |
-3.19 |
-3.06 |
-4.11 |
-4.53 |
-4.17 |
-4.12 |
-1.73 |
-0.62 |
-0.76 |
0.13 |
2.30 |
2.95 |
3.93 |
4.17 |
4.48 |
4.35 |
4.41 |
4.68 |
3.95 |
3.46 |
4.05 |
4.26 |
2 |
-0.08 |
-0.07 |
-0.04 |
-0.04 |
-0.04 |
-0.04 |
-0.06 |
-0.04 |
-0.04 |
-0.02 |
-0.03 |
0.01 |
0.01 |
0.04 |
0.03 |
0.06 |
0.03 |
0.07 |
0.06 |
0.08 |
0.07 |
0.05 |
0.02 |
0.05 |
0.02 |
| GM.5.0.Unknown.0108 |
LOC101734178,ETV6,ELF2,ELF1,ETS2, (...) |
|
2.85 |
2.66 |
2.06 |
1.16 |
1.91 |
1.55 |
1.56 |
0.63 |
2.28 |
0.33 |
0.69 |
-0.61 |
-1.51 |
-1.16 |
-1.15 |
-2.63 |
-2.40 |
-2.42 |
-2.74 |
-3.29 |
-2.22 |
-2.88 |
-1.75 |
-0.86 |
-0.17 |
1 |
0.02 |
0.01 |
0.02 |
0.03 |
0.03 |
0.03 |
0.01 |
0.02 |
0.02 |
-0.00 |
-0.01 |
-0.00 |
-0.00 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.03 |
-0.03 |
-0.03 |
-0.02 |
-0.02 |
-0.02 |
-0.01 |
-0.02 |
| GM.5.0.C2H2_ZF.0228 |
SP8,SP3,SP6,SP5,SP4, (...) |
|
1.52 |
0.77 |
1.00 |
1.80 |
0.36 |
2.22 |
3.07 |
2.61 |
0.92 |
0.20 |
-0.85 |
1.32 |
0.56 |
-0.56 |
-1.23 |
-0.02 |
-0.05 |
-1.16 |
-0.70 |
-1.45 |
-0.91 |
-2.35 |
-2.69 |
-2.29 |
-2.60 |
2 |
0.02 |
0.00 |
0.02 |
0.04 |
0.02 |
0.04 |
0.03 |
0.04 |
0.03 |
0.01 |
-0.00 |
0.02 |
0.01 |
0.00 |
-0.01 |
-0.00 |
-0.01 |
-0.02 |
-0.02 |
-0.04 |
-0.03 |
-0.04 |
-0.04 |
-0.05 |
-0.05 |
| GM.5.0.Sox.0015 |
TCF7L2,TCF7L1,TCF7,LEF1,TCF12, (...) |
|
-4.32 |
-4.56 |
-4.24 |
-4.12 |
-4.06 |
-2.74 |
-1.98 |
3.39 |
2.62 |
4.15 |
4.20 |
5.00 |
5.64 |
5.03 |
5.15 |
3.25 |
-0.15 |
2.16 |
0.67 |
0.58 |
-1.27 |
-0.62 |
-2.13 |
-2.23 |
-2.78 |
1 |
-0.06 |
-0.05 |
-0.03 |
-0.03 |
-0.03 |
-0.01 |
-0.01 |
0.02 |
0.03 |
0.05 |
0.04 |
0.07 |
0.06 |
0.06 |
0.05 |
0.03 |
-0.00 |
0.01 |
0.00 |
0.01 |
-0.00 |
-0.01 |
-0.03 |
-0.01 |
-0.02 |
| GM.5.0.Homeodomain.0034 |
PITX3,PITX1,PITX2,OTX1,DRGX, (...) |
|
-3.63 |
-3.17 |
-3.14 |
-3.72 |
-2.29 |
-2.64 |
-2.92 |
2.09 |
2.57 |
4.20 |
3.61 |
4.09 |
2.95 |
3.11 |
3.90 |
3.25 |
1.84 |
2.85 |
2.20 |
-1.55 |
-1.82 |
-0.81 |
-2.48 |
-1.87 |
-1.30 |
<1 |
-0.04 |
-0.04 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
0.03 |
0.04 |
0.05 |
0.04 |
0.05 |
0.04 |
0.03 |
0.03 |
0.02 |
0.01 |
0.02 |
0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
| GM.5.0.Homeodomain.0069 |
HMX3,BARHL1,MSX1,BARX1,SOHO1, (...) |
|
-3.19 |
-3.42 |
-3.17 |
-1.25 |
-2.25 |
-3.11 |
-2.65 |
-0.87 |
-0.29 |
1.87 |
-0.48 |
1.99 |
2.40 |
1.70 |
1.04 |
1.67 |
1.23 |
1.88 |
2.79 |
2.42 |
2.29 |
2.70 |
1.68 |
1.50 |
1.96 |
<1 |
-0.03 |
-0.03 |
-0.01 |
0.00 |
-0.00 |
-0.01 |
-0.01 |
0.02 |
0.03 |
0.04 |
0.03 |
0.03 |
0.03 |
0.02 |
0.01 |
0.01 |
-0.00 |
0.01 |
0.00 |
0.00 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.03 |
| GM.5.0.T-box.0019 |
TBX2,ZEB1,TBX3,ZEB2,TBR1, (...) |
|
2.87 |
2.88 |
1.36 |
0.19 |
1.23 |
1.47 |
3.91 |
3.41 |
1.88 |
1.45 |
1.93 |
1.67 |
1.18 |
-0.79 |
-2.48 |
-2.90 |
-4.03 |
-3.90 |
-2.82 |
-2.14 |
-1.53 |
-3.12 |
-1.97 |
-3.66 |
-3.80 |
<1 |
0.02 |
0.01 |
0.01 |
0.02 |
0.02 |
0.02 |
0.02 |
0.02 |
0.02 |
0.01 |
0.00 |
0.02 |
0.01 |
-0.00 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.03 |
-0.02 |
-0.03 |
-0.03 |
| GM.5.0.Nuclear_receptor.0043 |
FOXL2,FOXA2,FOXL1,FOXA1,FOXQ1, (...) |
|
-0.33 |
-0.64 |
-1.78 |
-1.02 |
-3.05 |
-0.96 |
0.12 |
0.31 |
2.04 |
2.37 |
2.14 |
2.05 |
2.81 |
1.60 |
1.97 |
-0.36 |
-1.00 |
0.68 |
0.22 |
0.34 |
0.89 |
0.57 |
-0.88 |
0.03 |
-1.76 |
1 |
0.02 |
0.01 |
-0.01 |
0.00 |
-0.01 |
0.00 |
0.00 |
0.02 |
0.03 |
0.04 |
0.04 |
0.04 |
0.04 |
0.02 |
0.01 |
-0.01 |
-0.03 |
-0.00 |
-0.00 |
-0.02 |
-0.03 |
-0.03 |
-0.04 |
-0.03 |
-0.03 |
| GM.5.0.C2H2_ZF.0251 |
NO ORTHOLOGS FOUND |
|
-0.94 |
-0.11 |
-0.17 |
-0.68 |
-1.44 |
-2.07 |
-2.19 |
-3.82 |
-2.89 |
-1.40 |
-2.00 |
-1.63 |
-1.36 |
0.56 |
0.14 |
0.81 |
2.33 |
2.19 |
2.43 |
2.60 |
1.11 |
2.49 |
1.92 |
1.88 |
2.61 |
1 |
-0.00 |
-0.00 |
0.01 |
0.01 |
0.00 |
-0.00 |
-0.01 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.02 |
-0.02 |
-0.01 |
-0.01 |
0.00 |
0.01 |
0.01 |
0.01 |
0.02 |
0.03 |
0.01 |
0.01 |
0.02 |
0.02 |
| GM.5.0.Homeodomain.0180 |
HOXA6,HOXB6,HOXD9,HOXA11,HOXC4, (...) |
|
-2.49 |
-2.16 |
-0.38 |
-0.93 |
-1.89 |
-1.96 |
-3.83 |
-1.33 |
-1.97 |
-2.43 |
-1.48 |
-0.40 |
0.16 |
1.42 |
1.62 |
1.72 |
2.21 |
3.85 |
3.87 |
3.82 |
2.31 |
1.08 |
1.08 |
2.04 |
2.51 |
1 |
-0.03 |
-0.02 |
0.02 |
0.01 |
0.01 |
-0.01 |
-0.02 |
-0.01 |
-0.00 |
-0.01 |
-0.02 |
0.00 |
0.01 |
0.01 |
0.00 |
0.02 |
0.01 |
0.02 |
0.02 |
0.01 |
0.01 |
0.00 |
-0.00 |
0.00 |
-0.01 |
| GM.5.0.Forkhead.0015 |
FOXE3,FOXL2,FOXI1,FOXB2,FOXF2, (...) |
|
3.59 |
2.61 |
0.25 |
-2.77 |
-3.83 |
1.67 |
3.41 |
2.76 |
3.17 |
1.99 |
0.38 |
-0.01 |
1.18 |
-1.74 |
-1.56 |
-1.30 |
-1.42 |
-1.66 |
-2.39 |
-2.77 |
-2.30 |
0.79 |
0.39 |
-1.16 |
-2.81 |
2 |
0.05 |
0.03 |
0.01 |
0.00 |
-0.01 |
0.02 |
0.02 |
0.03 |
0.03 |
0.03 |
0.03 |
0.02 |
0.02 |
-0.00 |
-0.01 |
-0.02 |
-0.03 |
-0.03 |
-0.02 |
-0.03 |
-0.05 |
-0.02 |
-0.03 |
-0.04 |
-0.04 |
| GM.5.0.bHLH.0084 |
TAL1 |
|
-4.39 |
-4.44 |
-4.24 |
-4.77 |
-3.57 |
-4.21 |
-3.62 |
-0.99 |
0.86 |
4.56 |
4.07 |
3.68 |
3.70 |
2.76 |
4.28 |
2.92 |
1.06 |
3.37 |
3.87 |
3.67 |
3.13 |
1.38 |
-0.42 |
2.36 |
-0.02 |
<1 |
-0.11 |
-0.09 |
-0.07 |
-0.08 |
-0.07 |
-0.05 |
-0.05 |
-0.00 |
0.01 |
0.08 |
0.07 |
0.07 |
0.06 |
0.08 |
0.07 |
0.05 |
0.01 |
0.06 |
0.05 |
0.06 |
0.03 |
0.02 |
-0.00 |
0.01 |
-0.01 |
| GM.5.0.Mixed.0048 |
SIX3,SIX6,SIX4,ETS1,ZNF143 |
|
-0.03 |
0.12 |
0.44 |
1.12 |
0.78 |
0.66 |
0.65 |
2.38 |
2.00 |
1.02 |
0.48 |
1.28 |
0.17 |
0.05 |
-0.88 |
0.69 |
0.72 |
-1.46 |
-0.68 |
-0.35 |
-0.33 |
-3.06 |
-3.51 |
-1.16 |
-1.93 |
1 |
0.02 |
0.01 |
0.02 |
0.03 |
0.02 |
0.02 |
0.01 |
0.03 |
0.02 |
0.01 |
-0.00 |
0.01 |
0.01 |
-0.00 |
-0.01 |
-0.00 |
-0.00 |
-0.01 |
-0.01 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.02 |
-0.03 |
| GM.5.0.Unknown.0109 |
NO ORTHOLOGS FOUND |
|
2.20 |
0.90 |
-0.41 |
-1.76 |
-2.01 |
-0.96 |
-0.41 |
3.97 |
4.49 |
5.44 |
4.63 |
5.19 |
4.12 |
1.63 |
0.45 |
-2.53 |
-4.10 |
-1.51 |
-1.52 |
-2.63 |
-3.08 |
-2.14 |
-4.88 |
-4.04 |
-4.09 |
2 |
0.06 |
0.04 |
0.01 |
0.01 |
-0.00 |
0.01 |
0.02 |
0.06 |
0.06 |
0.09 |
0.09 |
0.06 |
0.05 |
0.01 |
-0.01 |
-0.04 |
-0.06 |
-0.04 |
-0.04 |
-0.06 |
-0.08 |
-0.06 |
-0.06 |
-0.07 |
-0.06 |
| GM.5.0.bHLH.0028 |
TCF12,TCF3,TCF4,MYF5,MYOD1, (...) |
|
-2.60 |
-3.09 |
-1.94 |
-3.26 |
-2.89 |
-3.29 |
-4.00 |
-2.38 |
-1.60 |
1.67 |
2.16 |
3.03 |
1.87 |
2.99 |
3.16 |
3.54 |
1.17 |
3.52 |
2.31 |
2.39 |
0.27 |
0.76 |
-1.84 |
2.00 |
2.40 |
3 |
-0.06 |
-0.05 |
-0.02 |
-0.01 |
-0.01 |
-0.03 |
-0.03 |
-0.02 |
-0.02 |
-0.00 |
-0.01 |
0.03 |
0.02 |
0.03 |
0.03 |
0.05 |
0.02 |
0.06 |
0.05 |
0.04 |
0.03 |
0.02 |
-0.01 |
0.02 |
0.01 |
| GM.5.0.Homeodomain.0068 |
ISL2,NKX2-5,NKX2-3,TLX3,TLX2, (...) |
|
-2.06 |
-1.87 |
-2.60 |
-1.79 |
-2.34 |
0.16 |
-1.14 |
-0.08 |
1.23 |
3.24 |
2.19 |
1.06 |
-0.15 |
0.93 |
0.36 |
-0.84 |
-0.34 |
0.22 |
1.50 |
0.26 |
1.22 |
1.56 |
2.76 |
-0.25 |
-0.68 |
6 |
-0.01 |
-0.01 |
0.00 |
0.02 |
0.01 |
0.01 |
-0.00 |
0.03 |
0.04 |
0.04 |
0.03 |
0.03 |
0.02 |
0.01 |
0.00 |
-0.01 |
-0.02 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.03 |
-0.04 |
| GM.5.0.C2H2_ZF.0108 |
GLIS3,ZIC4,LOC100493456,ZIC5,GLIS2, (...) |
|
2.72 |
2.98 |
0.18 |
0.00 |
-2.02 |
2.34 |
2.58 |
4.78 |
4.54 |
4.04 |
4.94 |
4.21 |
3.61 |
4.05 |
3.06 |
1.07 |
-3.13 |
-2.38 |
-3.84 |
-4.18 |
-4.94 |
-3.20 |
-5.37 |
-4.04 |
-4.42 |
2 |
0.01 |
0.01 |
0.01 |
0.02 |
0.00 |
0.03 |
0.02 |
0.07 |
0.08 |
0.09 |
0.08 |
0.07 |
0.07 |
0.05 |
0.04 |
0.00 |
-0.03 |
-0.03 |
-0.04 |
-0.09 |
-0.09 |
-0.07 |
-0.08 |
-0.09 |
-0.08 |
| GM.5.0.C2H2_ZF.0172 |
ZBTB47,ZNF652,MYBL1,MYBL2 |
|
-1.28 |
-1.58 |
-1.51 |
-2.48 |
-2.40 |
-2.62 |
-1.70 |
0.06 |
0.06 |
1.17 |
1.97 |
2.36 |
2.91 |
3.06 |
1.50 |
0.98 |
-0.00 |
0.62 |
1.03 |
1.43 |
1.02 |
0.54 |
-0.10 |
0.05 |
-0.65 |
2 |
-0.03 |
-0.03 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
-0.01 |
0.02 |
0.02 |
0.04 |
0.03 |
0.04 |
0.03 |
0.03 |
0.02 |
0.01 |
-0.01 |
0.00 |
-0.00 |
-0.00 |
-0.01 |
-0.01 |
-0.02 |
-0.01 |
-0.02 |
| GM.5.0.Forkhead.0007 |
FOXO6,FOXO4,FOXO3,FOXE3,FOXL2, (...) |
|
1.50 |
-1.08 |
-3.12 |
-4.05 |
-4.15 |
-4.44 |
-4.21 |
2.87 |
2.31 |
4.45 |
4.15 |
4.11 |
3.41 |
4.72 |
3.22 |
0.21 |
-3.88 |
0.81 |
3.57 |
0.24 |
-3.28 |
-1.50 |
-3.98 |
-2.35 |
-2.88 |
<1 |
0.03 |
0.00 |
-0.02 |
-0.02 |
-0.03 |
-0.02 |
-0.01 |
0.03 |
0.03 |
0.07 |
0.08 |
0.07 |
0.06 |
0.04 |
0.02 |
-0.00 |
-0.04 |
0.00 |
0.01 |
-0.02 |
-0.05 |
-0.03 |
-0.05 |
-0.04 |
-0.04 |
| GM.5.0.Nuclear_receptor.0104 |
RXRG,RAD21,RXRA,RXRB |
|
-2.17 |
-3.88 |
-4.31 |
-3.29 |
-2.73 |
-2.42 |
-2.00 |
1.44 |
1.36 |
0.81 |
2.87 |
3.42 |
3.84 |
3.46 |
4.35 |
2.52 |
1.97 |
1.95 |
2.10 |
2.94 |
-0.06 |
1.95 |
0.37 |
-0.03 |
-2.12 |
<1 |
-0.03 |
-0.05 |
-0.03 |
-0.02 |
-0.02 |
-0.00 |
-0.01 |
0.02 |
0.02 |
0.02 |
0.02 |
0.03 |
0.03 |
0.03 |
0.03 |
0.03 |
0.00 |
0.02 |
0.02 |
0.01 |
0.00 |
0.00 |
-0.01 |
-0.00 |
-0.02 |
| GM.5.0.Homeodomain.0158 |
ISL2,HOXA6,HOXB6,NKX2-3,NKX2-6, (...) |
|
-0.44 |
-0.33 |
-2.28 |
-1.42 |
-1.96 |
-2.57 |
-1.26 |
2.24 |
2.70 |
2.58 |
3.25 |
3.01 |
3.61 |
2.63 |
2.66 |
0.39 |
-2.20 |
-0.50 |
0.12 |
-0.87 |
-1.32 |
-0.71 |
-0.15 |
-1.51 |
-2.47 |
2 |
0.00 |
-0.01 |
-0.00 |
0.00 |
-0.00 |
0.01 |
0.00 |
0.06 |
0.07 |
0.06 |
0.05 |
0.05 |
0.04 |
0.02 |
0.01 |
-0.00 |
-0.02 |
-0.01 |
-0.02 |
-0.04 |
-0.04 |
-0.03 |
-0.03 |
-0.05 |
-0.05 |
| GM.5.0.C2H2_ZF.0133 |
NO ORTHOLOGS FOUND,MAZ,SP1,VEZF1,STAT3.1, (...) |
|
-2.84 |
-3.70 |
-0.81 |
-0.84 |
-0.89 |
-1.03 |
-1.34 |
1.35 |
2.37 |
3.24 |
2.17 |
3.84 |
3.60 |
4.42 |
4.01 |
2.41 |
-0.37 |
1.32 |
0.64 |
0.66 |
-2.02 |
0.70 |
-1.71 |
-1.94 |
-2.40 |
2 |
-0.03 |
-0.04 |
-0.01 |
0.00 |
-0.01 |
0.01 |
0.00 |
0.05 |
0.05 |
0.06 |
0.04 |
0.06 |
0.06 |
0.05 |
0.03 |
0.02 |
-0.01 |
0.00 |
-0.01 |
-0.02 |
-0.04 |
-0.03 |
-0.05 |
-0.04 |
-0.05 |
| GM.5.0.AP-2.0002 |
TFAP2A,TFAP2E,TFAP2D,TFAP2C,TFAP2B, (...) |
|
-5.12 |
-5.10 |
-5.41 |
-2.66 |
-2.62 |
-1.72 |
-4.02 |
-4.24 |
-3.69 |
-4.28 |
-5.49 |
-3.41 |
-4.71 |
4.44 |
4.95 |
5.57 |
5.33 |
5.33 |
4.06 |
5.49 |
5.57 |
4.86 |
5.04 |
4.57 |
0.99 |
2 |
-0.07 |
-0.05 |
-0.02 |
-0.00 |
0.00 |
-0.00 |
-0.01 |
-0.01 |
-0.02 |
-0.06 |
-0.08 |
-0.03 |
-0.03 |
0.03 |
0.04 |
0.06 |
0.06 |
0.04 |
0.02 |
0.06 |
0.09 |
0.04 |
0.03 |
0.02 |
-0.01 |
| GM.5.0.GATA.0027 |
GATA1,GATA3,GATA2,BCL3,SIX3, (...) |
|
-2.18 |
-1.40 |
-2.46 |
-2.48 |
-2.91 |
-3.20 |
-3.24 |
1.45 |
0.86 |
3.21 |
2.81 |
3.08 |
3.25 |
2.47 |
3.02 |
-0.55 |
-2.46 |
0.38 |
0.39 |
1.14 |
0.36 |
-0.96 |
-1.02 |
-0.30 |
-0.17 |
1 |
-0.08 |
-0.06 |
-0.04 |
-0.05 |
-0.04 |
-0.04 |
-0.03 |
0.02 |
0.02 |
0.07 |
0.07 |
0.07 |
0.06 |
0.07 |
0.06 |
0.03 |
-0.00 |
0.03 |
0.02 |
0.03 |
0.01 |
-0.00 |
-0.02 |
-0.01 |
-0.02 |
| GM.5.0.C2H2_ZF.0094 |
MTF1,NO ORTHOLOGS FOUND |
|
0.71 |
0.77 |
0.23 |
0.92 |
1.00 |
-0.43 |
1.60 |
0.69 |
-0.42 |
-1.00 |
-0.27 |
0.26 |
1.23 |
-0.94 |
-0.94 |
-2.50 |
-3.44 |
-0.59 |
-0.09 |
1.25 |
-1.38 |
0.03 |
-1.39 |
-0.93 |
0.98 |
1 |
0.07 |
0.06 |
0.04 |
0.06 |
0.05 |
0.04 |
0.03 |
0.02 |
0.02 |
0.00 |
0.01 |
0.00 |
0.00 |
-0.03 |
-0.04 |
-0.05 |
-0.04 |
-0.05 |
-0.04 |
-0.06 |
-0.06 |
-0.04 |
-0.03 |
-0.04 |
-0.02 |
| GM.5.0.TEA.0006 |
TEAD1,TEAD4,NO ORTHOLOGS FOUND |
|
-3.29 |
-1.52 |
-1.93 |
-2.65 |
-1.78 |
-3.85 |
-3.43 |
-3.77 |
-4.13 |
-5.03 |
-4.24 |
-3.72 |
-3.46 |
-1.97 |
0.77 |
3.29 |
4.94 |
4.06 |
3.03 |
4.78 |
4.85 |
4.54 |
4.81 |
4.43 |
5.64 |
2 |
-0.01 |
-0.01 |
-0.00 |
-0.00 |
-0.01 |
-0.01 |
-0.02 |
-0.04 |
-0.05 |
-0.07 |
-0.08 |
-0.05 |
-0.05 |
-0.03 |
-0.01 |
0.02 |
0.03 |
0.03 |
0.02 |
0.06 |
0.07 |
0.04 |
0.04 |
0.06 |
0.04 |
| GM.5.0.T-box.0014 |
MGA,TBX21,TBX4,TBX2,TBX3, (...) |
|
1.46 |
-0.57 |
-2.40 |
-1.95 |
-1.97 |
-2.21 |
-0.30 |
1.66 |
1.46 |
0.67 |
3.07 |
2.35 |
3.52 |
3.46 |
3.12 |
2.02 |
0.14 |
1.60 |
-0.50 |
-0.17 |
0.58 |
-1.41 |
-3.06 |
-2.12 |
-2.18 |
1 |
0.02 |
0.00 |
-0.01 |
-0.00 |
-0.01 |
-0.00 |
-0.00 |
0.02 |
0.02 |
0.02 |
0.03 |
0.03 |
0.03 |
0.03 |
0.02 |
0.01 |
-0.01 |
-0.00 |
-0.00 |
-0.01 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.03 |
| GM.5.0.Nuclear_receptor.0046 |
PPARD,PPARG,PPARA,NR2F5,NR2F2, (...) |
|
-0.24 |
-0.26 |
0.89 |
-0.01 |
-0.19 |
2.01 |
1.14 |
1.31 |
0.91 |
-2.75 |
-2.76 |
-1.82 |
-1.15 |
-2.59 |
-1.84 |
-1.23 |
-1.04 |
-0.87 |
-0.20 |
0.30 |
0.21 |
1.29 |
2.84 |
3.34 |
3.63 |
2 |
-0.03 |
-0.01 |
0.02 |
0.02 |
0.02 |
0.01 |
-0.00 |
-0.00 |
-0.01 |
-0.03 |
-0.04 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
-0.01 |
-0.00 |
0.00 |
0.00 |
0.01 |
0.02 |
0.01 |
0.01 |
0.03 |
0.03 |
| GM.5.0.Homeodomain_POU.0014 |
SMARCA1,SMARCA5,LOC101731232,LOC100496074,POU2F1, (...) |
|
1.82 |
2.87 |
-1.21 |
-0.35 |
1.12 |
1.60 |
2.17 |
2.58 |
3.00 |
2.84 |
2.16 |
1.43 |
2.19 |
1.74 |
1.02 |
-0.34 |
-2.53 |
-2.01 |
-3.19 |
-1.19 |
-1.05 |
-3.18 |
-2.32 |
-2.87 |
-3.37 |
1 |
0.09 |
0.06 |
0.02 |
0.04 |
0.03 |
0.04 |
0.04 |
0.05 |
0.05 |
0.03 |
0.02 |
0.02 |
0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.04 |
-0.07 |
-0.07 |
-0.07 |
-0.06 |
-0.06 |
-0.05 |
-0.07 |
-0.06 |
| GM.5.0.C2H2_ZF.0148 |
EGR4,EGR1,EGR2,EGR3,RREB1, (...) |
|
-2.51 |
-3.40 |
-2.14 |
-2.03 |
-3.50 |
-0.46 |
0.58 |
2.24 |
3.95 |
3.19 |
2.68 |
2.49 |
1.66 |
3.14 |
3.13 |
3.99 |
0.07 |
-0.34 |
-0.21 |
0.76 |
-1.31 |
-0.76 |
-1.90 |
0.12 |
-1.62 |
2 |
-0.00 |
-0.03 |
-0.01 |
-0.00 |
-0.01 |
0.02 |
0.01 |
0.06 |
0.06 |
0.07 |
0.05 |
0.06 |
0.06 |
0.05 |
0.03 |
0.02 |
-0.01 |
-0.00 |
-0.01 |
-0.04 |
-0.05 |
-0.05 |
-0.06 |
-0.05 |
-0.07 |
| GM.5.0.T-box.0006 |
TBXT,TBXT.2,LOC101732592,TBR1,EOMES |
|
-1.89 |
-2.41 |
-3.15 |
-2.44 |
-3.22 |
-1.28 |
-3.03 |
0.50 |
1.69 |
3.58 |
3.07 |
2.38 |
2.92 |
2.32 |
2.98 |
1.55 |
-0.97 |
1.59 |
1.66 |
2.49 |
1.31 |
-0.71 |
-2.12 |
0.15 |
0.03 |
2 |
-0.00 |
-0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.01 |
-0.01 |
0.02 |
0.02 |
0.02 |
0.03 |
0.03 |
0.03 |
0.02 |
0.02 |
0.01 |
-0.01 |
0.00 |
0.00 |
-0.00 |
-0.01 |
-0.02 |
-0.02 |
-0.02 |
-0.02 |
| GM.5.0.Forkhead.0038 |
FOXA2,FOXA1,FOXE3,FOXL2,FOXI1, (...) |
|
-0.65 |
-1.03 |
-1.61 |
-2.07 |
-2.46 |
-2.72 |
-1.29 |
1.86 |
1.63 |
1.87 |
3.30 |
2.53 |
3.80 |
2.63 |
2.80 |
0.88 |
-2.47 |
1.47 |
2.87 |
1.82 |
-1.19 |
-1.22 |
-1.44 |
-0.74 |
-0.84 |
2 |
0.01 |
-0.00 |
-0.01 |
-0.00 |
-0.01 |
-0.01 |
-0.01 |
0.03 |
0.03 |
0.05 |
0.06 |
0.06 |
0.06 |
0.03 |
0.02 |
-0.00 |
-0.04 |
0.00 |
0.00 |
-0.02 |
-0.04 |
-0.03 |
-0.04 |
-0.04 |
-0.04 |
| GM.5.0.C2H2_ZF.0177 |
TFAP2A,TFAP2E,TFAP2B,ZIC2,TFAP2C, (...) |
|
-0.69 |
1.02 |
3.12 |
3.10 |
1.99 |
1.24 |
2.22 |
1.83 |
3.16 |
1.61 |
0.75 |
1.17 |
1.27 |
1.38 |
0.77 |
1.52 |
0.69 |
-1.57 |
-2.91 |
-2.87 |
-1.88 |
-3.17 |
-1.29 |
-3.88 |
-3.70 |
3 |
-0.01 |
-0.00 |
0.02 |
0.03 |
0.02 |
0.03 |
0.02 |
0.05 |
0.05 |
0.03 |
0.02 |
0.04 |
0.03 |
0.03 |
0.02 |
0.01 |
-0.00 |
-0.02 |
-0.03 |
-0.05 |
-0.04 |
-0.05 |
-0.04 |
-0.06 |
-0.06 |
| GM.5.0.Sox.0019 |
SOX10,SOX9,SOX4,SOX2,SOX3, (...) |
|
2.23 |
-0.36 |
-3.09 |
-1.67 |
-3.14 |
-1.60 |
0.62 |
4.21 |
4.03 |
4.46 |
5.12 |
4.48 |
5.01 |
2.61 |
1.24 |
-2.40 |
-3.49 |
0.97 |
0.36 |
-1.05 |
-3.20 |
-4.07 |
-5.04 |
-3.62 |
-4.18 |
3 |
0.06 |
0.03 |
-0.01 |
-0.00 |
-0.02 |
0.00 |
0.01 |
0.06 |
0.07 |
0.11 |
0.12 |
0.09 |
0.07 |
0.03 |
0.00 |
-0.03 |
-0.06 |
-0.03 |
-0.03 |
-0.06 |
-0.09 |
-0.06 |
-0.08 |
-0.07 |
-0.07 |
| GM.5.0.TEA.0003 |
TEAD1,TEAD4,BCL11B,BCL11A |
|
-4.81 |
-4.19 |
-4.83 |
-5.04 |
-5.25 |
-5.26 |
-5.33 |
-5.19 |
-4.76 |
-4.29 |
-4.54 |
-3.28 |
-3.40 |
2.25 |
1.63 |
3.85 |
4.75 |
5.10 |
5.64 |
5.23 |
5.04 |
5.57 |
4.90 |
5.57 |
5.05 |
2 |
-0.03 |
-0.03 |
-0.02 |
-0.03 |
-0.03 |
-0.03 |
-0.04 |
-0.06 |
-0.06 |
-0.07 |
-0.07 |
-0.05 |
-0.04 |
-0.00 |
0.01 |
0.04 |
0.03 |
0.05 |
0.04 |
0.09 |
0.09 |
0.07 |
0.05 |
0.07 |
0.05 |
| GM.5.0.Forkhead.0042 |
LOC101730723,FOXH1 |
|
6.17 |
6.02 |
3.66 |
2.38 |
1.24 |
2.44 |
2.54 |
-0.88 |
-0.58 |
-2.29 |
-0.82 |
-1.95 |
-3.23 |
-4.96 |
-4.90 |
-3.40 |
-3.14 |
-4.46 |
-4.00 |
-4.61 |
-4.19 |
-3.68 |
-2.92 |
-2.88 |
-1.92 |
4 |
0.16 |
0.12 |
0.04 |
0.04 |
0.02 |
0.03 |
0.02 |
0.02 |
0.02 |
0.00 |
0.01 |
-0.00 |
-0.00 |
-0.04 |
-0.04 |
-0.05 |
-0.04 |
-0.07 |
-0.06 |
-0.08 |
-0.08 |
-0.05 |
-0.04 |
-0.05 |
-0.03 |
| GM.5.0.C2H2_ZF.0128 |
RUNX1,MECOM,RUNX2,RUNX3,PRDM16 |
|
-3.59 |
-3.31 |
-3.36 |
-3.03 |
-3.46 |
-2.71 |
-1.89 |
2.39 |
2.38 |
2.24 |
3.07 |
2.84 |
2.50 |
2.57 |
3.00 |
3.41 |
1.63 |
1.63 |
1.89 |
1.10 |
-0.35 |
1.57 |
1.25 |
-0.24 |
-1.78 |
2 |
-0.09 |
-0.08 |
-0.06 |
-0.05 |
-0.05 |
-0.04 |
-0.03 |
0.01 |
0.01 |
0.06 |
0.05 |
0.06 |
0.05 |
0.07 |
0.05 |
0.05 |
0.02 |
0.04 |
0.03 |
0.05 |
0.02 |
0.02 |
0.00 |
0.01 |
-0.02 |
| GM.5.0.C2H2_ZF.0049 |
ZIC2,FOXA2,BCL11B,ZEB1,NR3C1, (...) |
|
-0.48 |
-1.38 |
-0.77 |
-1.61 |
-0.92 |
-1.73 |
-1.94 |
2.15 |
2.63 |
4.52 |
3.20 |
3.95 |
3.88 |
4.43 |
3.64 |
2.00 |
-0.70 |
0.41 |
-0.88 |
-2.81 |
-2.82 |
-3.63 |
-3.72 |
-4.07 |
-4.46 |
<1 |
-0.01 |
-0.01 |
-0.00 |
0.01 |
-0.00 |
0.00 |
-0.01 |
0.03 |
0.04 |
0.06 |
0.05 |
0.05 |
0.05 |
0.05 |
0.03 |
0.02 |
-0.01 |
0.00 |
-0.01 |
-0.04 |
-0.04 |
-0.04 |
-0.04 |
-0.05 |
-0.05 |
| GM.5.0.T-box.0008 |
TBX5,TBX21,TBX4,TBX20,TBX1, (...) |
|
3.86 |
2.68 |
1.54 |
0.99 |
0.15 |
3.03 |
1.10 |
3.16 |
3.81 |
1.44 |
0.99 |
1.71 |
0.40 |
-1.65 |
-1.07 |
0.22 |
0.18 |
-3.30 |
-3.57 |
-3.27 |
-3.63 |
-3.08 |
-3.48 |
-3.18 |
-3.82 |
2 |
0.03 |
0.02 |
0.01 |
0.01 |
0.00 |
0.02 |
0.01 |
0.03 |
0.03 |
0.02 |
0.02 |
0.03 |
0.03 |
0.01 |
-0.00 |
0.00 |
-0.01 |
-0.03 |
-0.02 |
-0.03 |
-0.03 |
-0.04 |
-0.04 |
-0.03 |
-0.03 |
| GM.5.0.C2H2_ZF.0013 |
SP8,SP3,SP6,SP5,SP2, (...) |
|
2.84 |
-2.04 |
-3.81 |
-0.27 |
-1.32 |
3.77 |
4.15 |
2.99 |
0.93 |
-0.83 |
-1.12 |
-2.10 |
-1.45 |
1.59 |
1.32 |
2.91 |
1.27 |
-0.46 |
0.05 |
2.26 |
3.38 |
-2.30 |
-3.33 |
-1.38 |
-1.93 |
2 |
0.02 |
-0.01 |
-0.00 |
0.02 |
0.00 |
0.04 |
0.04 |
0.06 |
0.04 |
0.03 |
0.02 |
0.02 |
0.02 |
0.02 |
0.01 |
0.01 |
-0.01 |
-0.01 |
-0.02 |
-0.03 |
-0.03 |
-0.05 |
-0.05 |
-0.05 |
-0.06 |
| GM.5.0.Homeodomain.0107 |
HOXA6,HOXB6,HOXC4,HOXA4,HOXD4, (...) |
|
3.27 |
-1.12 |
-3.04 |
-0.58 |
-2.41 |
0.44 |
1.84 |
2.03 |
3.32 |
4.40 |
2.37 |
3.01 |
3.24 |
3.73 |
1.76 |
1.53 |
-1.86 |
0.10 |
-0.96 |
-1.91 |
-2.07 |
-0.42 |
-2.22 |
-2.49 |
-3.18 |
3 |
0.02 |
-0.00 |
-0.00 |
0.01 |
-0.00 |
0.01 |
0.01 |
0.06 |
0.07 |
0.06 |
0.05 |
0.05 |
0.04 |
0.02 |
0.01 |
-0.00 |
-0.02 |
-0.02 |
-0.03 |
-0.04 |
-0.04 |
-0.04 |
-0.04 |
-0.06 |
-0.06 |
| GM.5.0.Homeodomain_POU.0013 |
HOXA6,HOXB4,HOXB6,HOXC4,HOXA4, (...) |
|
1.69 |
-1.88 |
-1.26 |
-1.72 |
-2.21 |
-2.26 |
-0.68 |
3.37 |
3.64 |
2.65 |
2.80 |
1.61 |
2.94 |
1.98 |
2.92 |
1.09 |
-1.23 |
-0.81 |
-2.55 |
-1.17 |
-2.29 |
1.09 |
-1.47 |
-1.49 |
-2.09 |
4 |
0.01 |
-0.01 |
-0.00 |
0.00 |
-0.01 |
0.01 |
0.01 |
0.05 |
0.06 |
0.05 |
0.04 |
0.04 |
0.04 |
0.02 |
0.01 |
0.00 |
-0.02 |
-0.02 |
-0.03 |
-0.03 |
-0.03 |
-0.03 |
-0.04 |
-0.05 |
-0.05 |
| GM.5.0.Nuclear_receptor.0100 |
HNF4B,HNF4G,HNF4A |
|
1.49 |
1.96 |
0.42 |
-0.40 |
-1.17 |
-0.95 |
-1.91 |
1.54 |
3.29 |
2.85 |
2.92 |
2.35 |
1.96 |
1.25 |
1.80 |
1.28 |
-1.46 |
0.75 |
-0.36 |
-1.57 |
-2.78 |
-3.56 |
-3.27 |
-2.50 |
-3.30 |
2 |
0.04 |
0.03 |
0.01 |
0.01 |
0.00 |
0.01 |
0.00 |
0.03 |
0.04 |
0.05 |
0.06 |
0.04 |
0.03 |
0.00 |
-0.01 |
-0.02 |
-0.03 |
-0.02 |
-0.02 |
-0.04 |
-0.06 |
-0.05 |
-0.05 |
-0.05 |
-0.04 |
| GM.5.0.Homeodomain_POU.0001 |
POU2F1,POU3F2,POU3F4,POU3F1,POU3F3, (...) |
|
5.41 |
5.72 |
3.49 |
4.69 |
3.15 |
5.28 |
5.72 |
5.87 |
4.52 |
4.00 |
3.57 |
3.55 |
3.66 |
-0.77 |
-0.34 |
-4.30 |
-5.17 |
-6.02 |
-5.87 |
-6.17 |
-5.88 |
-6.02 |
-5.46 |
-6.17 |
-6.02 |
<1 |
0.18 |
0.13 |
0.06 |
0.08 |
0.05 |
0.08 |
0.08 |
0.10 |
0.09 |
0.06 |
0.05 |
0.03 |
0.03 |
-0.02 |
-0.03 |
-0.06 |
-0.06 |
-0.13 |
-0.13 |
-0.16 |
-0.14 |
-0.13 |
-0.10 |
-0.13 |
-0.10 |
| GM.5.0.GATA.0005 |
GATA1,GATA3,GATA2,GATA6,GATA4, (...) |
|
-5.49 |
-5.31 |
-5.41 |
-5.48 |
-5.60 |
-5.51 |
-4.39 |
-1.82 |
2.16 |
5.56 |
6.17 |
5.88 |
5.20 |
5.54 |
5.80 |
3.89 |
3.26 |
4.14 |
3.29 |
4.25 |
3.68 |
2.53 |
-1.00 |
0.57 |
-3.50 |
5 |
-0.14 |
-0.12 |
-0.08 |
-0.09 |
-0.07 |
-0.07 |
-0.06 |
0.01 |
0.02 |
0.10 |
0.10 |
0.11 |
0.09 |
0.11 |
0.09 |
0.07 |
0.02 |
0.06 |
0.05 |
0.06 |
0.03 |
0.01 |
-0.01 |
-0.00 |
-0.03 |
| GM.5.0.Sox.0028 |
POU5F3.2,POU5F3.3,POU5F3.1,SOX3,SOX2, (...) |
|
5.41 |
5.20 |
2.66 |
4.52 |
2.39 |
5.79 |
6.02 |
5.87 |
5.27 |
3.09 |
1.67 |
3.23 |
2.91 |
-0.82 |
-1.75 |
-5.72 |
-4.92 |
-5.72 |
-5.87 |
-5.49 |
-5.72 |
-5.24 |
-4.29 |
-5.36 |
-5.51 |
3 |
0.14 |
0.09 |
0.02 |
0.05 |
0.02 |
0.07 |
0.07 |
0.09 |
0.09 |
0.07 |
0.05 |
0.05 |
0.04 |
0.00 |
-0.01 |
-0.06 |
-0.06 |
-0.09 |
-0.10 |
-0.12 |
-0.12 |
-0.10 |
-0.10 |
-0.11 |
-0.10 |
| GM.5.0.C2H2_ZF.0024 |
CTCF,SMC3,ESRRG,ESRRA,RXRB, (...) |
|
-4.75 |
-5.17 |
-2.38 |
-2.93 |
-2.65 |
-1.57 |
-4.32 |
2.46 |
3.26 |
3.32 |
3.11 |
4.32 |
4.40 |
5.57 |
5.28 |
4.96 |
1.48 |
4.70 |
1.88 |
1.96 |
1.73 |
0.91 |
-0.14 |
2.25 |
0.42 |
<1 |
-0.06 |
-0.05 |
-0.02 |
-0.02 |
-0.02 |
-0.00 |
-0.01 |
0.02 |
0.03 |
0.04 |
0.03 |
0.05 |
0.04 |
0.05 |
0.05 |
0.03 |
0.00 |
0.02 |
0.02 |
-0.00 |
-0.01 |
-0.02 |
-0.02 |
-0.01 |
-0.02 |
| GM.5.0.Grainyhead.0002 |
GRHL3,GRHL1,GRHL2,TFCP2,UBP1, (...) |
|
-6.02 |
-6.17 |
-6.17 |
-5.87 |
-5.87 |
-5.14 |
-4.67 |
-5.36 |
-5.66 |
-6.17 |
-6.17 |
-6.17 |
-6.17 |
3.16 |
4.85 |
6.17 |
6.17 |
6.02 |
5.87 |
6.17 |
6.17 |
6.17 |
6.17 |
6.17 |
5.23 |
<1 |
-0.16 |
-0.14 |
-0.10 |
-0.07 |
-0.05 |
-0.04 |
-0.04 |
-0.08 |
-0.11 |
-0.15 |
-0.18 |
-0.10 |
-0.09 |
0.02 |
0.04 |
0.13 |
0.14 |
0.11 |
0.09 |
0.21 |
0.23 |
0.16 |
0.14 |
0.13 |
0.05 |