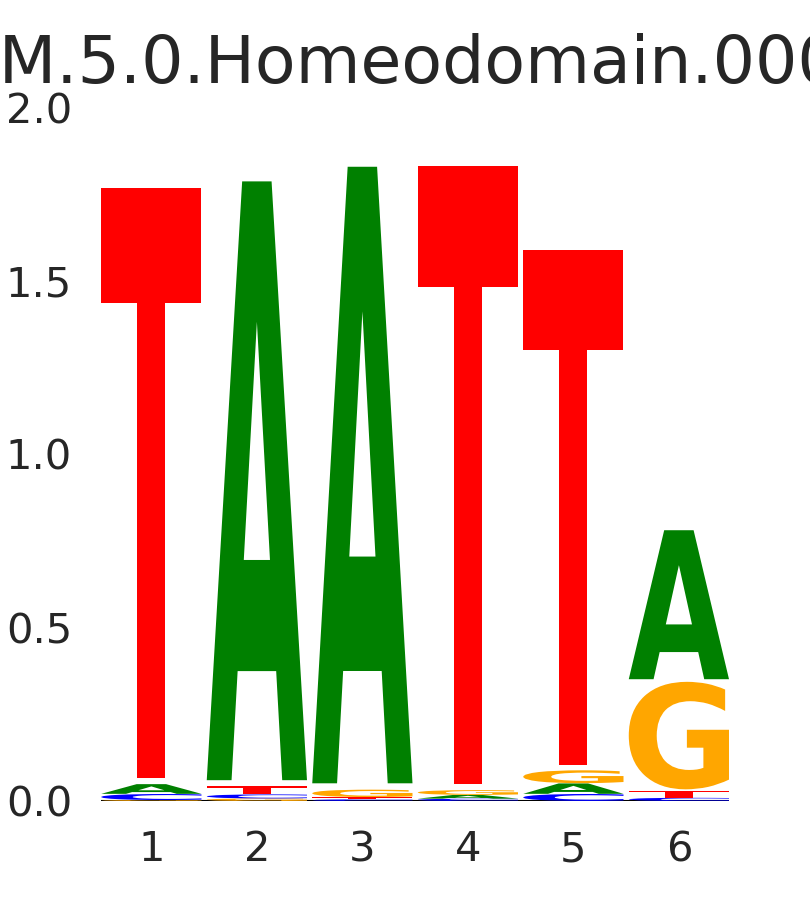
| GM.5.0.Homeodomain.0002 |
ISX,PRRXL1,DLX5,PRRX2,DLX2, (...) |
|
3.50 |
-1.49 |
-3.02 |
<1 |
0.09 |
-0.08 |
-0.04 |
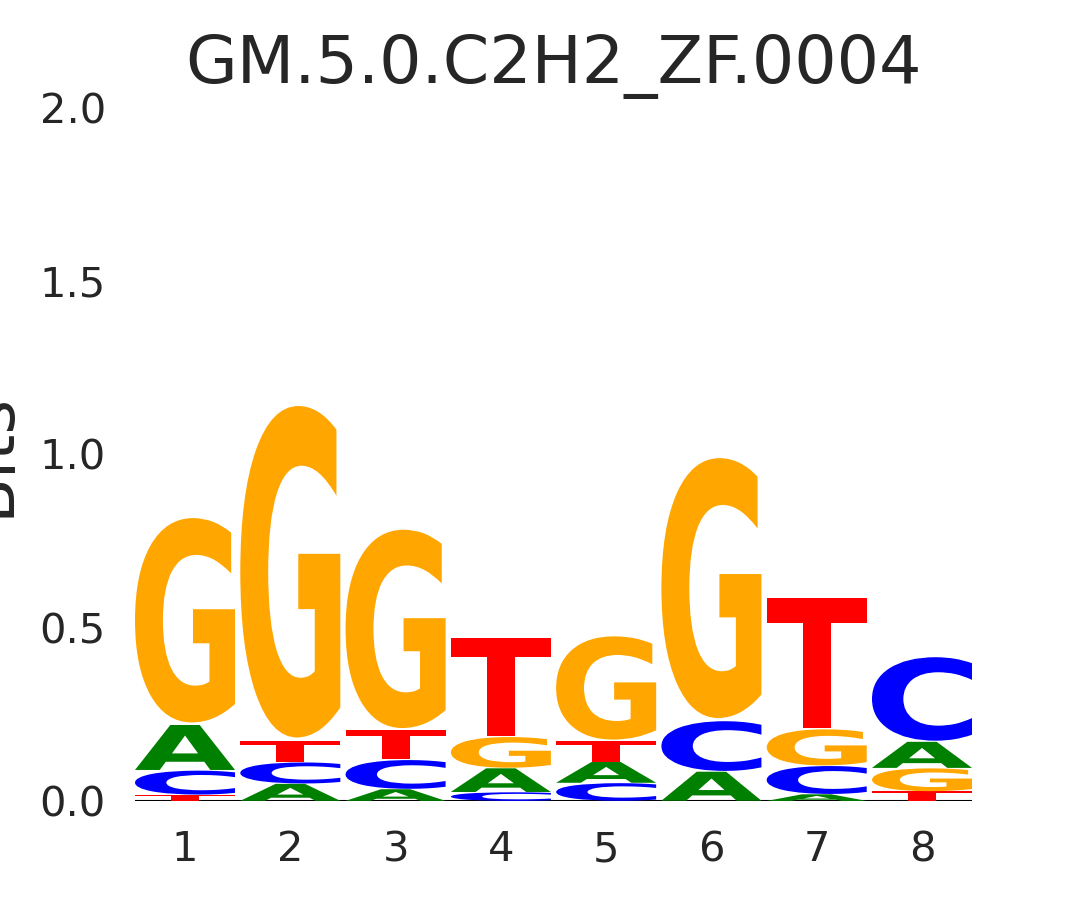
| GM.5.0.C2H2_ZF.0004 |
ZIC1,ZIC5,ZIC4 |
|
-1.58 |
-3.06 |
2.16 |
<1 |
-0.07 |
-0.08 |
0.10 |
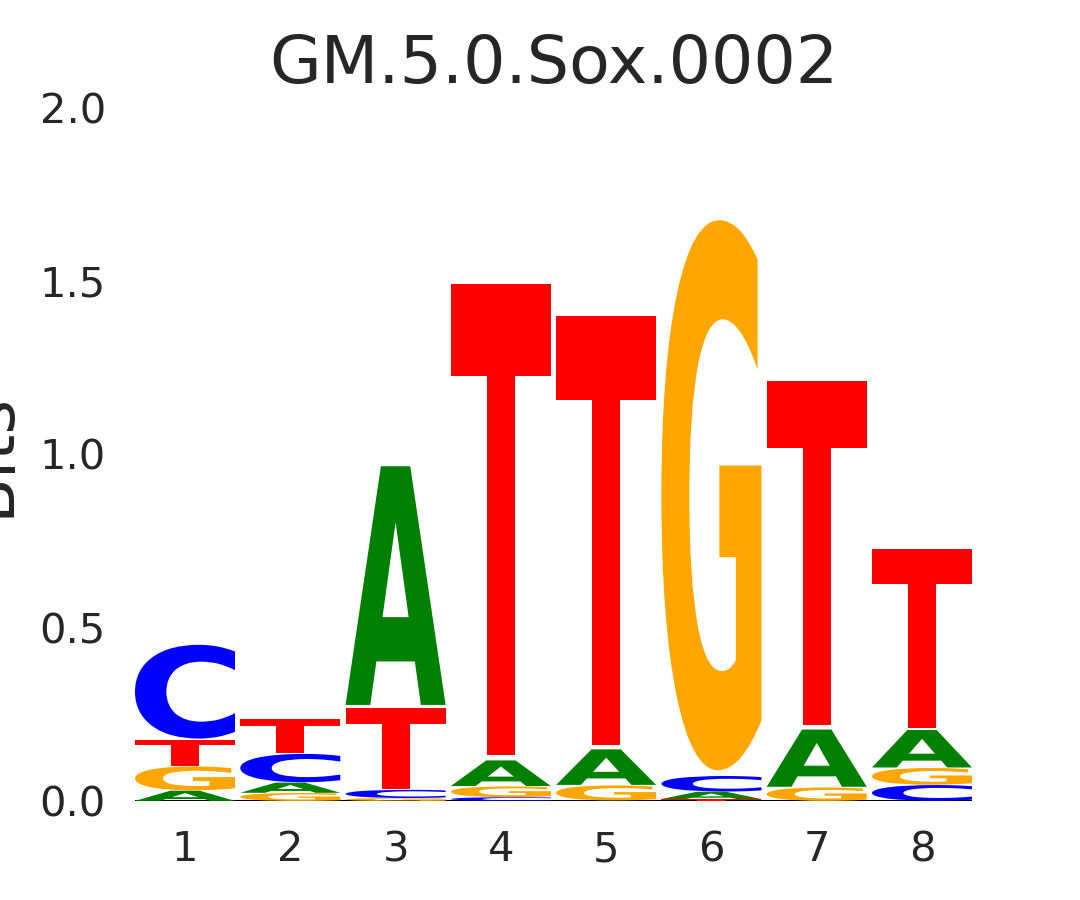
| GM.5.0.Sox.0002 |
SRY,SOX9 |
|
-3.27 |
-1.58 |
3.17 |
1 |
-0.01 |
-0.07 |
0.04 |
| GM.5.0.RFX.0001 |
ARID2,RFX8,RFX6,RFX5,RFX7, (...) |
|
-3.99 |
1.53 |
3.67 |
2 |
-0.02 |
-0.05 |
0.04 |
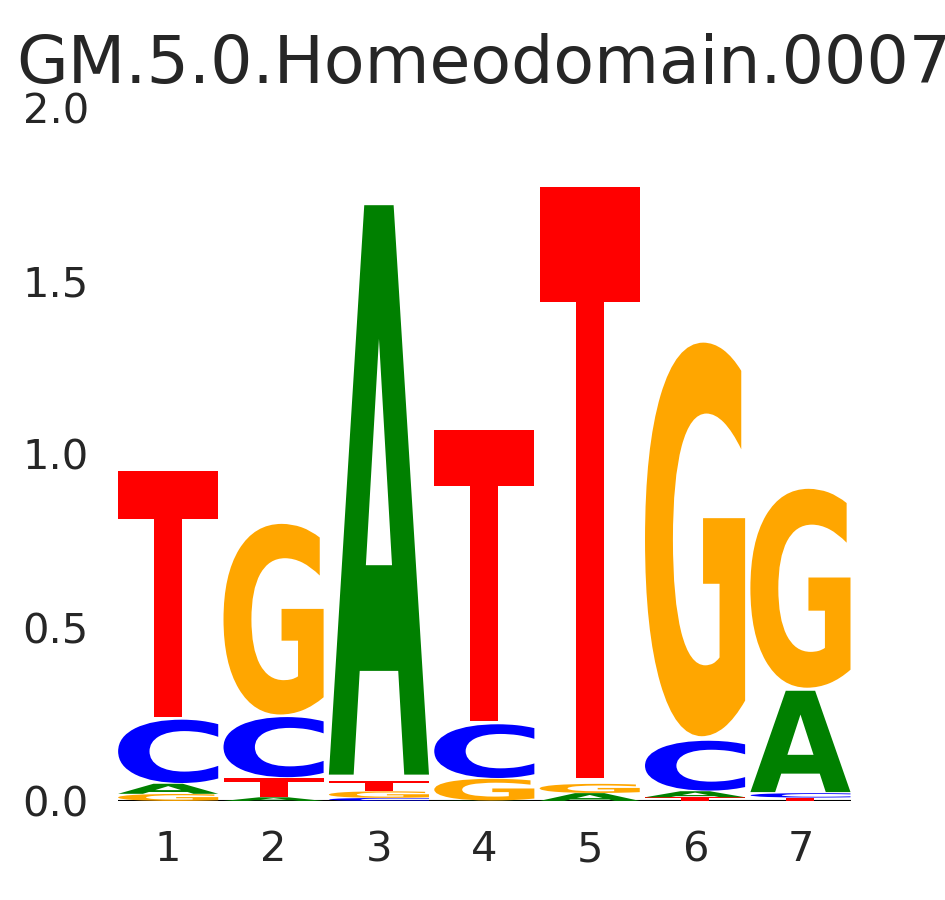
| GM.5.0.Homeodomain.0007 |
PBX3,FOS,AL662830.5,FOSB,PBX2, (...) |
|
-2.49 |
3.32 |
1.93 |
<1 |
-0.06 |
0.01 |
0.05 |
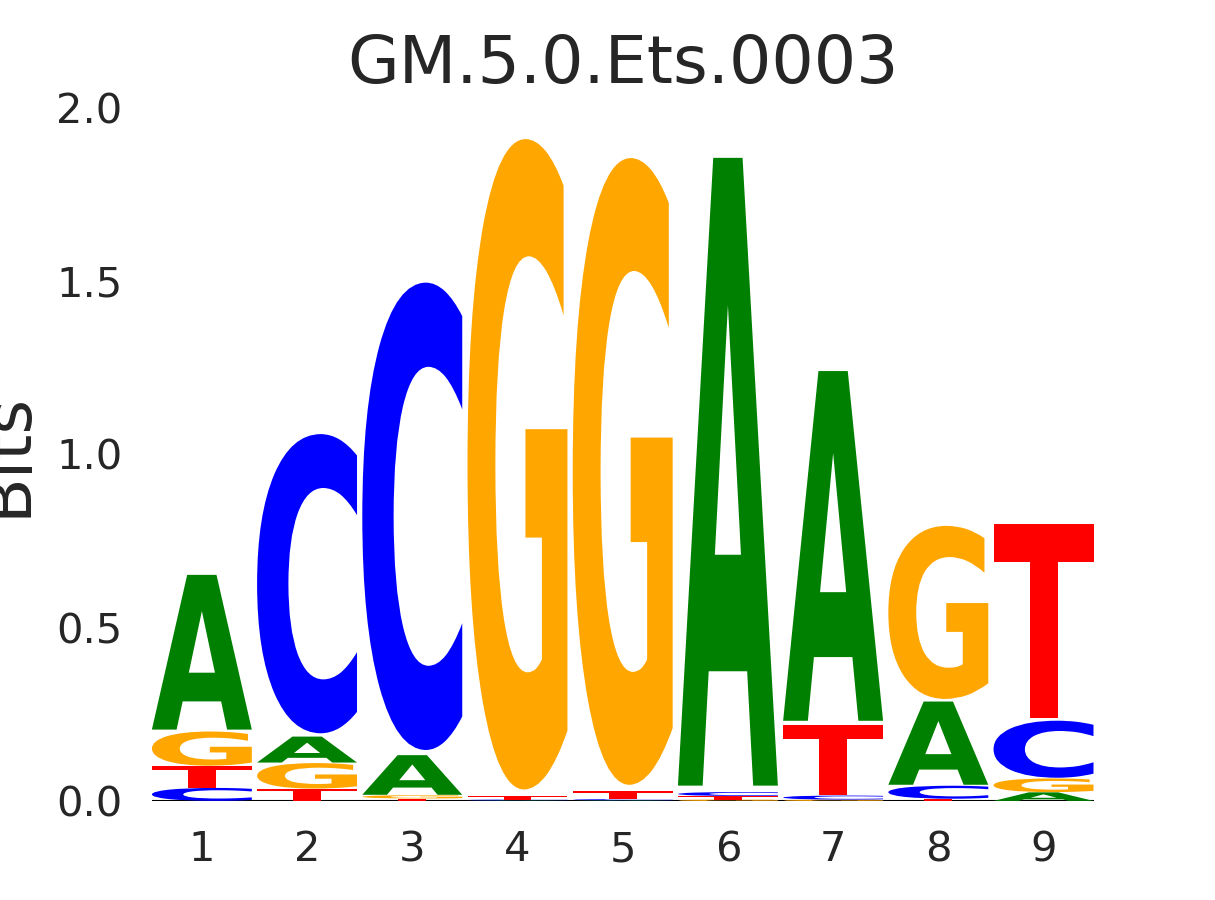
| GM.5.0.Ets.0003 |
ETV2,ERF,ELK3,ELK1,ERG, (...) |
|
-4.12 |
-0.78 |
3.92 |
2 |
-0.03 |
-0.06 |
0.05 |
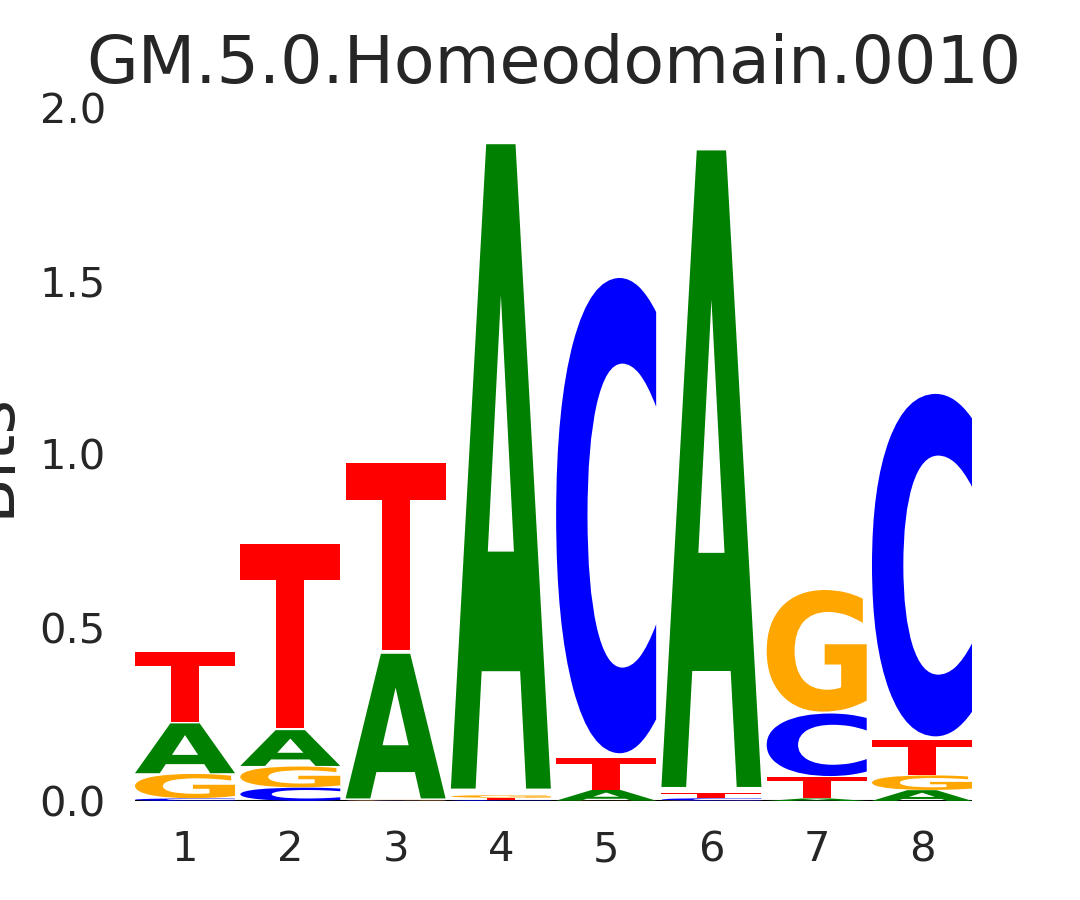
| GM.5.0.Homeodomain.0010 |
RHOX11,RHOX8,RHOXF1 |
|
-3.42 |
-2.10 |
3.53 |
2 |
-0.08 |
-0.10 |
0.12 |
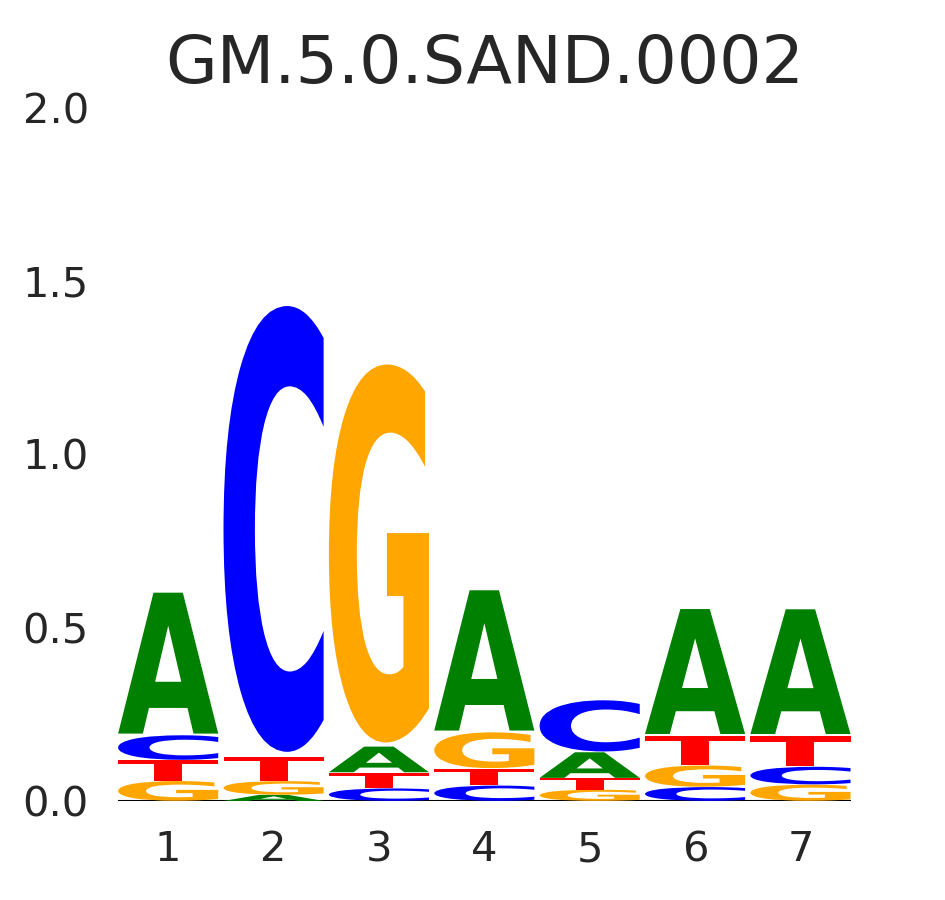
| GM.5.0.SAND.0002 |
IRC900814,ARHGEF12,SP140 |
|
3.00 |
-1.45 |
-3.15 |
2 |
0.13 |
-0.05 |
-0.09 |
| GM.5.0.C2H2_ZF.0009 |
KLF12,KLF3,KLF5,KLF7,KLF4, (...) |
|
-3.38 |
1.29 |
3.05 |
<1 |
-0.12 |
-0.06 |
0.13 |
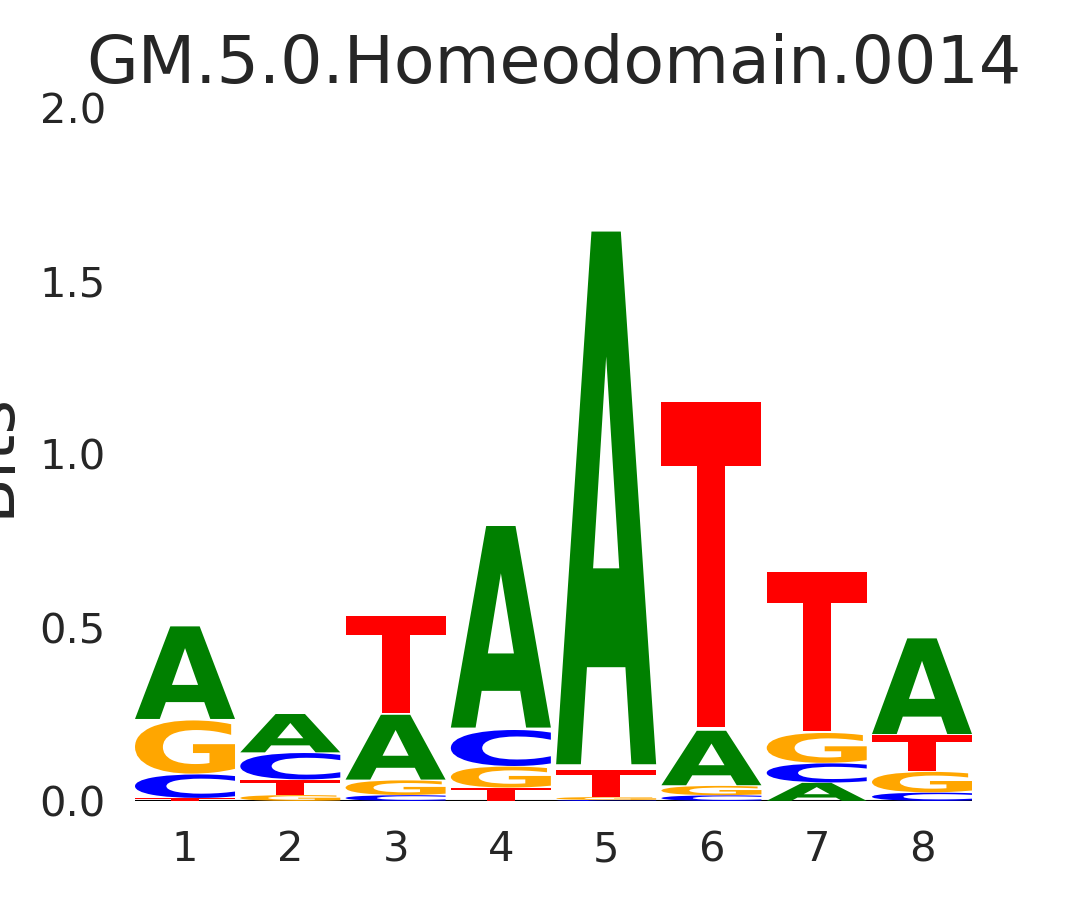
| GM.5.0.Homeodomain.0014 |
PAX4,HOXD8,HOXB9,HOXC5,HOXC9, (...) |
|
3.15 |
2.09 |
-3.62 |
2 |
0.12 |
-0.10 |
-0.06 |
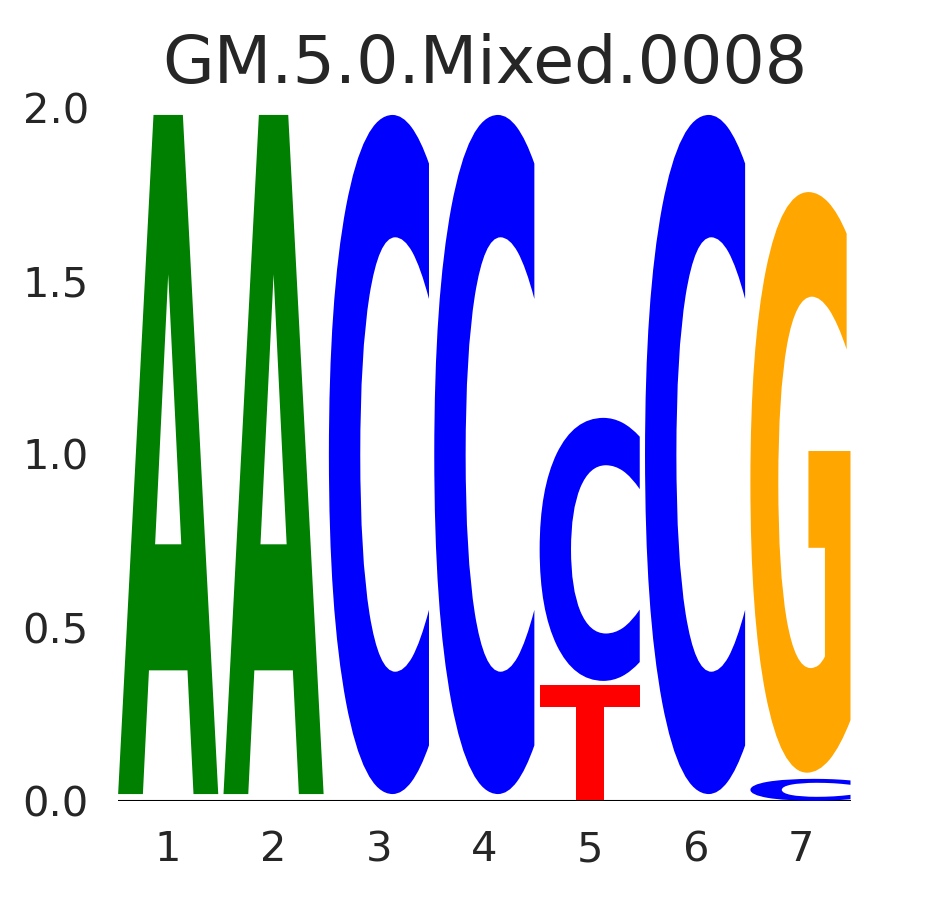
| GM.5.0.Mixed.0008 |
ZBTB33,NR2C2 |
|
3.57 |
-2.41 |
-3.22 |
<1 |
0.09 |
-0.07 |
-0.05 |
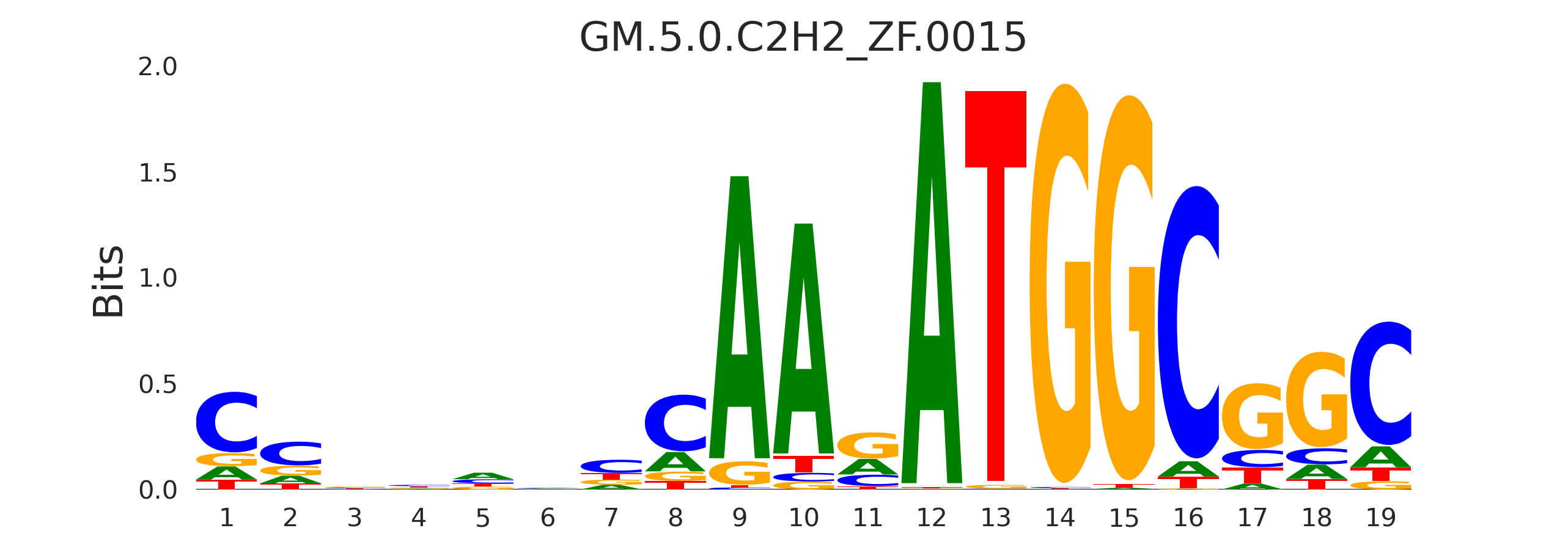
| GM.5.0.C2H2_ZF.0015 |
YY1,MBTPS2,ZFP42,TAF1,USF1, (...) |
|
-4.14 |
-2.30 |
4.37 |
1 |
-0.11 |
-0.10 |
0.15 |
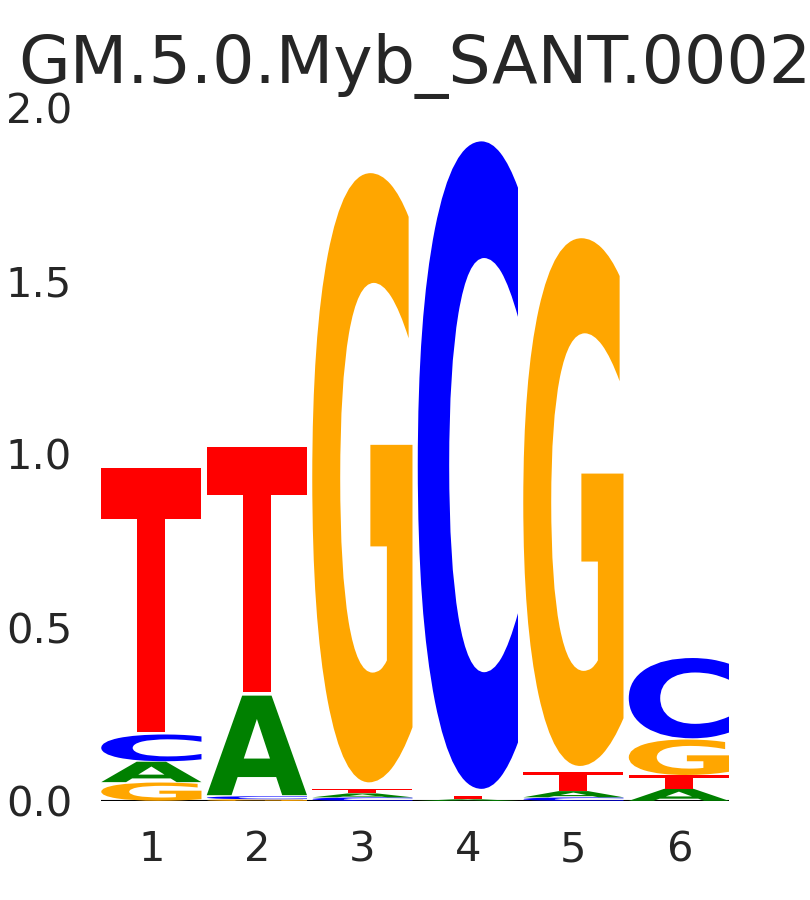
| GM.5.0.Myb_SANT.0002 |
MYPOP,PRDM11 |
|
5.02 |
-2.98 |
-4.64 |
<1 |
0.18 |
-0.10 |
-0.12 |
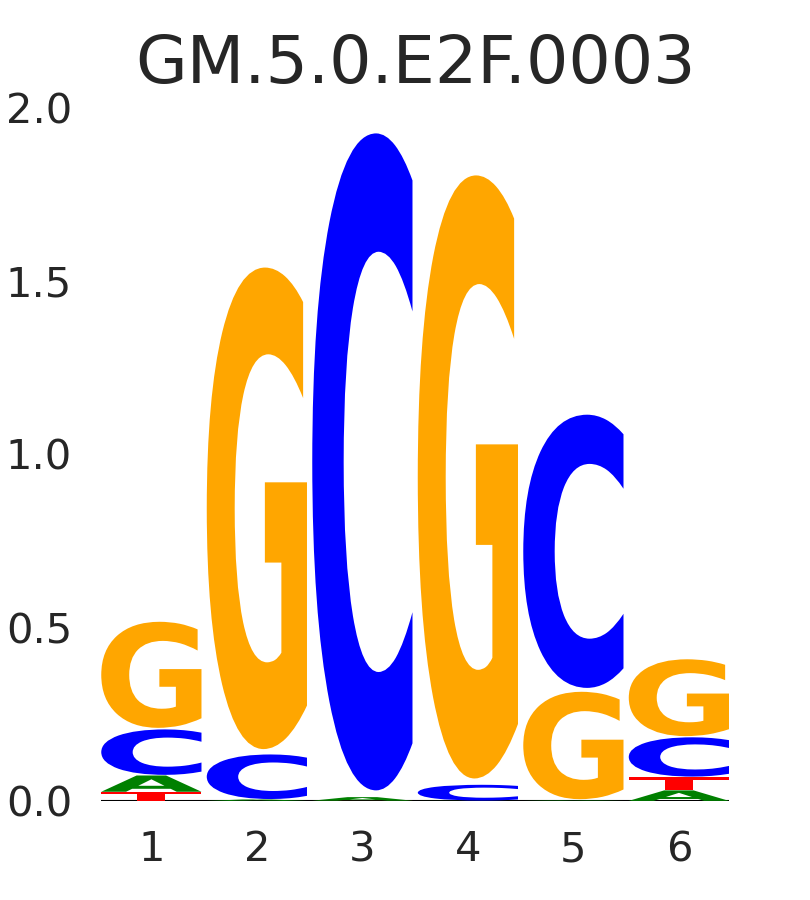
| GM.5.0.E2F.0003 |
E2F2,E2F4,E2F1,TFDP1 |
|
4.69 |
3.87 |
-5.14 |
<1 |
0.20 |
-0.11 |
-0.13 |
| GM.5.0.C2H2_ZF.0019 |
KLF7,KLF5,SP4,KLF8,KLF12, (...) |
|
3.49 |
2.01 |
-3.54 |
<1 |
0.00 |
-0.08 |
0.04 |
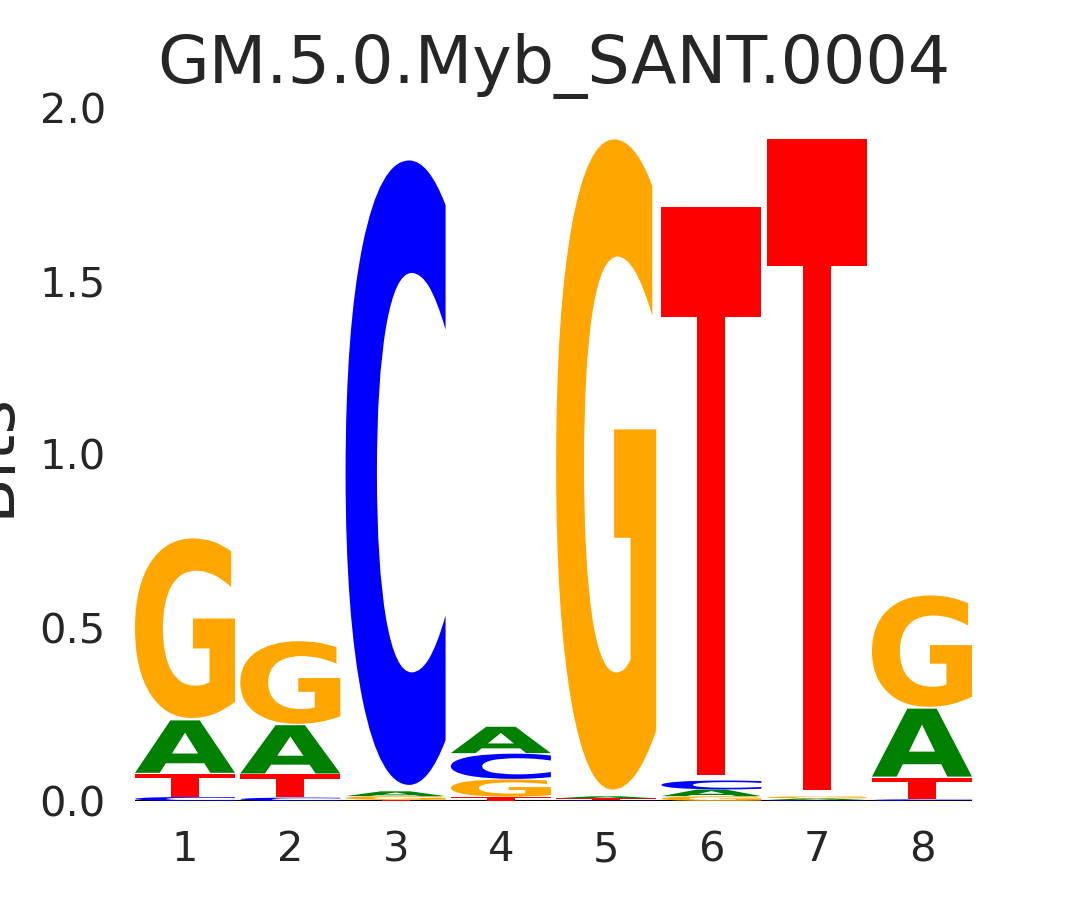
| GM.5.0.Myb_SANT.0004 |
MYB,AMYB,BMYB,MYBL1,MYBL2 |
|
-3.02 |
-2.21 |
3.12 |
<1 |
-0.04 |
-0.07 |
0.08 |
| GM.5.0.Mixed.0011 |
ETS2,STAT6 |
|
2.29 |
2.86 |
-3.02 |
<1 |
0.02 |
-0.06 |
0.01 |
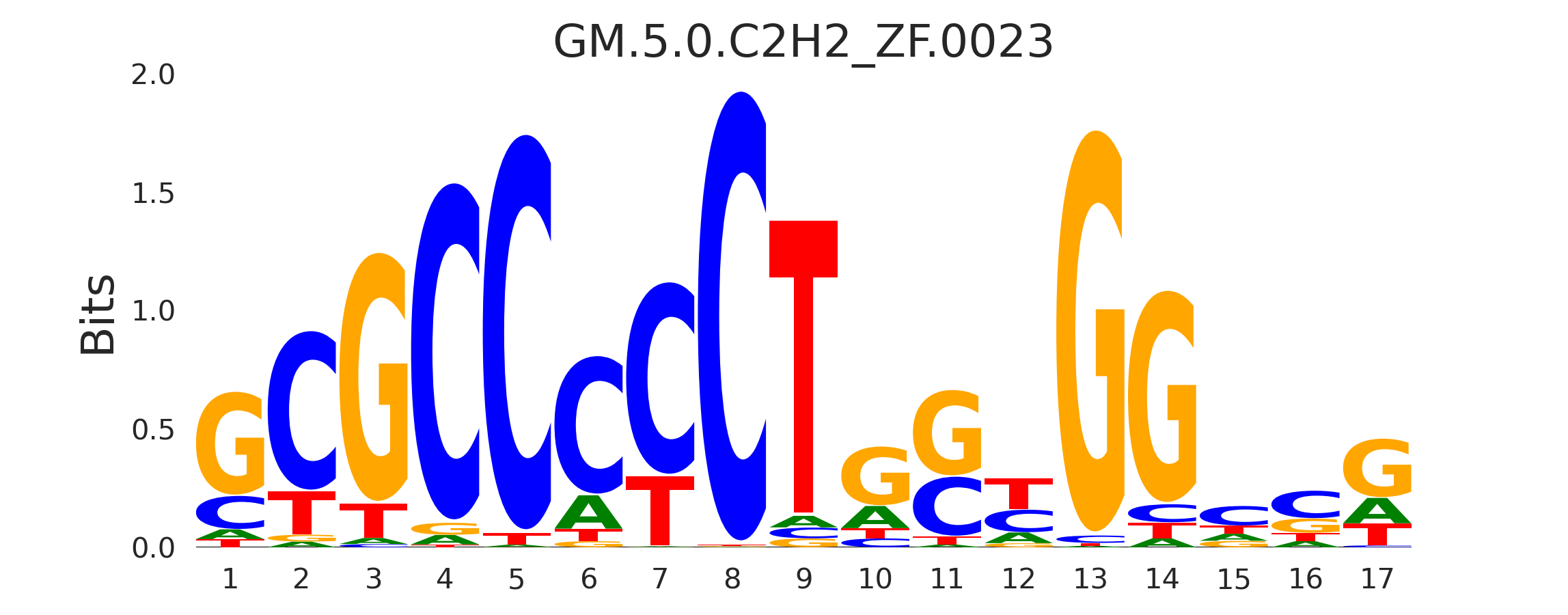
| GM.5.0.C2H2_ZF.0023 |
CTCFL,ZBTB7A,RXRA |
|
-4.87 |
5.01 |
4.09 |
2 |
-0.04 |
-0.01 |
0.04 |
| GM.5.0.SMAD.0003 |
NFIC,NFIB,NFIX,NFIA |
|
3.40 |
-3.85 |
-1.88 |
<1 |
0.12 |
-0.07 |
-0.08 |
| GM.5.0.C2H2_ZF.0024 |
CTCF,MYC,MAX,RAD21,SMC3, (...) |
|
-3.45 |
3.99 |
3.15 |
3 |
-0.04 |
0.03 |
0.02 |
| GM.5.0.C2H2_ZF.0027 |
MZF1,ZSCAN10 |
|
3.33 |
-0.43 |
-3.36 |
<1 |
-0.02 |
-0.09 |
0.06 |
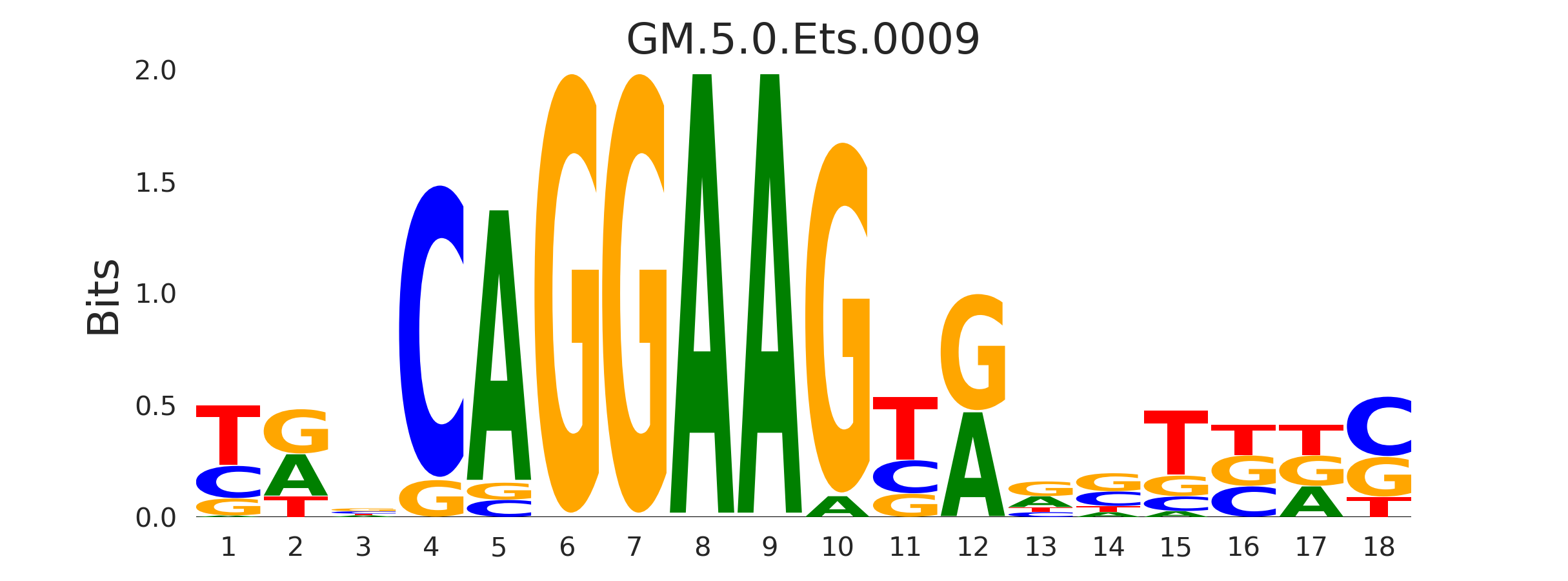
| GM.5.0.Ets.0009 |
ELF2,ELF1 |
|
-3.61 |
-1.40 |
3.45 |
<1 |
-0.12 |
-0.09 |
0.16 |
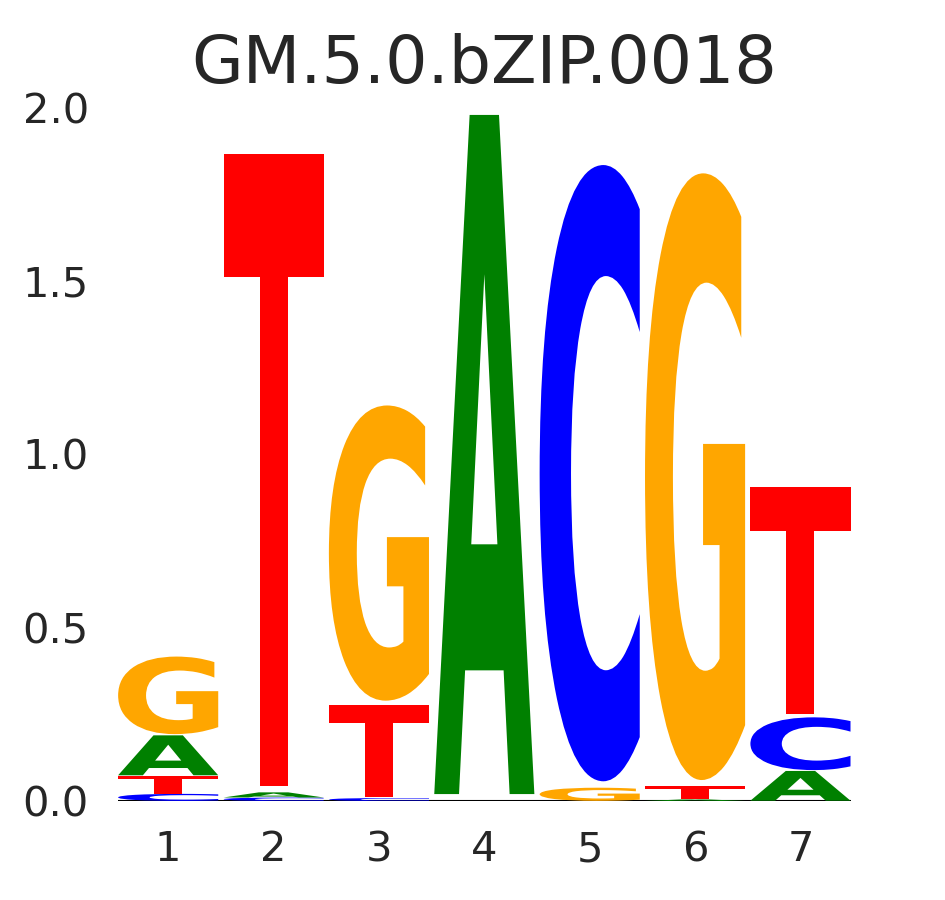
| GM.5.0.bZIP.0018 |
CREB1,E4F1,ATF1 |
|
-3.73 |
-1.90 |
3.86 |
<1 |
-0.00 |
-0.01 |
0.01 |
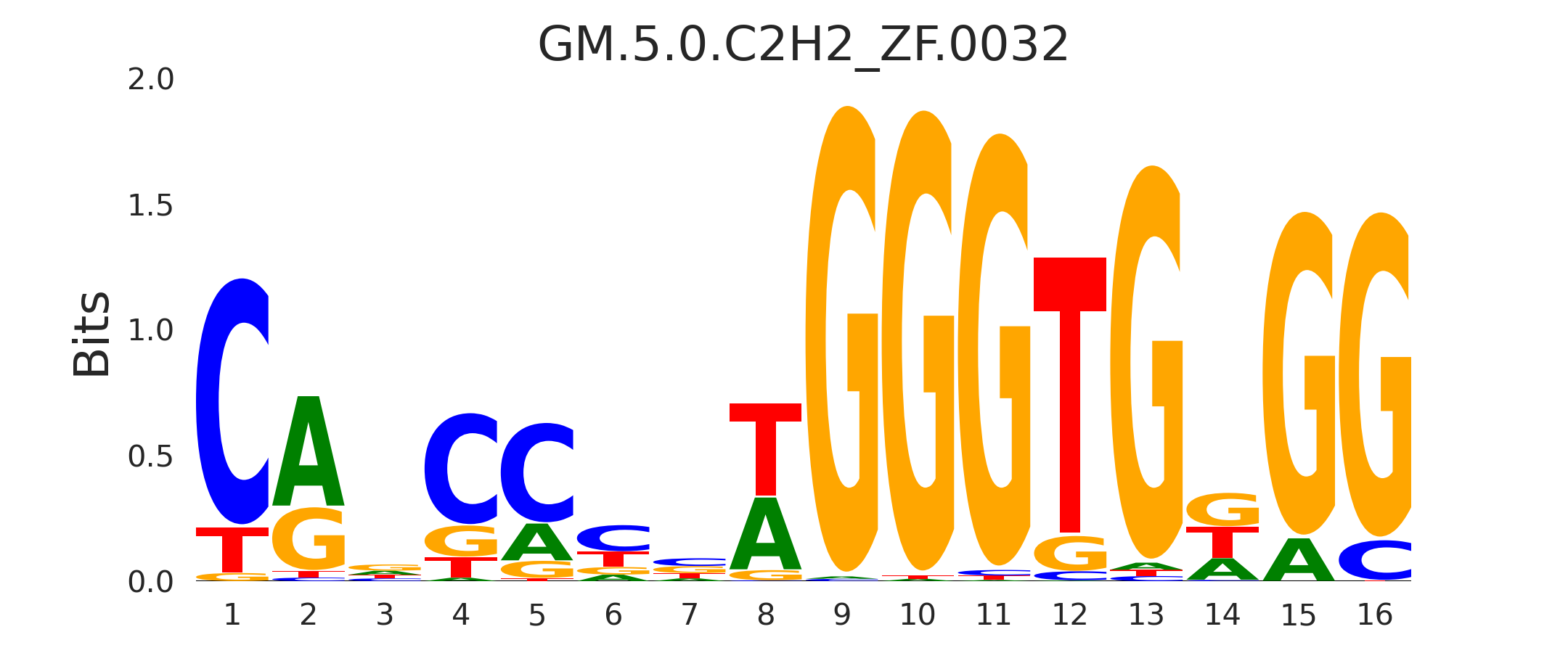
| GM.5.0.C2H2_ZF.0032 |
CACD,ZNF148 |
|
-0.98 |
-3.67 |
2.50 |
1 |
-0.13 |
-0.10 |
0.17 |
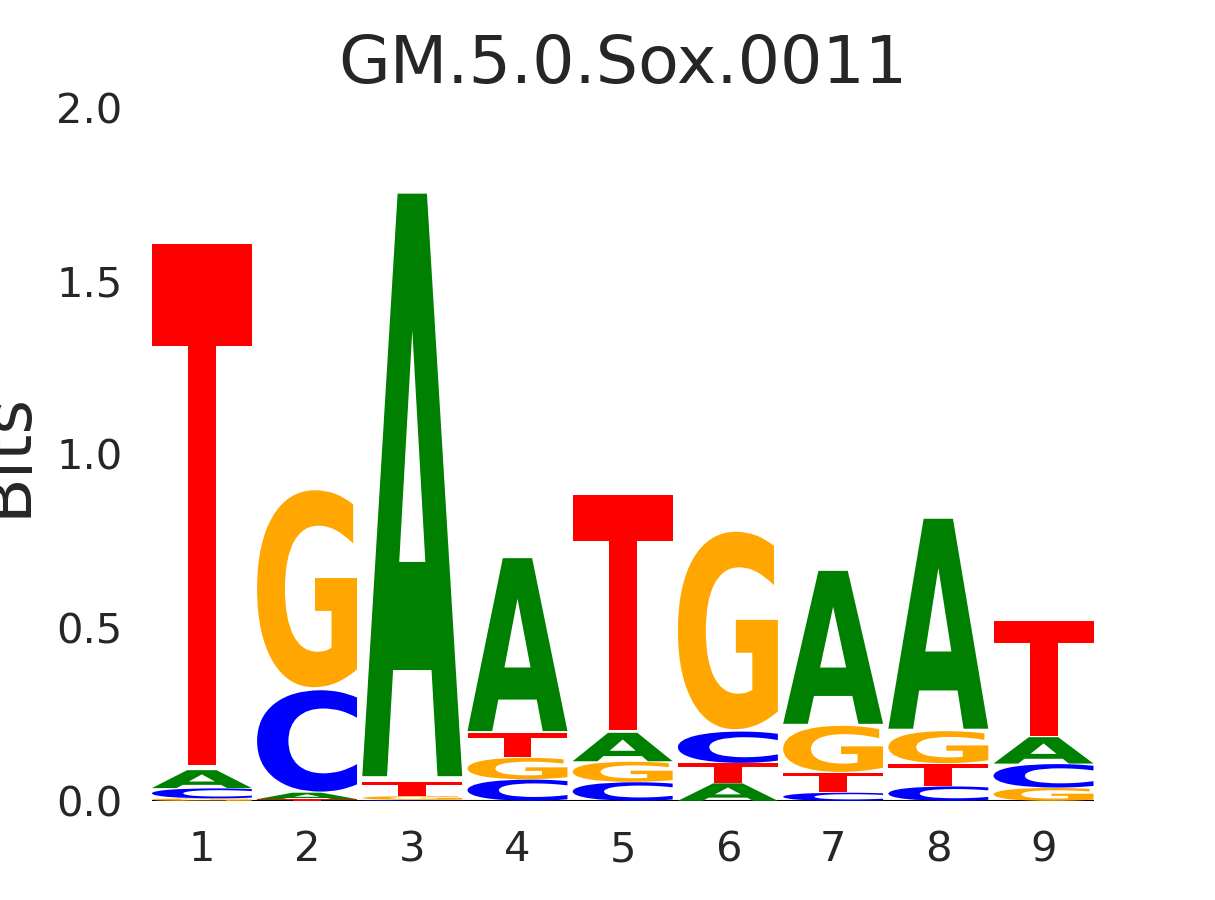
| GM.5.0.Sox.0011 |
HBP1 |
|
-0.42 |
3.10 |
-1.84 |
<1 |
0.00 |
0.04 |
-0.02 |
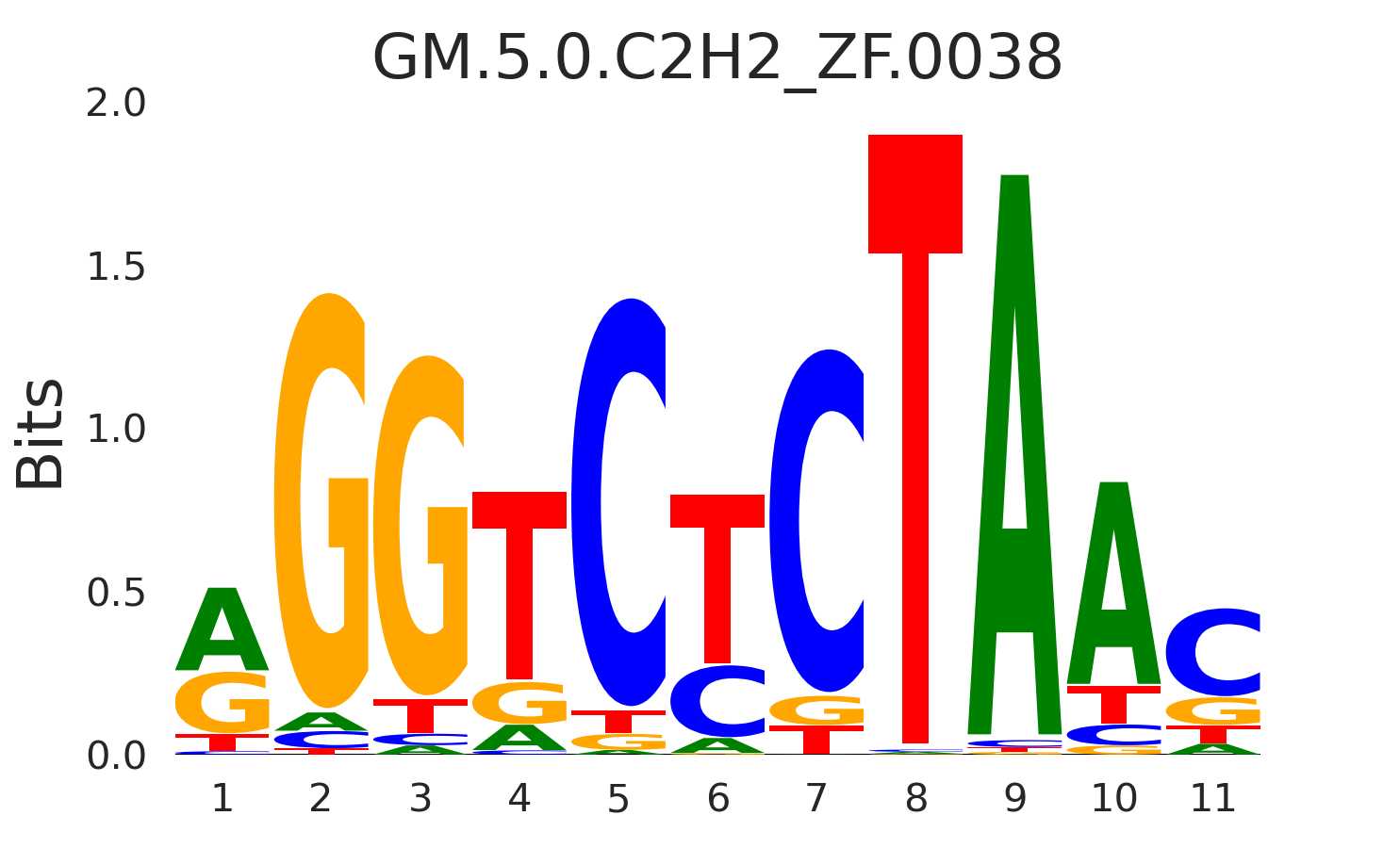
| GM.5.0.C2H2_ZF.0038 |
PRDM14,ZEB1 |
|
-1.61 |
-3.72 |
2.46 |
2 |
-0.01 |
-0.09 |
0.05 |
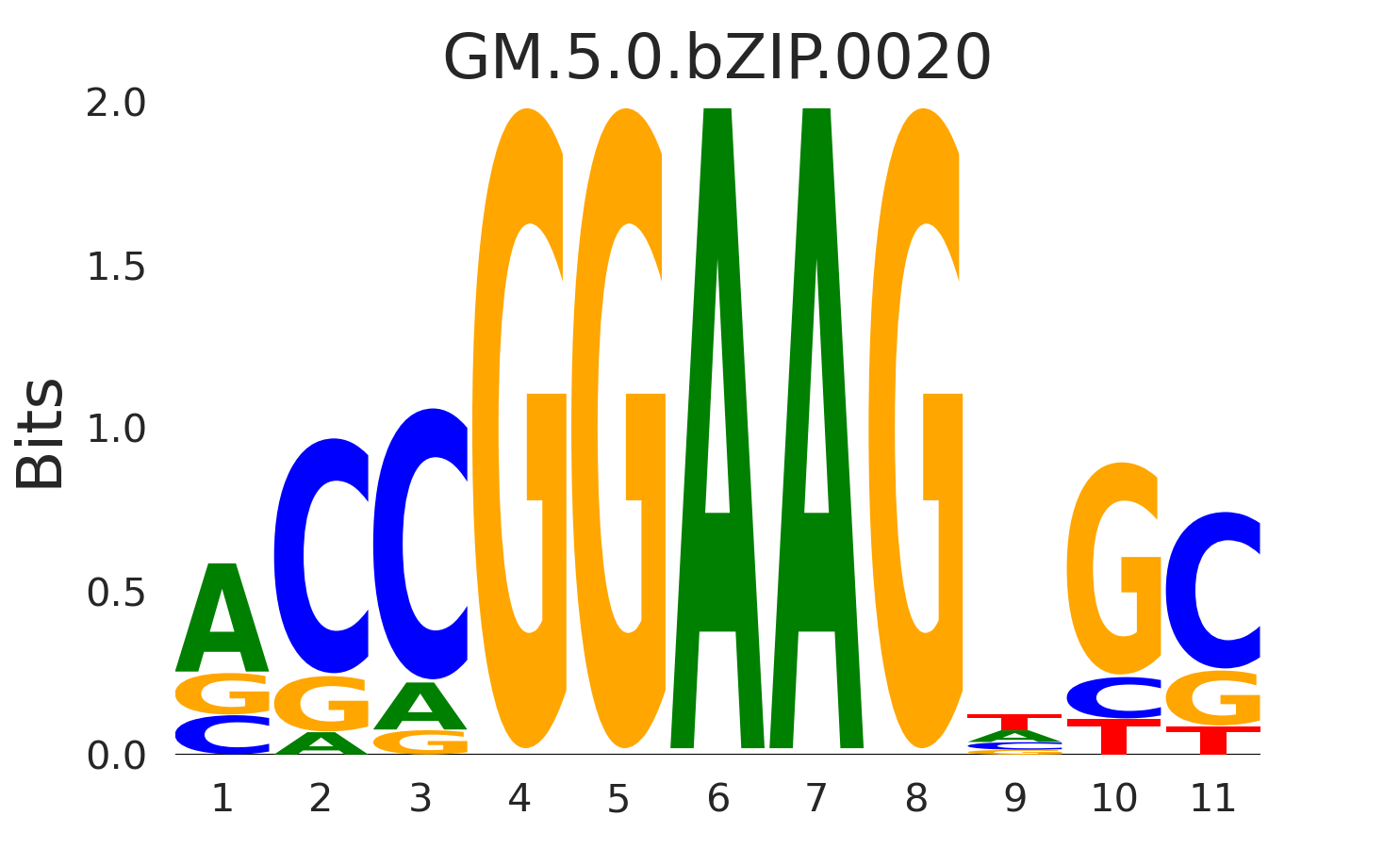
| GM.5.0.bZIP.0020 |
NFE2L2,GABP |
|
-3.07 |
-0.87 |
3.05 |
1 |
0.01 |
-0.06 |
0.02 |
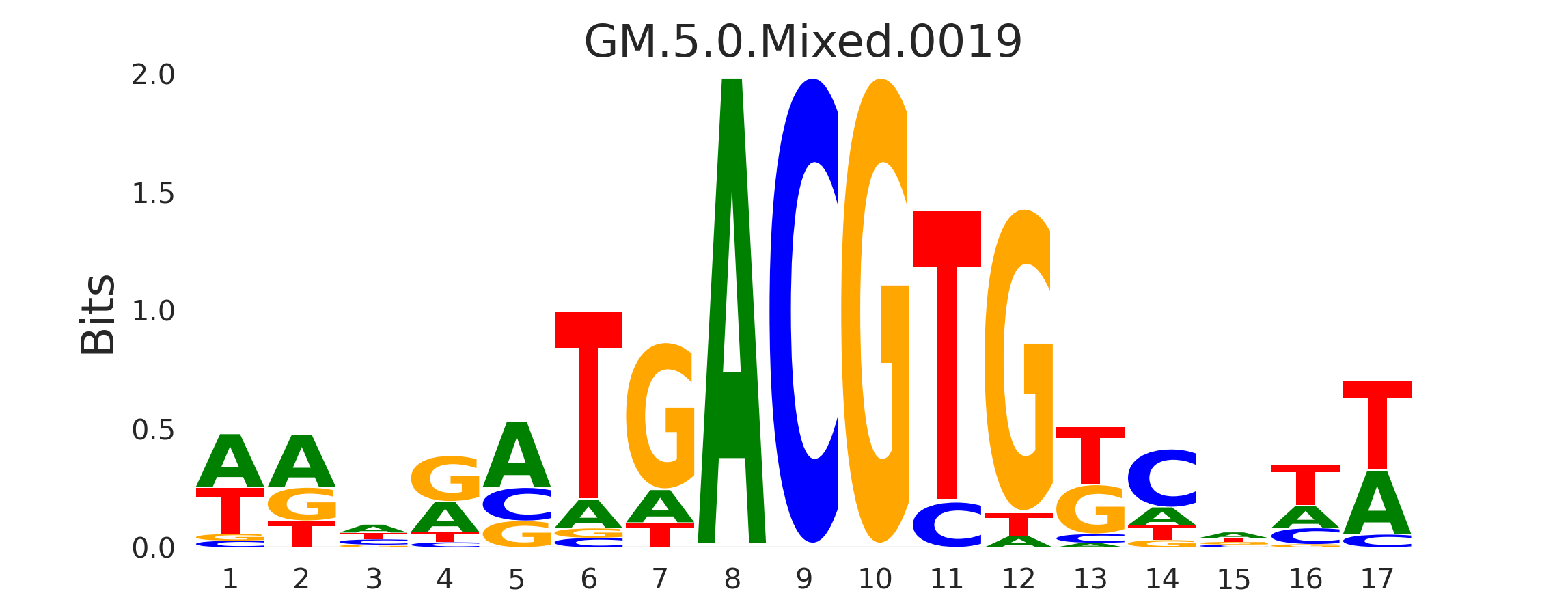
| GM.5.0.Mixed.0019 |
XBP1 |
|
3.09 |
1.07 |
-3.12 |
1 |
0.07 |
-0.02 |
-0.06 |
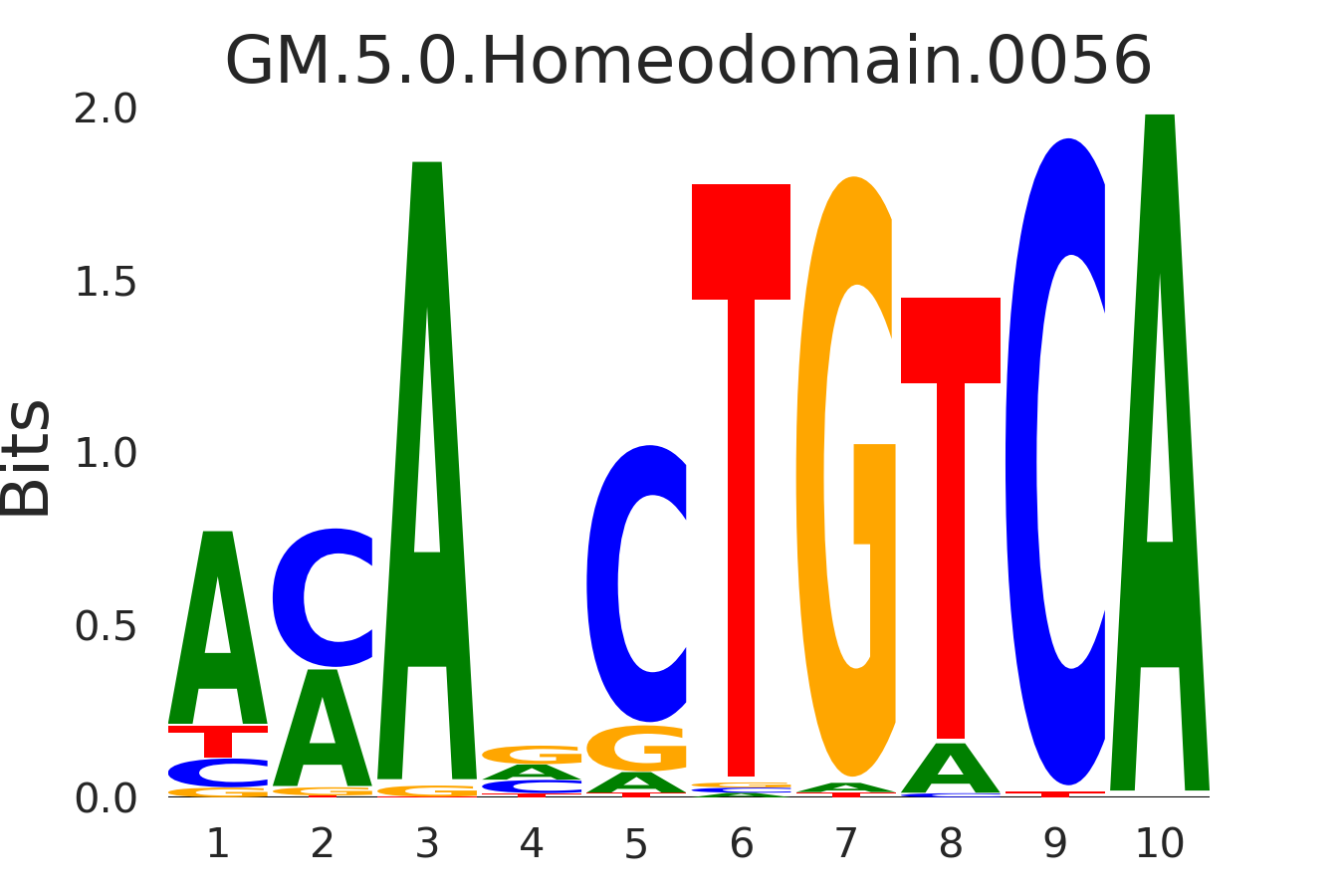
| GM.5.0.Homeodomain.0056 |
MEIS2,MEIS1 |
|
-1.44 |
3.75 |
0.67 |
1 |
-0.01 |
-0.00 |
0.01 |
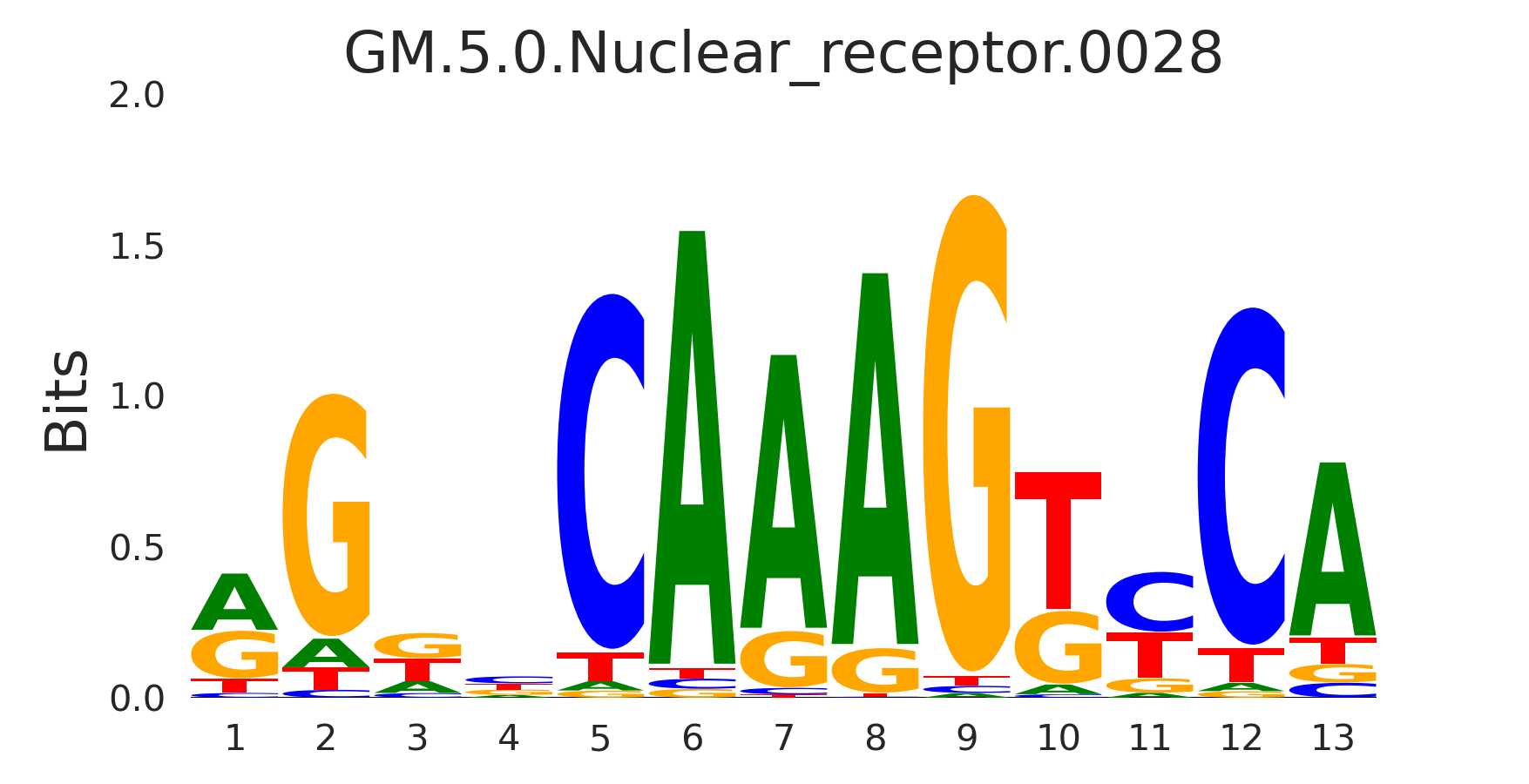
| GM.5.0.Nuclear_receptor.0028 |
HNF4A,HNF4G,NR1H3 |
|
3.04 |
0.45 |
-3.33 |
<1 |
-0.00 |
-0.06 |
0.03 |
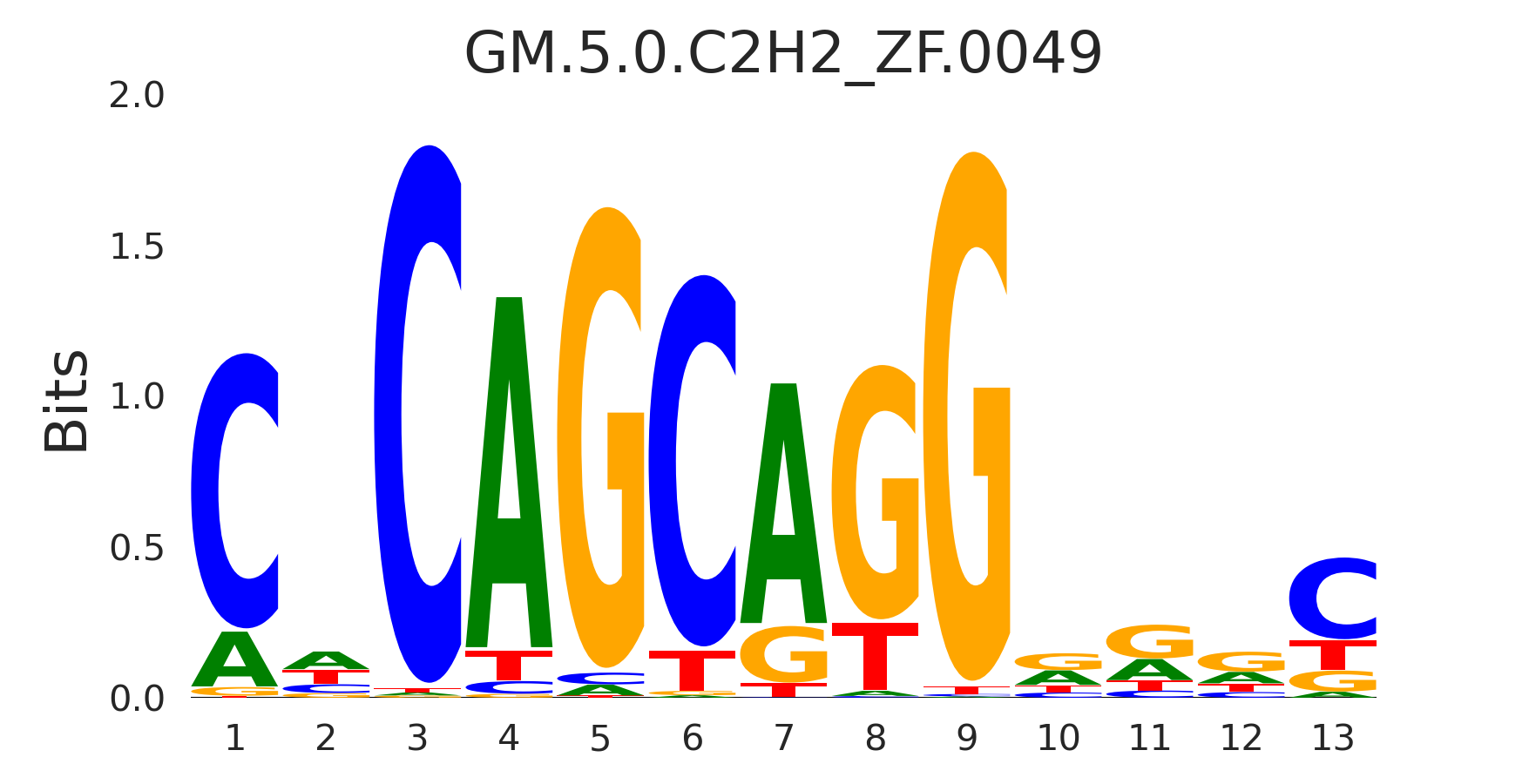
| GM.5.0.C2H2_ZF.0049 |
ZIC3,ZIC2,ZIC1 |
|
-3.25 |
2.03 |
3.32 |
<1 |
-0.14 |
-0.08 |
0.17 |
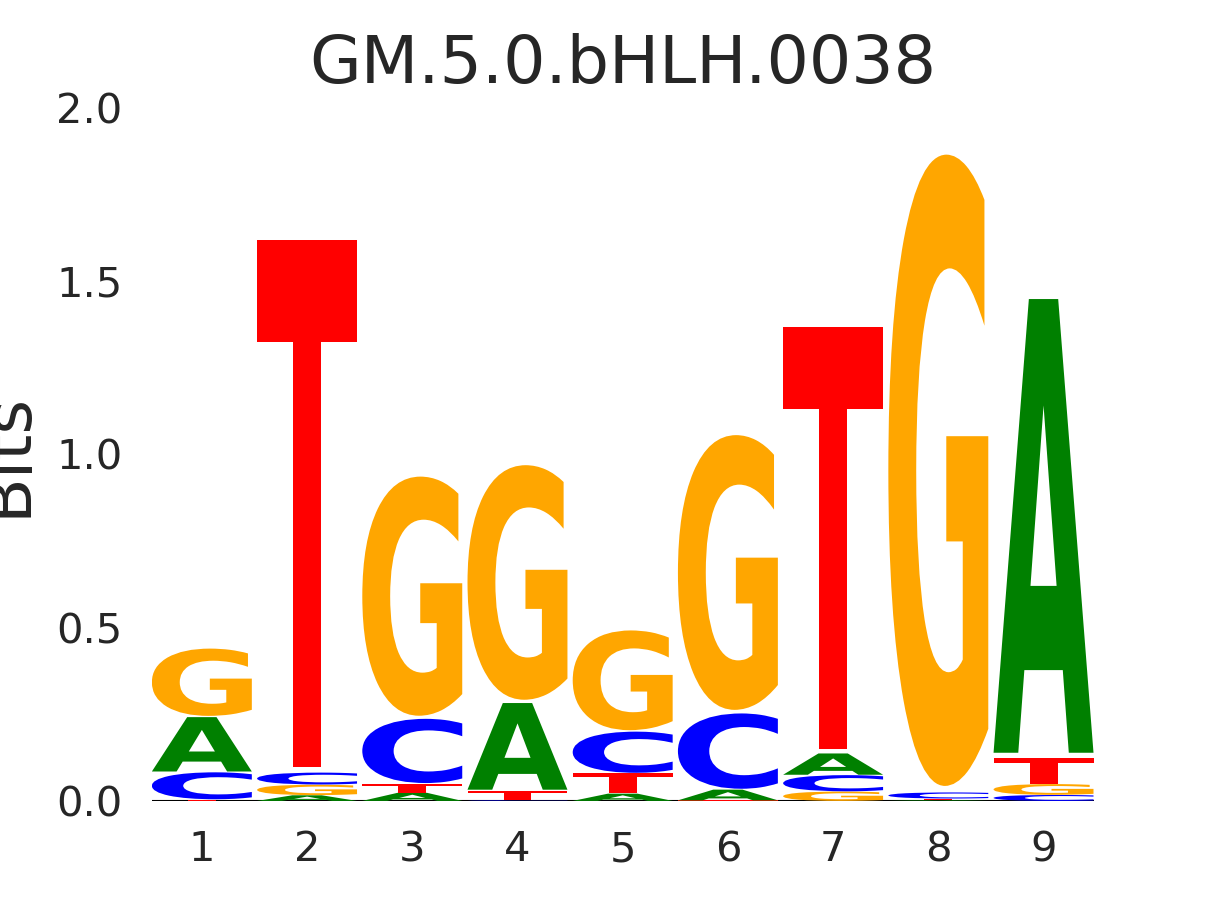
| GM.5.0.bHLH.0038 |
SREBF1,SREBF2 |
|
-2.09 |
3.86 |
-0.71 |
<1 |
-0.06 |
0.05 |
0.03 |
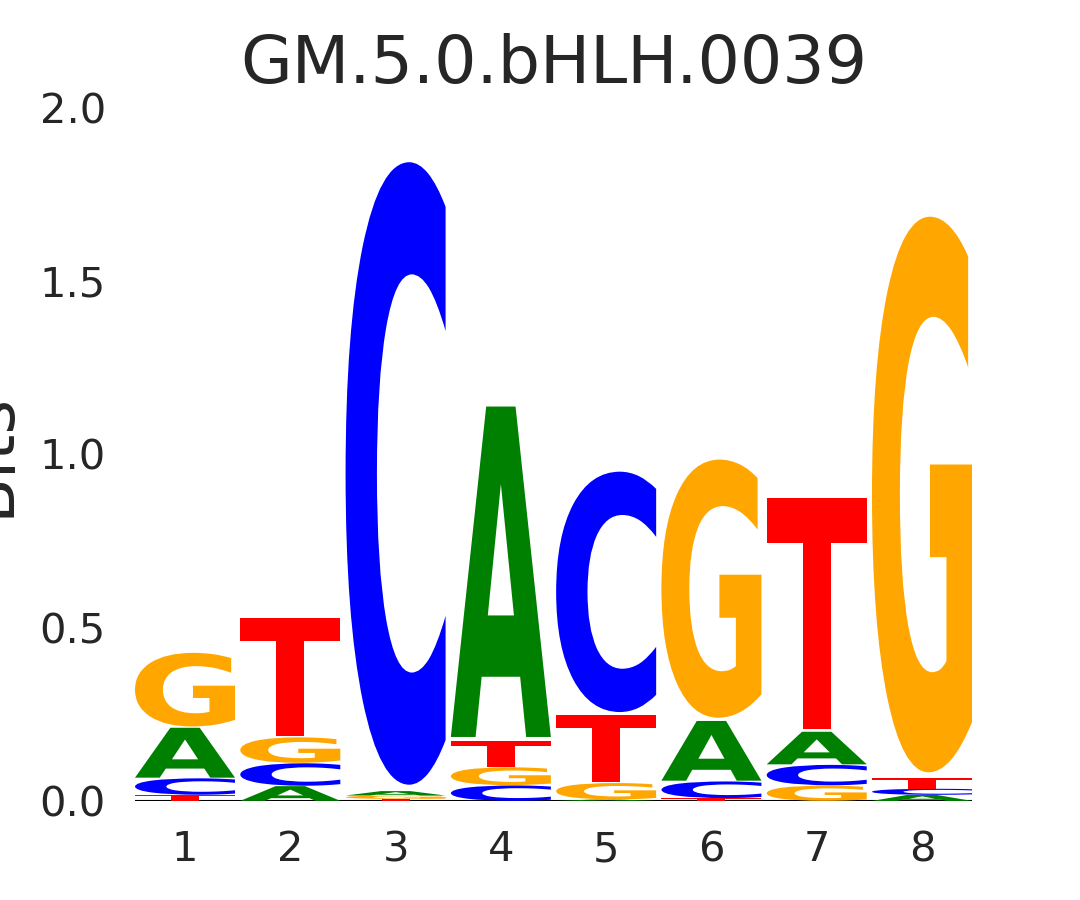
| GM.5.0.bHLH.0039 |
TFEC,TFE3,TFEB,MITF,TCFE3, (...) |
|
-3.44 |
-2.10 |
3.50 |
<1 |
-0.07 |
-0.05 |
0.09 |
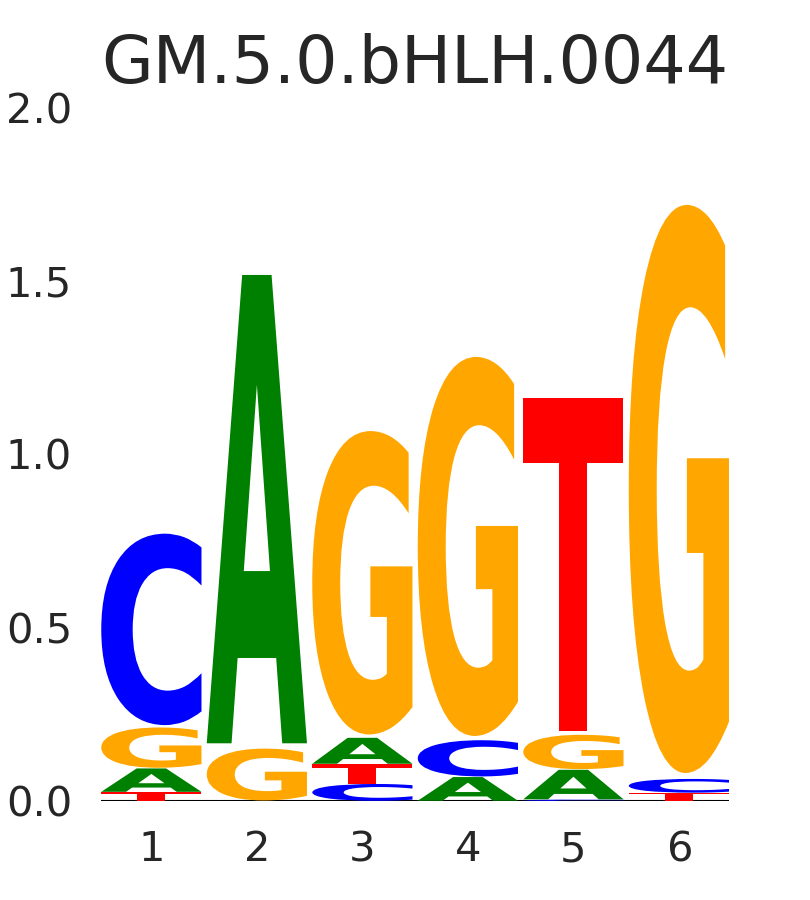
| GM.5.0.bHLH.0044 |
MESP1,TCF4,MESP2 |
|
-2.02 |
3.53 |
-0.34 |
<1 |
-0.00 |
0.02 |
-0.01 |
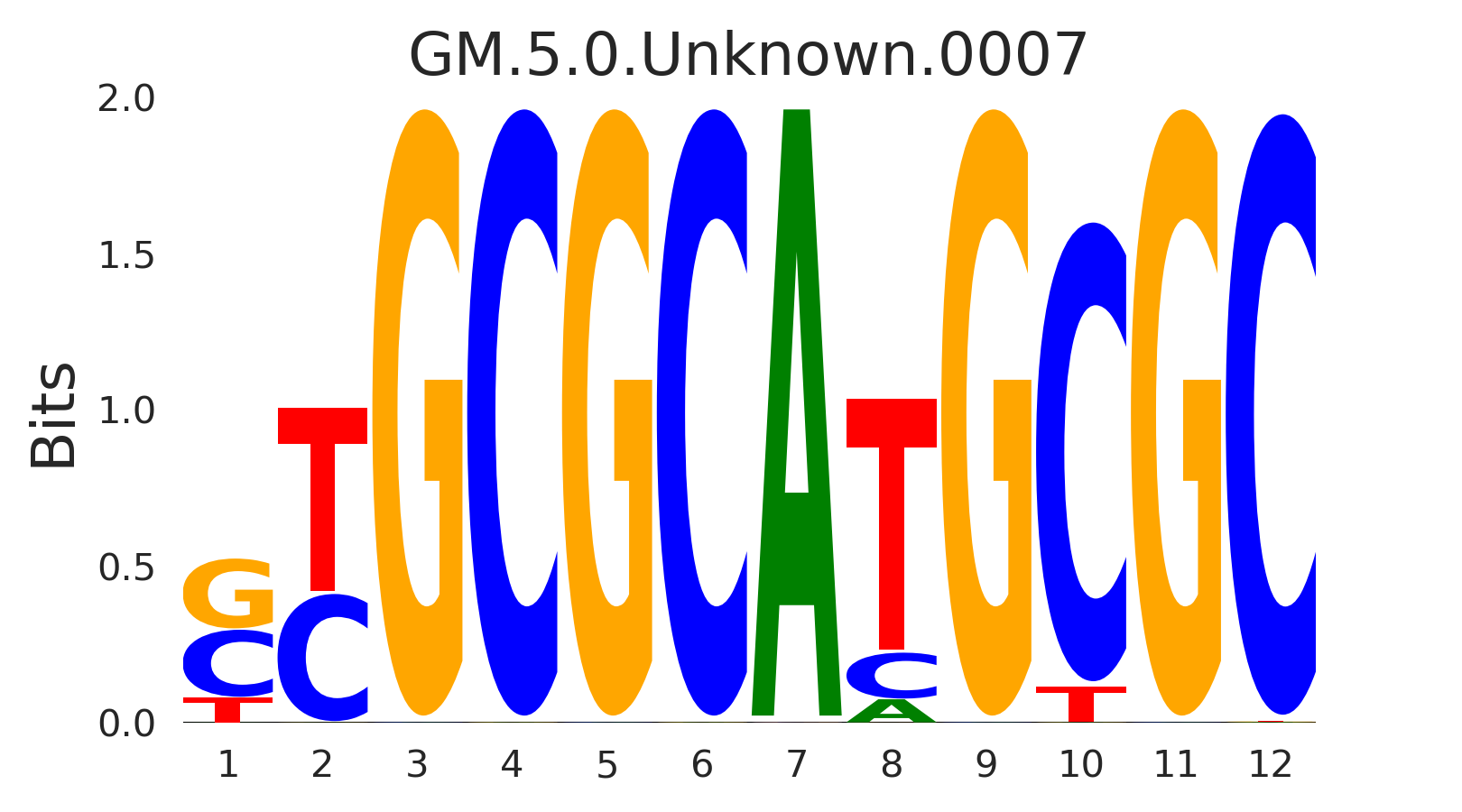
| GM.5.0.Unknown.0007 |
NRF1 |
|
-3.24 |
-1.53 |
3.44 |
1 |
0.12 |
-0.10 |
-0.06 |
| GM.5.0.C2H2_ZF.0058 |
GLI2,ZBTB7C,ZBTB7A,ZBTB7B,GLI1 |
|
2.42 |
3.93 |
-3.80 |
1 |
0.01 |
-0.02 |
-0.00 |
| GM.5.0.TEA.0001 |
TEAD4,TEAD3,TEAD1,TEAD2 |
|
-3.98 |
2.99 |
3.32 |
<1 |
-0.13 |
-0.02 |
0.13 |
| GM.5.0.Nuclear_receptor.0034 |
HNF4A,HNF4G |
|
2.93 |
2.58 |
-3.25 |
1 |
0.05 |
-0.03 |
-0.03 |
| GM.5.0.Nuclear_receptor.0036 |
RARB,NR2F6,RARG,RARA,NR2F1 |
|
-3.06 |
-3.69 |
3.69 |
<1 |
-0.03 |
-0.09 |
0.07 |
| GM.5.0.C2H2_ZF.0064 |
CTCFL,CTCF |
|
-3.18 |
-1.42 |
3.28 |
2 |
-0.05 |
-0.05 |
0.07 |
| GM.5.0.CBF_NF-Y.0002 |
NFYA,NFYB,NFYC,NFY,E2F4, (...) |
|
-4.50 |
-1.60 |
4.08 |
1 |
-0.06 |
-0.02 |
0.07 |
| GM.5.0.C2H2_ZF.0070 |
ZNF586 |
|
2.18 |
-3.77 |
-0.75 |
<1 |
0.05 |
-0.07 |
-0.01 |
| GM.5.0.Unknown.0011 |
ZBED1 |
|
3.44 |
-1.18 |
-2.88 |
1 |
0.06 |
-0.07 |
-0.02 |
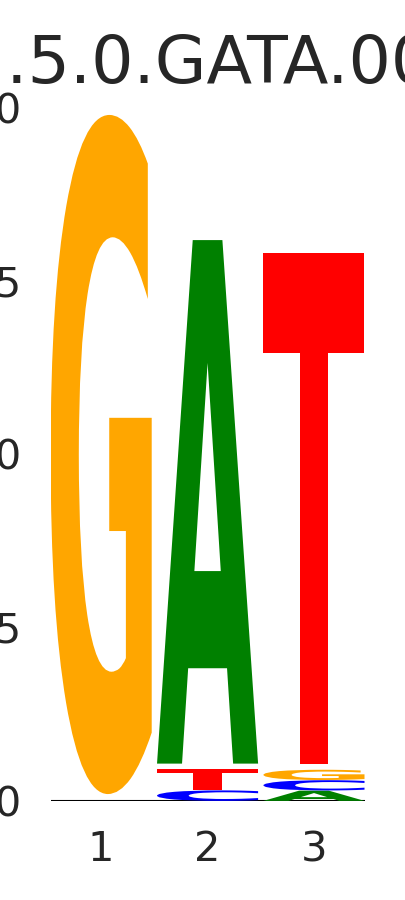
| GM.5.0.GATA.0010 |
GATA3 |
|
2.90 |
2.82 |
-3.73 |
1 |
0.05 |
-0.03 |
-0.03 |
| GM.5.0.HSF.0003 |
HSFY2,HSFY1 |
|
3.54 |
1.84 |
-3.89 |
2 |
0.12 |
0.02 |
-0.12 |
| GM.5.0.C2H2_ZF.0075 |
ZNF354C,ZFP354C |
|
2.67 |
3.87 |
-3.39 |
<1 |
0.07 |
0.03 |
-0.08 |
| GM.5.0.CUT_Homeodomain.0002 |
ONECUT1,ONECUT2,ONECUT3 |
|
3.93 |
-1.26 |
-3.21 |
<1 |
0.07 |
-0.07 |
-0.03 |
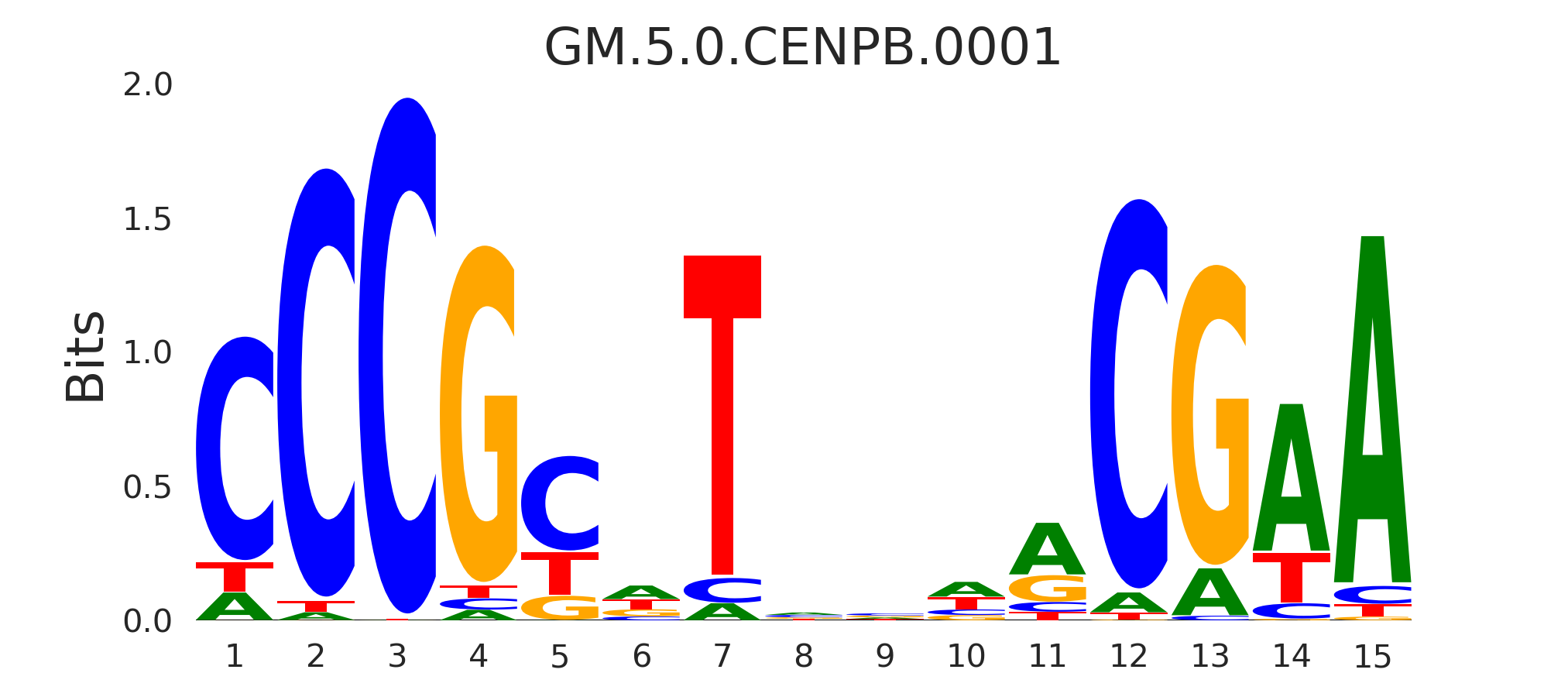
| GM.5.0.CENPB.0001 |
CENPB |
|
4.77 |
-3.73 |
-4.36 |
2 |
0.13 |
-0.08 |
-0.08 |
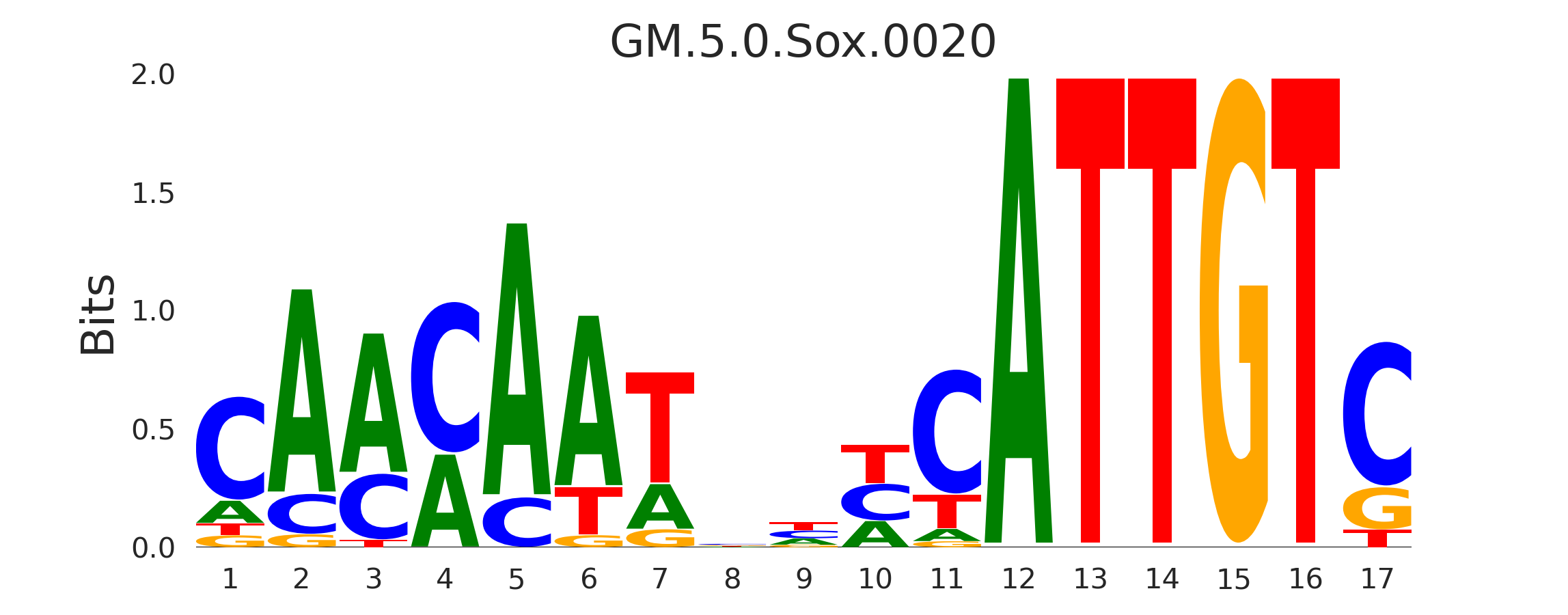
| GM.5.0.Sox.0020 |
SOX17 |
|
-1.53 |
-3.20 |
2.42 |
<1 |
0.01 |
-0.09 |
0.03 |
| GM.5.0.C2H2_ZF.0084 |
SP2,SP6,SP9,SP7,SP5, (...) |
|
-2.46 |
-3.45 |
2.68 |
3 |
-0.13 |
-0.08 |
0.16 |
| GM.5.0.Unknown.0016 |
|
|
-2.74 |
-1.15 |
3.21 |
<1 |
0.00 |
-0.04 |
0.02 |
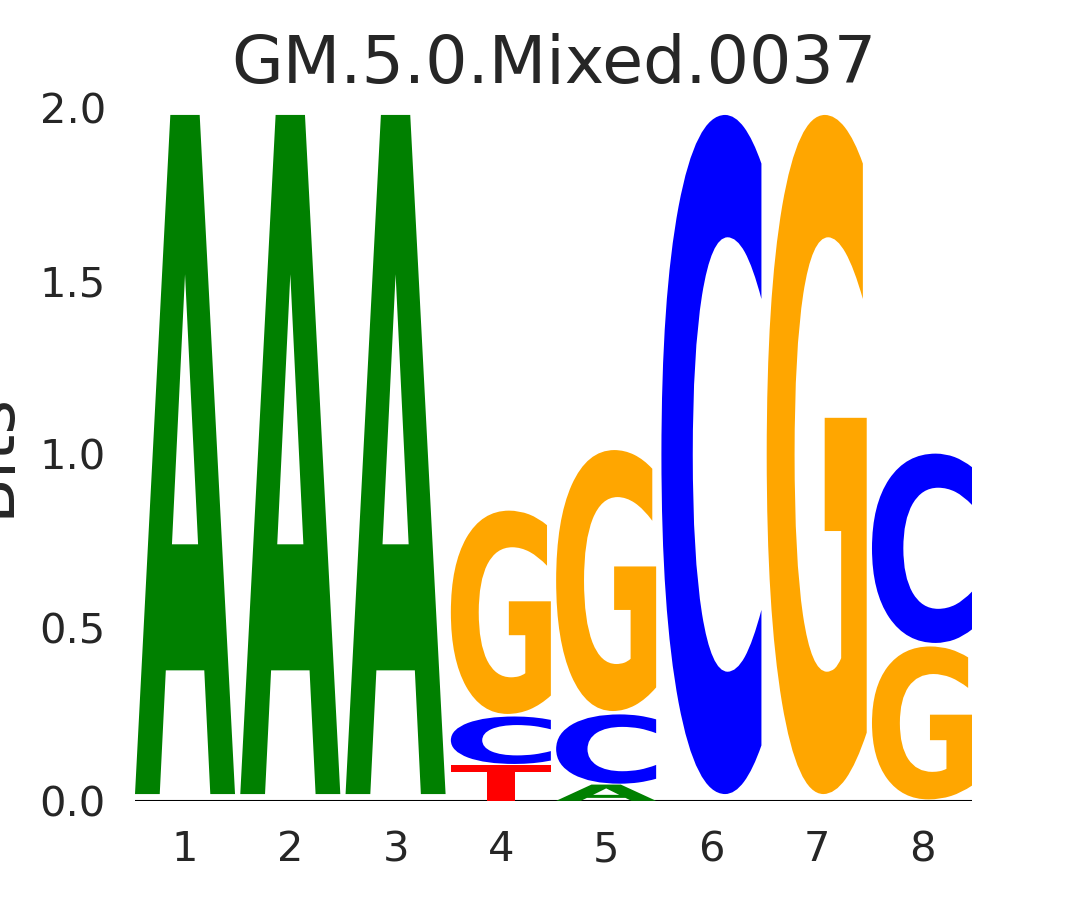
| GM.5.0.Mixed.0037 |
CHD2,E2F1 |
|
3.74 |
-3.77 |
-3.04 |
<1 |
0.13 |
-0.11 |
-0.06 |
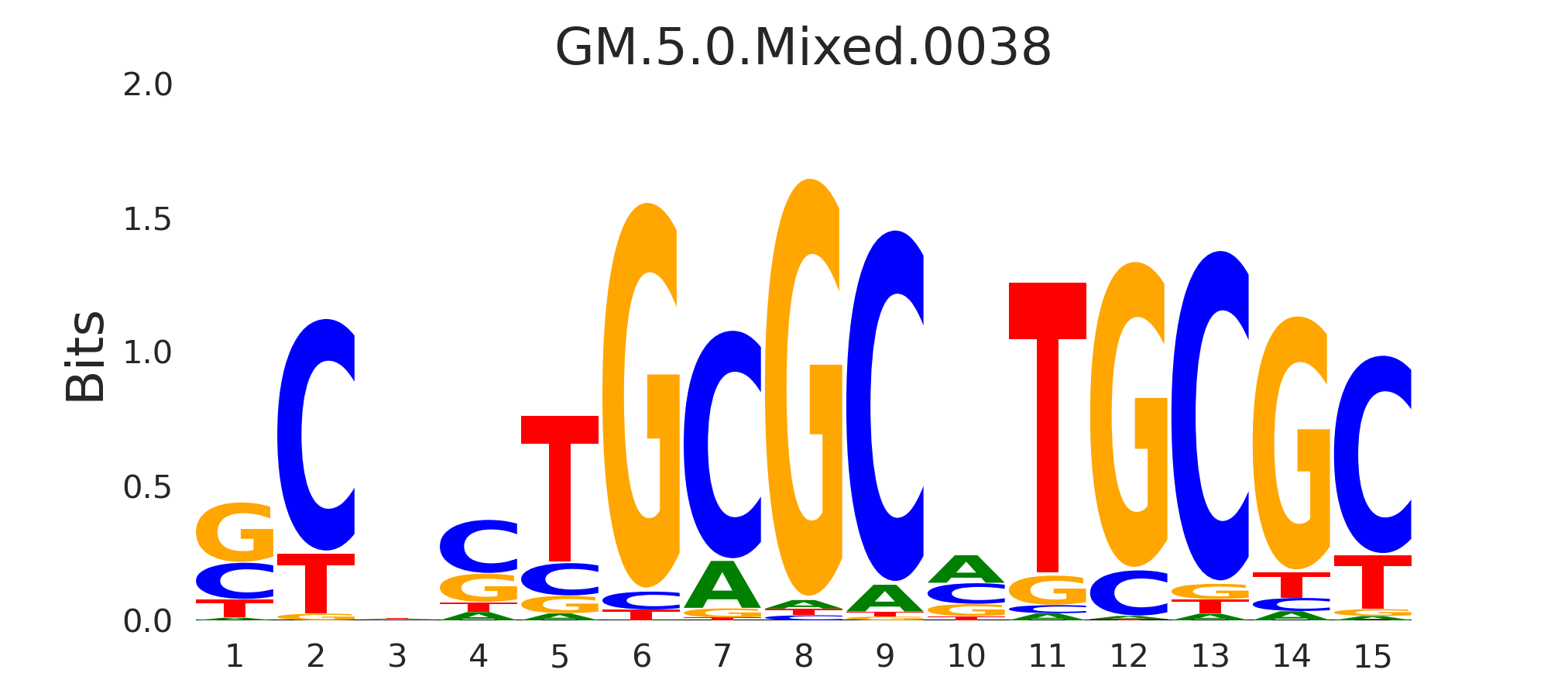
| GM.5.0.Mixed.0038 |
NRF1,EGR1 |
|
-3.48 |
-1.82 |
3.28 |
1 |
0.05 |
-0.09 |
0.00 |
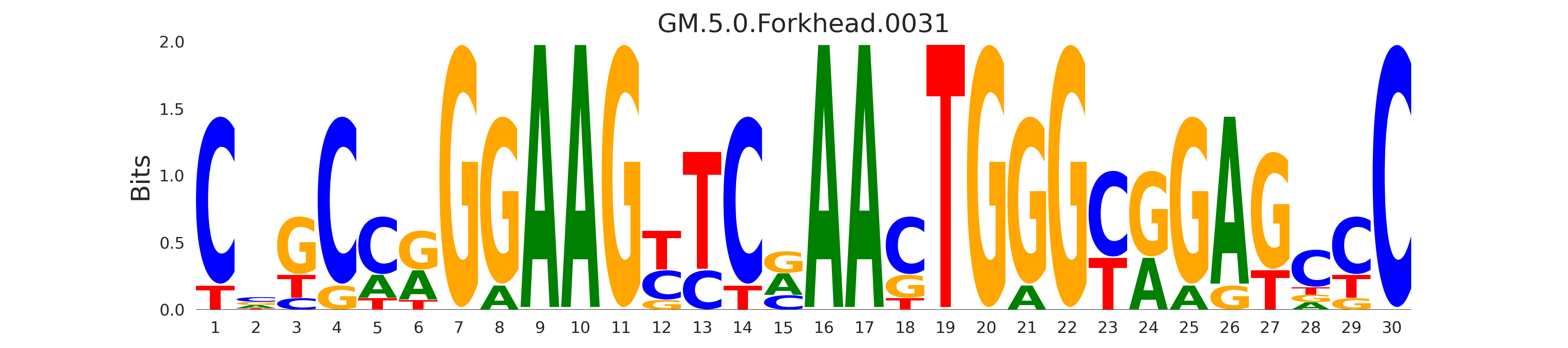
| GM.5.0.Forkhead.0031 |
FOXA1,FOXA2 |
|
1.46 |
3.53 |
-2.44 |
1 |
0.02 |
0.02 |
-0.03 |
| GM.5.0.C2H2_ZF.0108 |
ZIC3,ZIC4,ZIC1,ZIC5 |
|
-3.70 |
-1.42 |
3.67 |
1 |
-0.08 |
-0.10 |
0.13 |
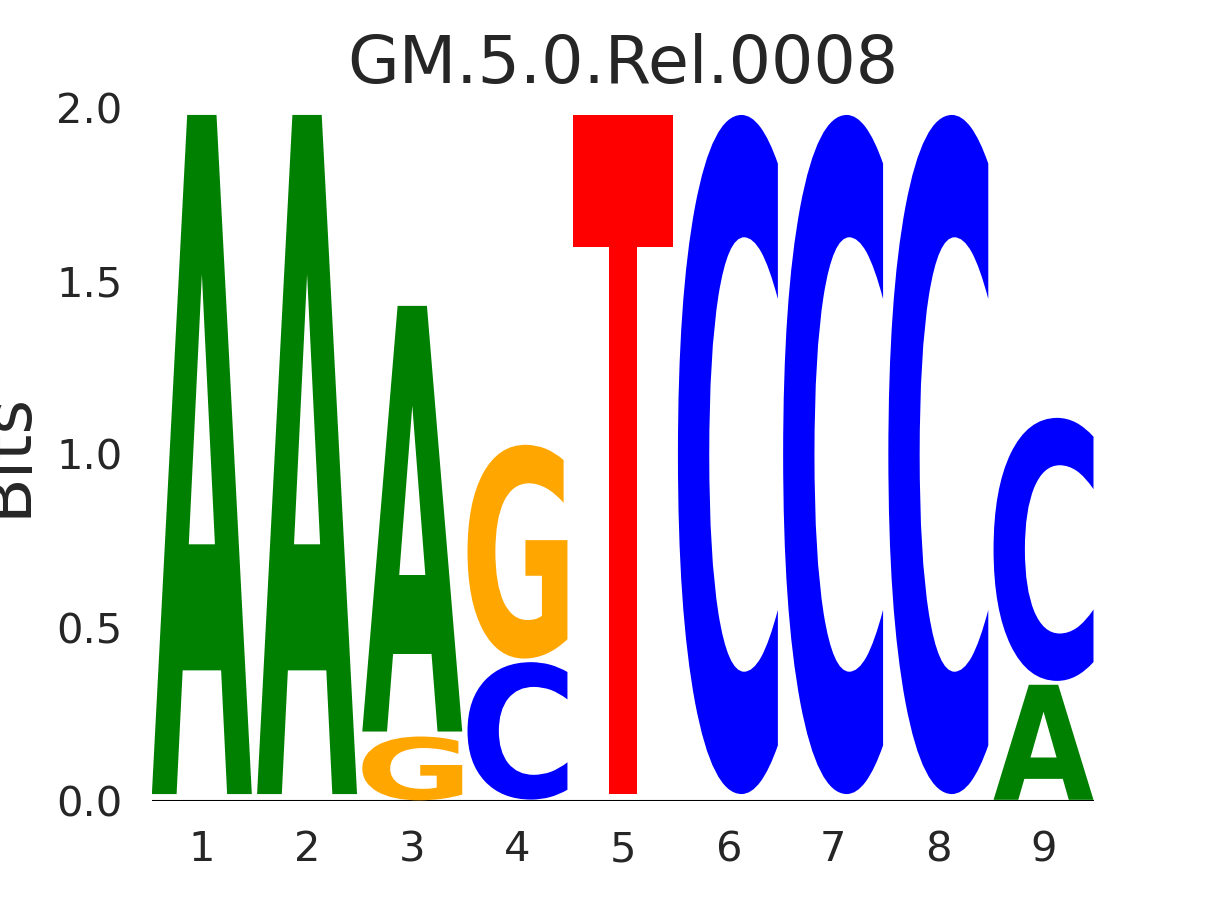
| GM.5.0.Rel.0008 |
NFKB1 |
|
2.25 |
-3.04 |
-1.32 |
1 |
0.03 |
-0.10 |
0.02 |
| GM.5.0.Unknown.0021 |
SPZ1 |
|
-3.49 |
-1.95 |
3.60 |
<1 |
-0.05 |
-0.04 |
0.07 |
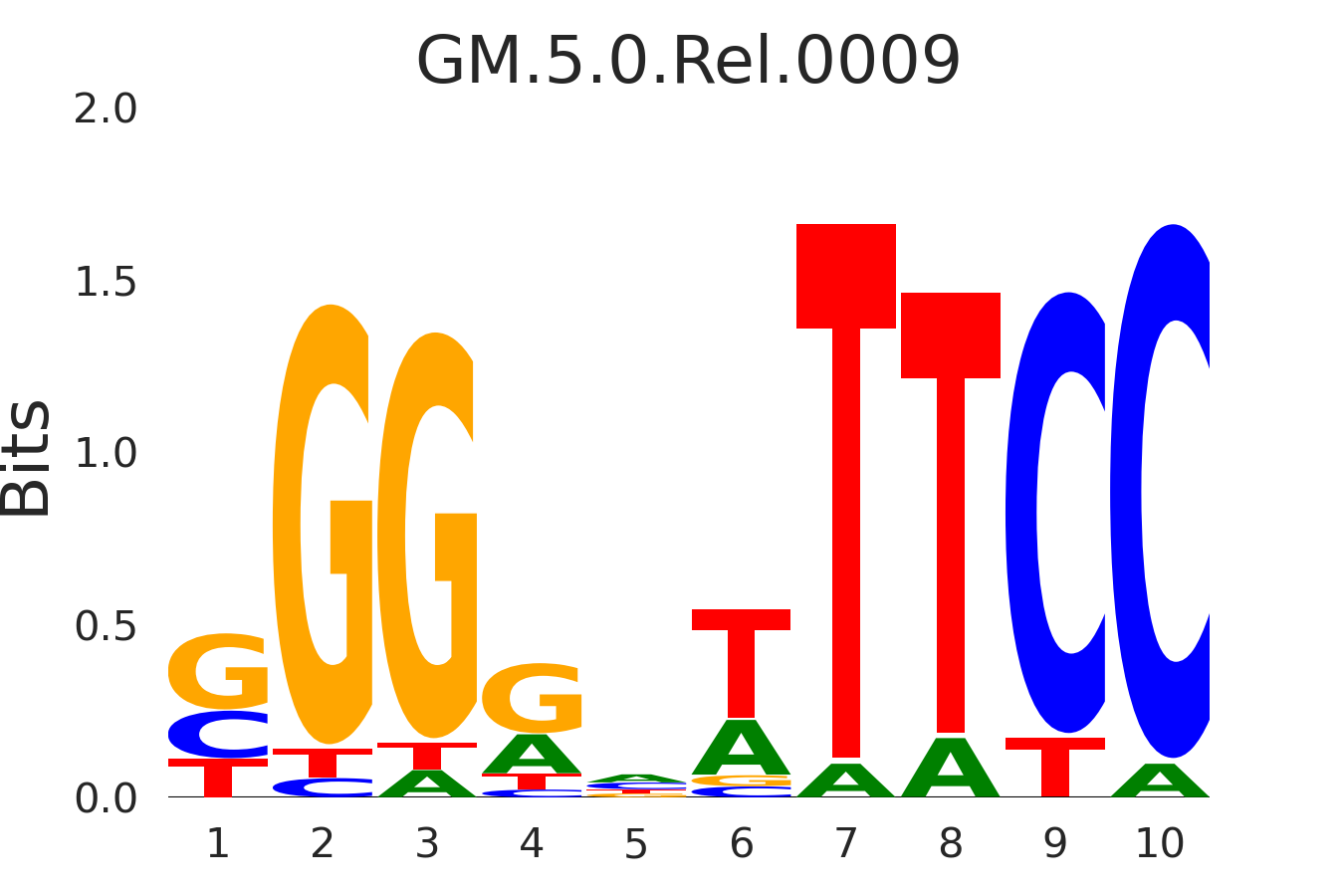
| GM.5.0.Rel.0009 |
REL |
|
-3.43 |
-0.04 |
3.01 |
2 |
-0.04 |
-0.09 |
0.08 |
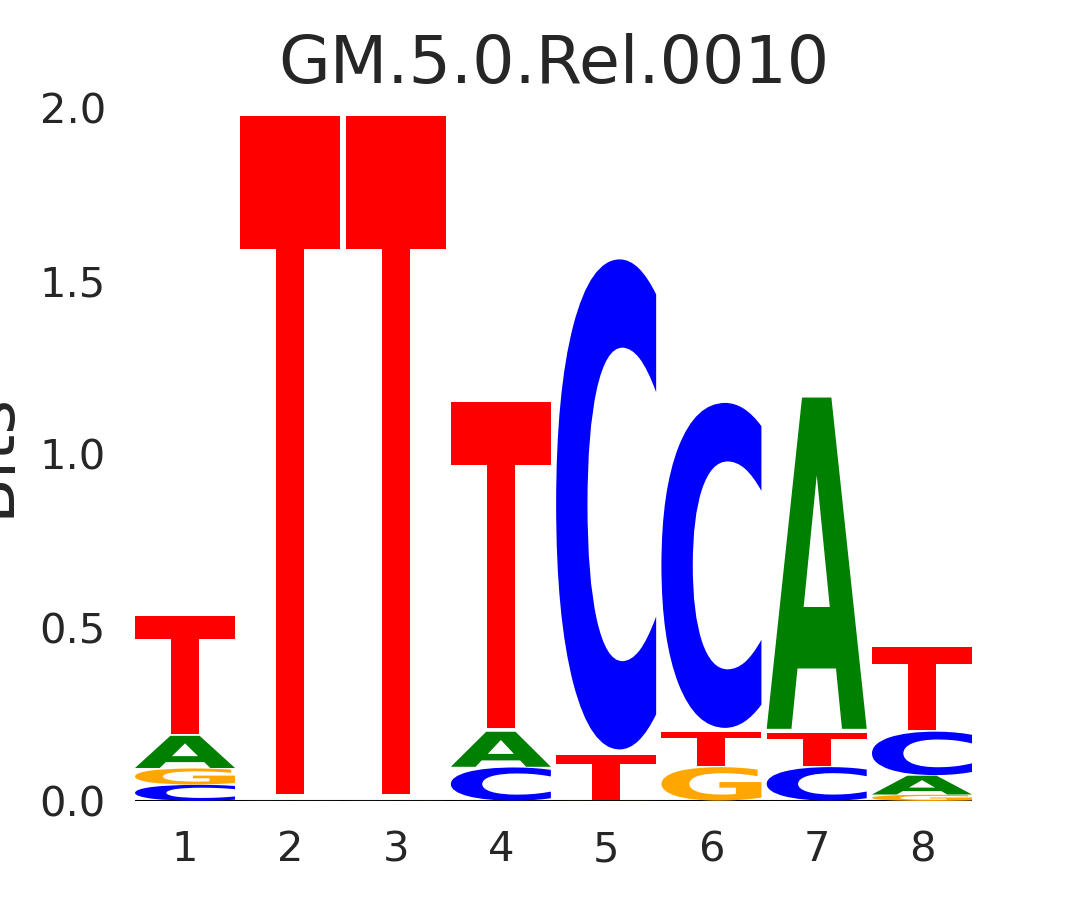
| GM.5.0.Rel.0010 |
NFAT5 |
|
2.15 |
2.90 |
-3.03 |
<1 |
0.07 |
0.00 |
-0.07 |
| GM.5.0.C2H2_ZF.0125 |
KLF6,KLF5,KLF3,KLF7,KLF2 |
|
-3.59 |
1.98 |
3.25 |
<1 |
-0.10 |
-0.07 |
0.13 |
| GM.5.0.C2H2_ZF.0126 |
ZFX,ZFY,ZFY1,ZFA |
|
-0.28 |
-4.15 |
3.12 |
<1 |
0.04 |
-0.12 |
0.03 |
| GM.5.0.E2F.0012 |
E2F1 |
|
3.52 |
-3.14 |
-3.20 |
2 |
0.19 |
-0.13 |
-0.11 |
| GM.5.0.C2H2_ZF.0129 |
ZFP410 |
|
1.28 |
3.97 |
-2.80 |
2 |
-0.07 |
0.11 |
0.01 |
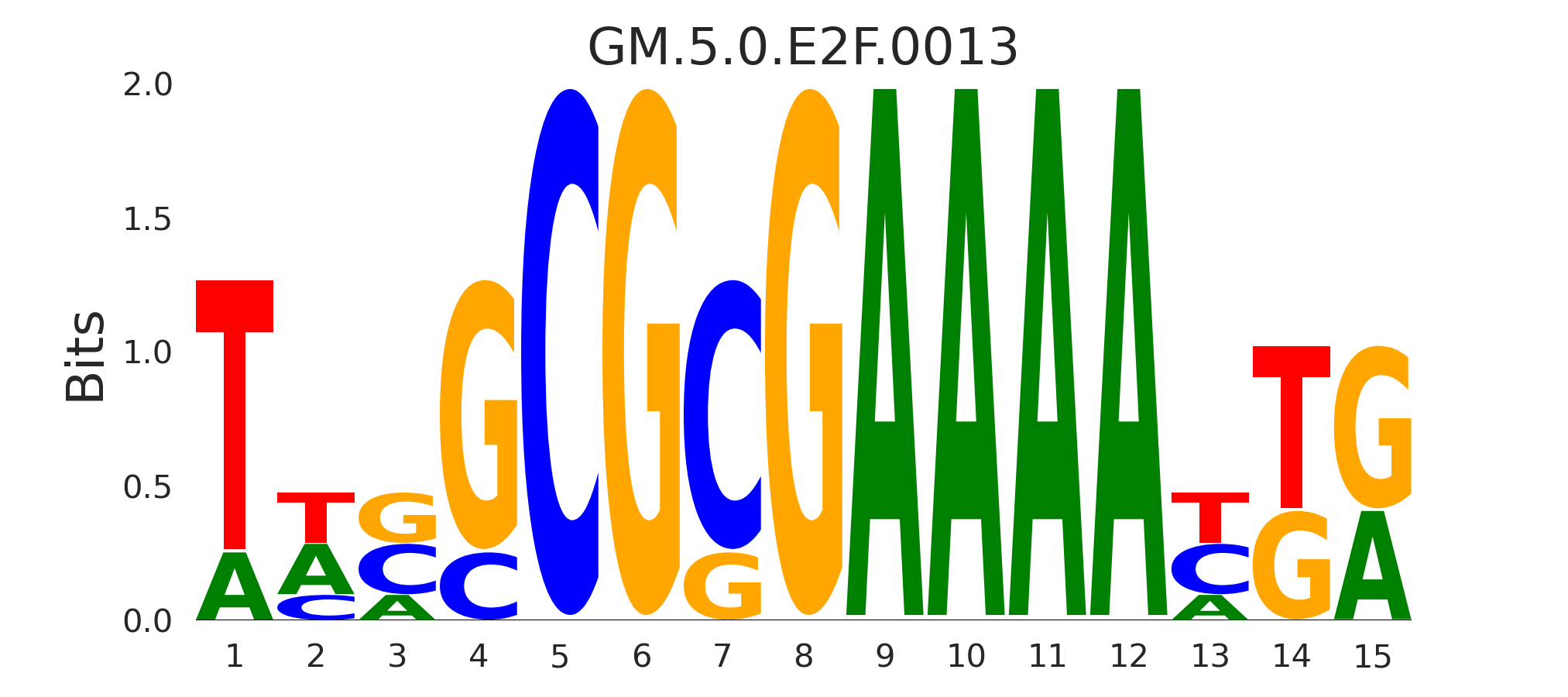
| GM.5.0.E2F.0013 |
E2F1 |
|
2.03 |
-3.11 |
-1.47 |
2 |
0.09 |
-0.13 |
-0.01 |
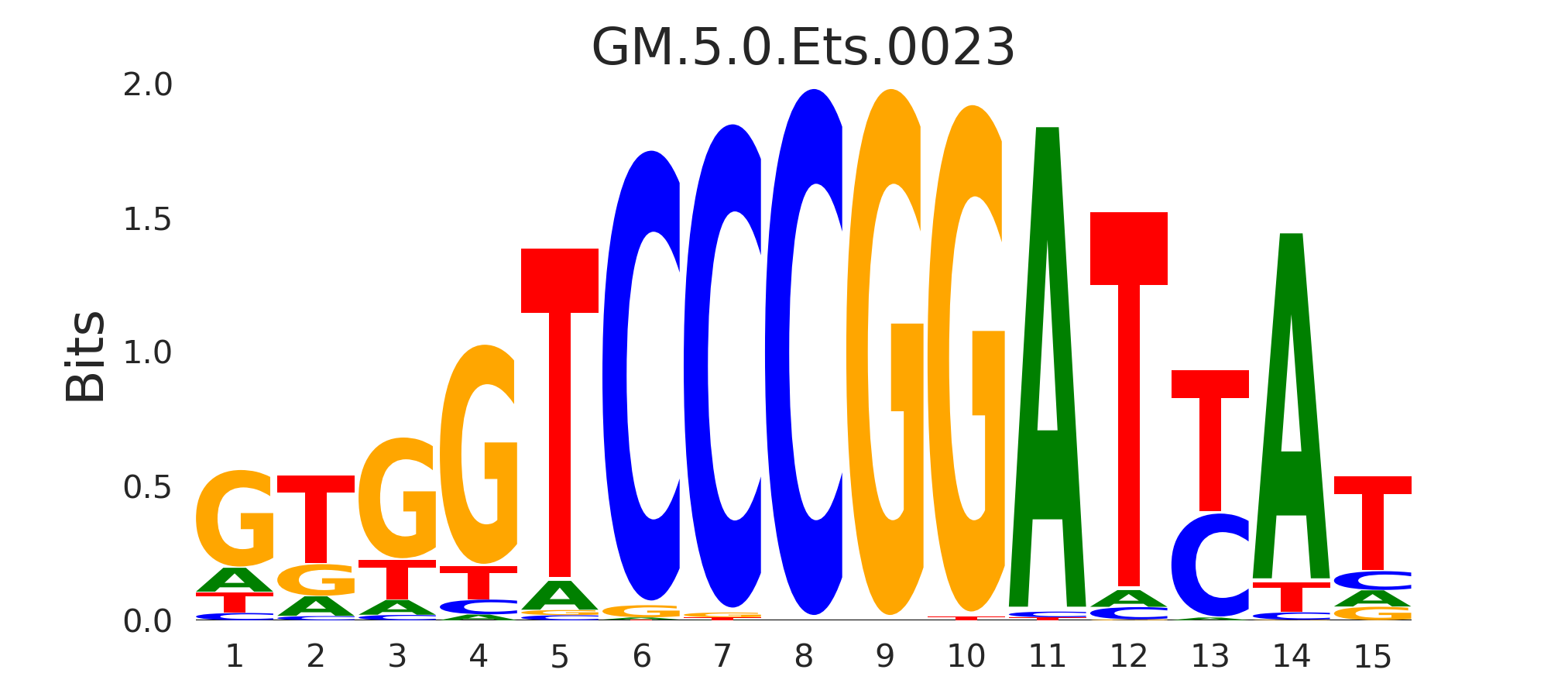
| GM.5.0.Ets.0023 |
SPDEF |
|
4.18 |
-4.23 |
-2.57 |
1 |
0.08 |
-0.14 |
-0.00 |
| GM.5.0.C2H2_ZF.0133 |
ZNF281,ZFP281,ZEB1,ZFP148,POU2F2 |
|
-3.21 |
0.69 |
3.28 |
2 |
-0.12 |
-0.03 |
0.12 |
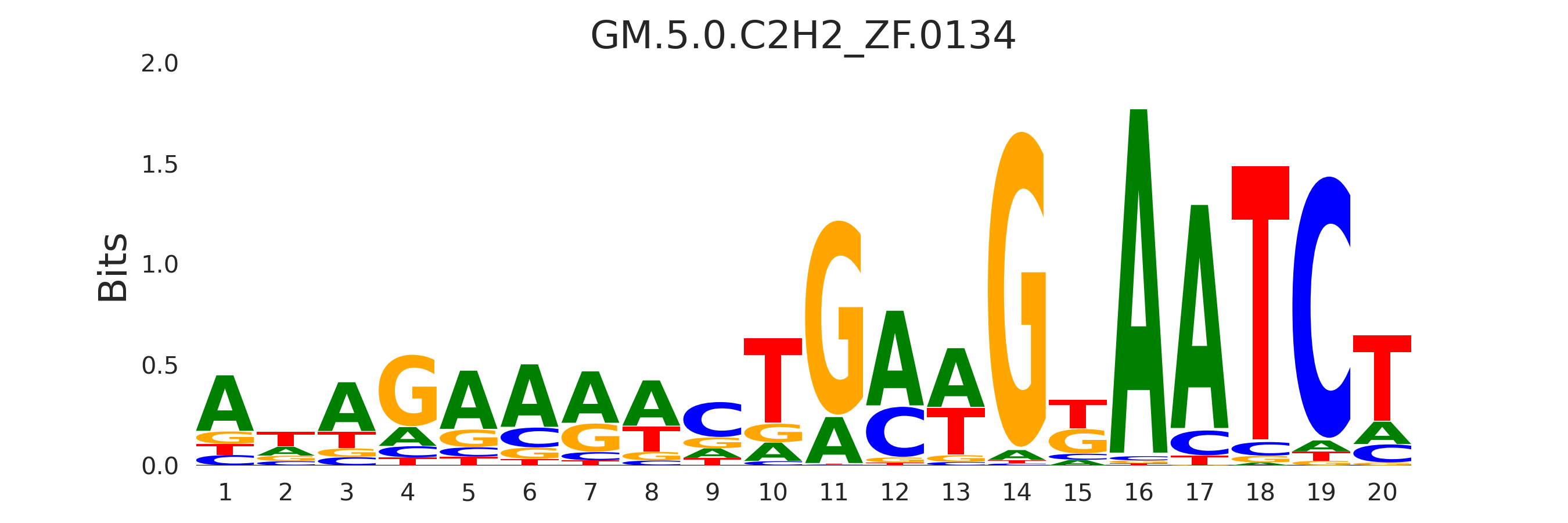
| GM.5.0.C2H2_ZF.0134 |
ZNF85 |
|
-2.97 |
3.32 |
1.63 |
4 |
-0.04 |
0.07 |
-0.00 |
| GM.5.0.Nuclear_receptor.0067 |
AR,NR3C2,PGR,HSF2 |
|
-2.85 |
-3.02 |
3.11 |
<1 |
-0.05 |
-0.12 |
0.11 |
| GM.5.0.C2H2_ZF.0136 |
SP6,SP9,SP7,SP5,SP4, (...) |
|
-5.64 |
3.24 |
5.40 |
<1 |
-0.14 |
-0.05 |
0.15 |
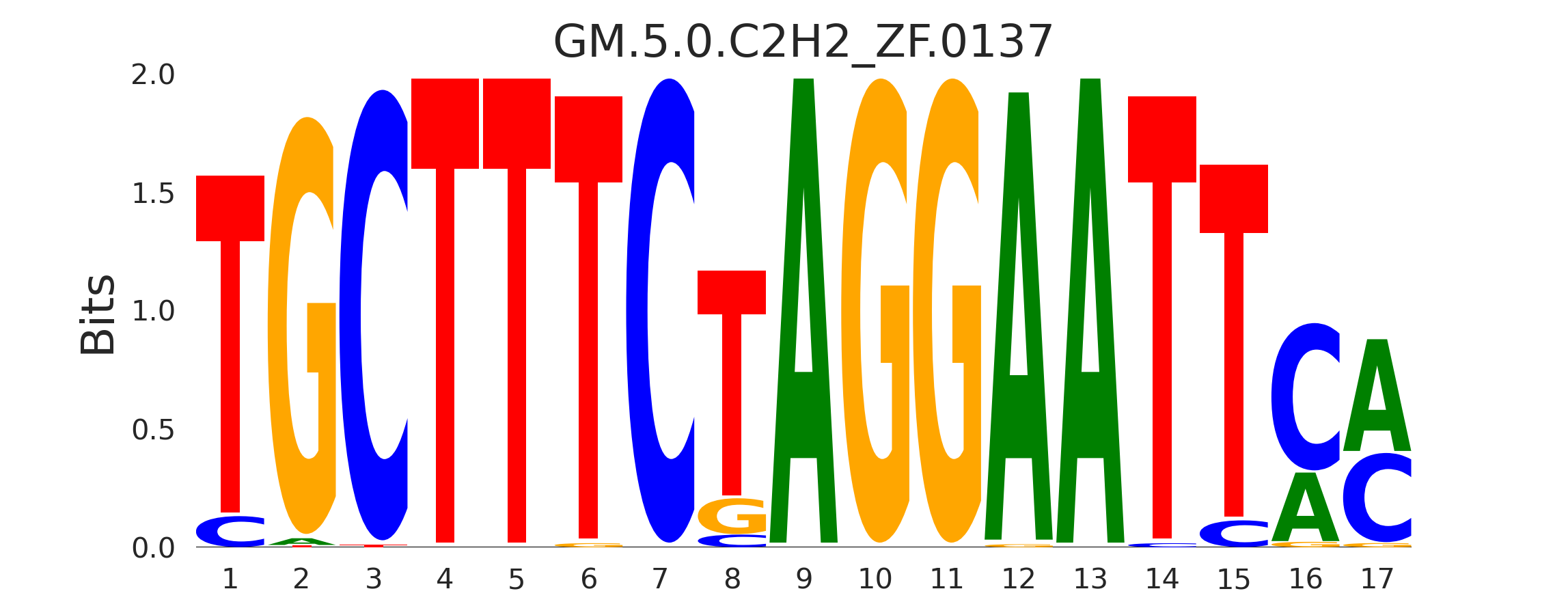
| GM.5.0.C2H2_ZF.0137 |
BCL6B |
|
3.62 |
-2.49 |
-2.98 |
1 |
-0.00 |
-0.04 |
0.02 |
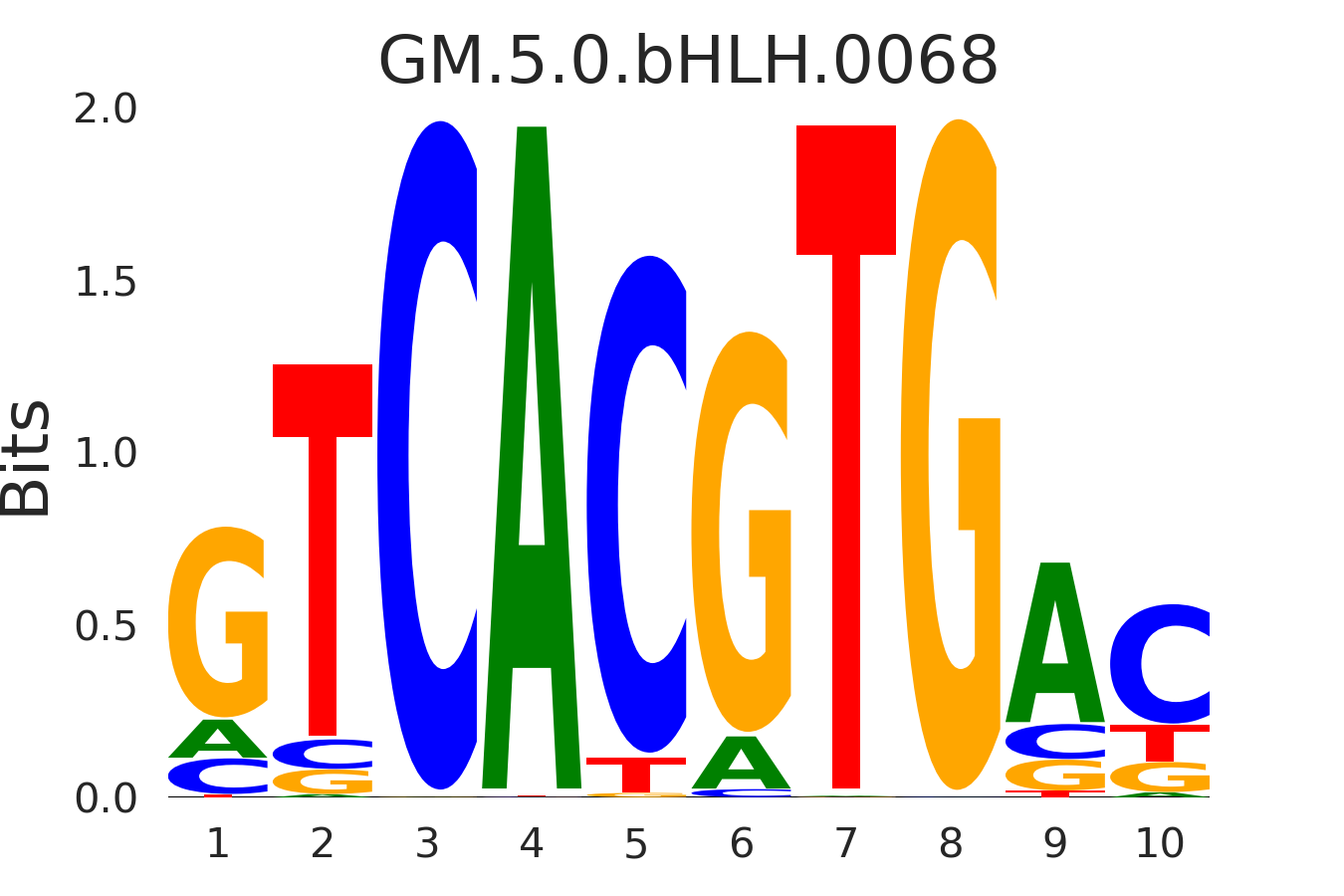
| GM.5.0.bHLH.0068 |
USF2,BHLHE40,MITF,ATF3,MAX, (...) |
|
-3.29 |
2.49 |
2.98 |
1 |
-0.09 |
-0.03 |
0.10 |
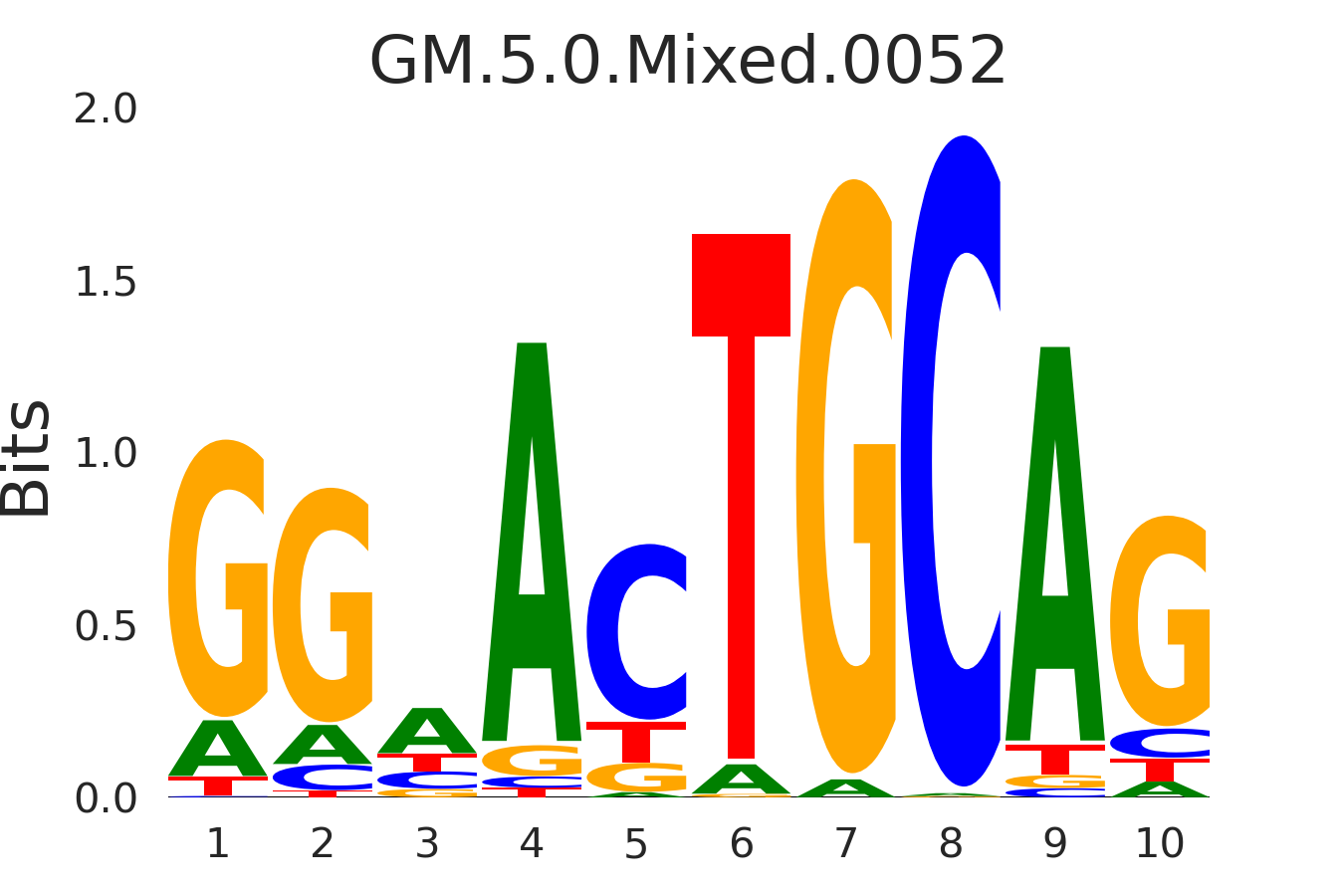
| GM.5.0.Mixed.0052 |
CTCF,RAD21,SMC3,CTCFL |
|
-3.07 |
-0.49 |
2.97 |
4 |
-0.11 |
-0.07 |
0.13 |
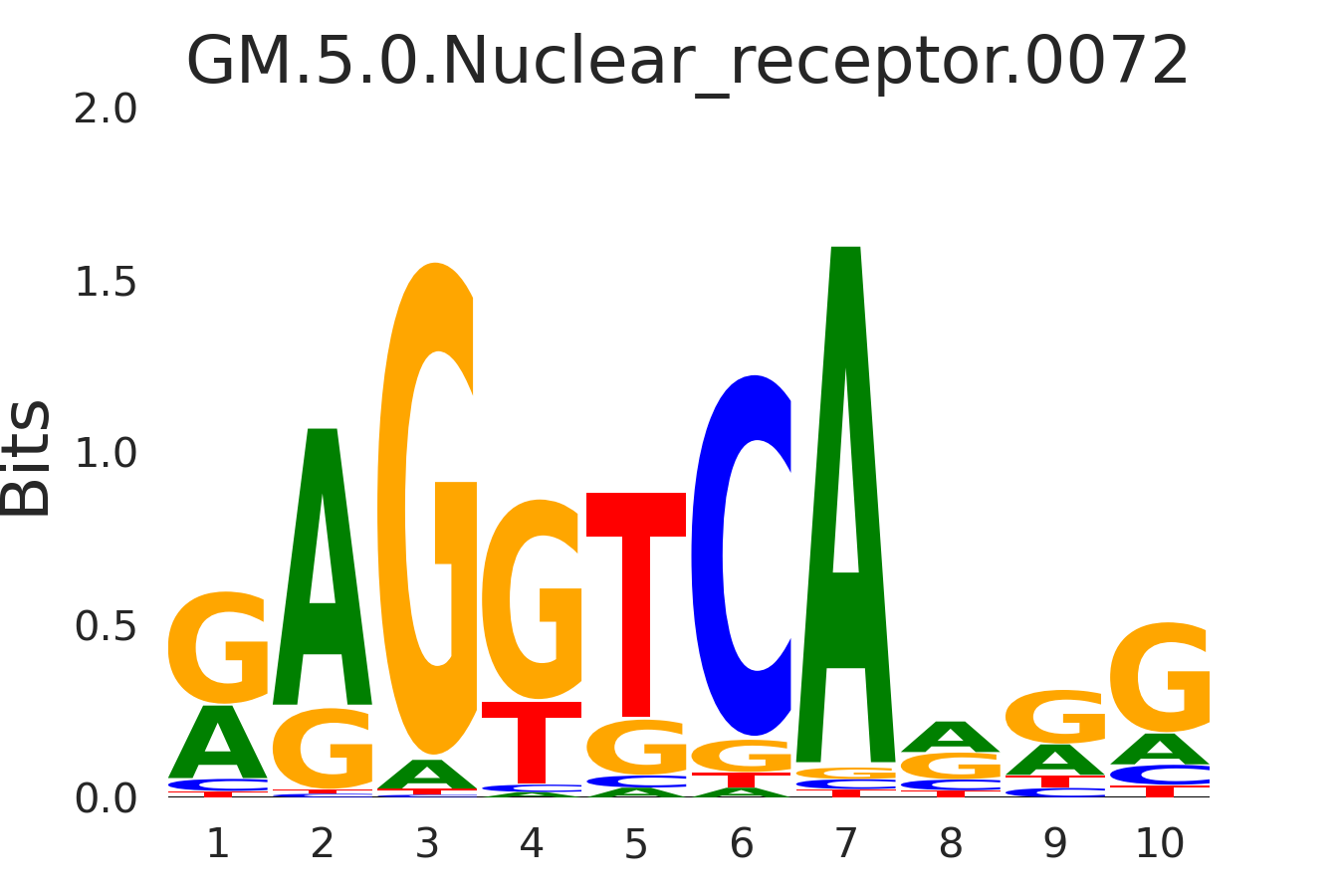
| GM.5.0.Nuclear_receptor.0072 |
RXRA,RARA |
|
-0.36 |
3.16 |
-2.27 |
<1 |
-0.03 |
-0.00 |
0.03 |
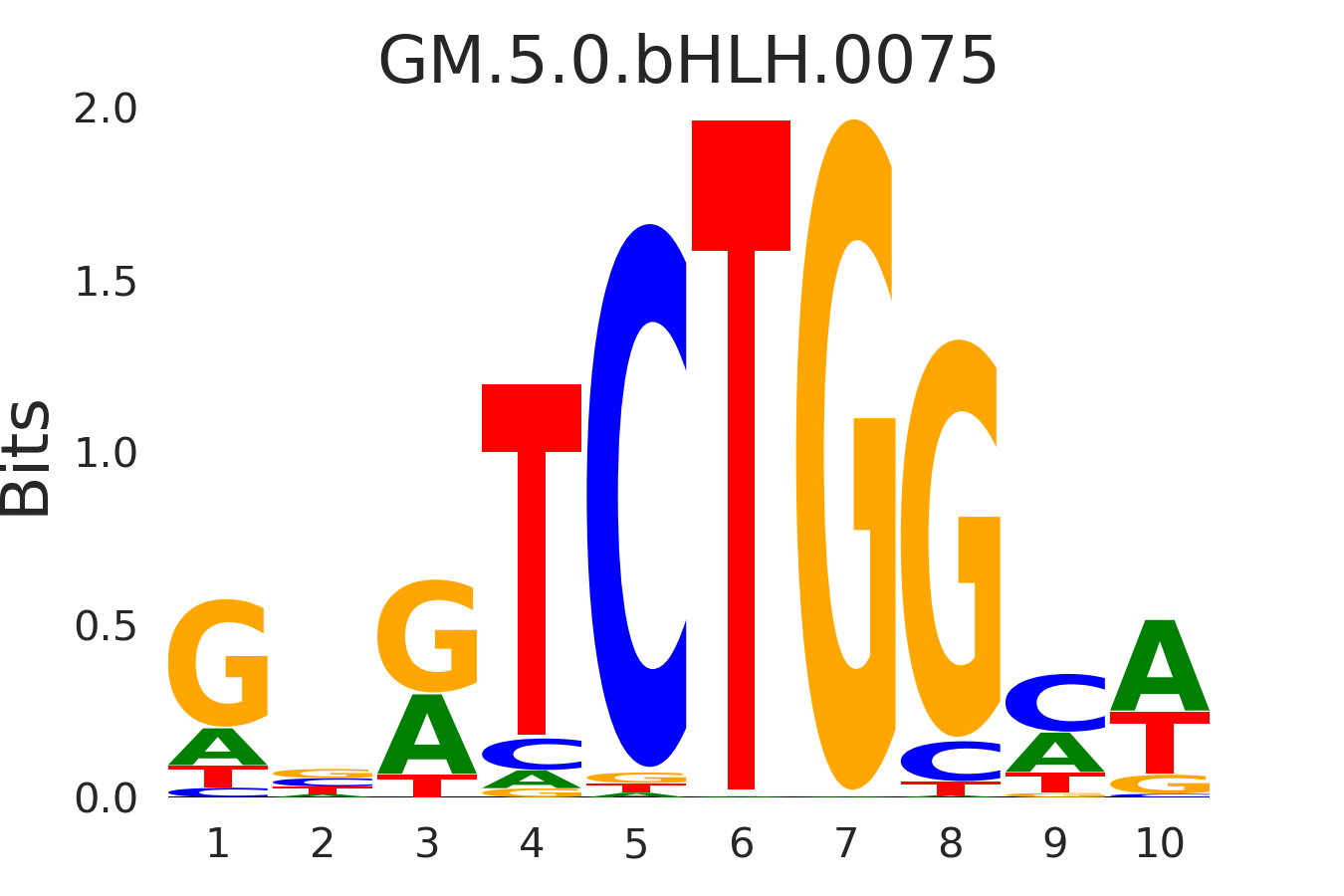
| GM.5.0.bHLH.0075 |
HAND1,HAND2,TCF3 |
|
1.81 |
-3.07 |
0.67 |
1 |
0.01 |
-0.07 |
0.03 |
| GM.5.0.GATA.0013 |
GATA2 |
|
5.88 |
-4.60 |
-5.88 |
<1 |
0.16 |
-0.04 |
-0.12 |
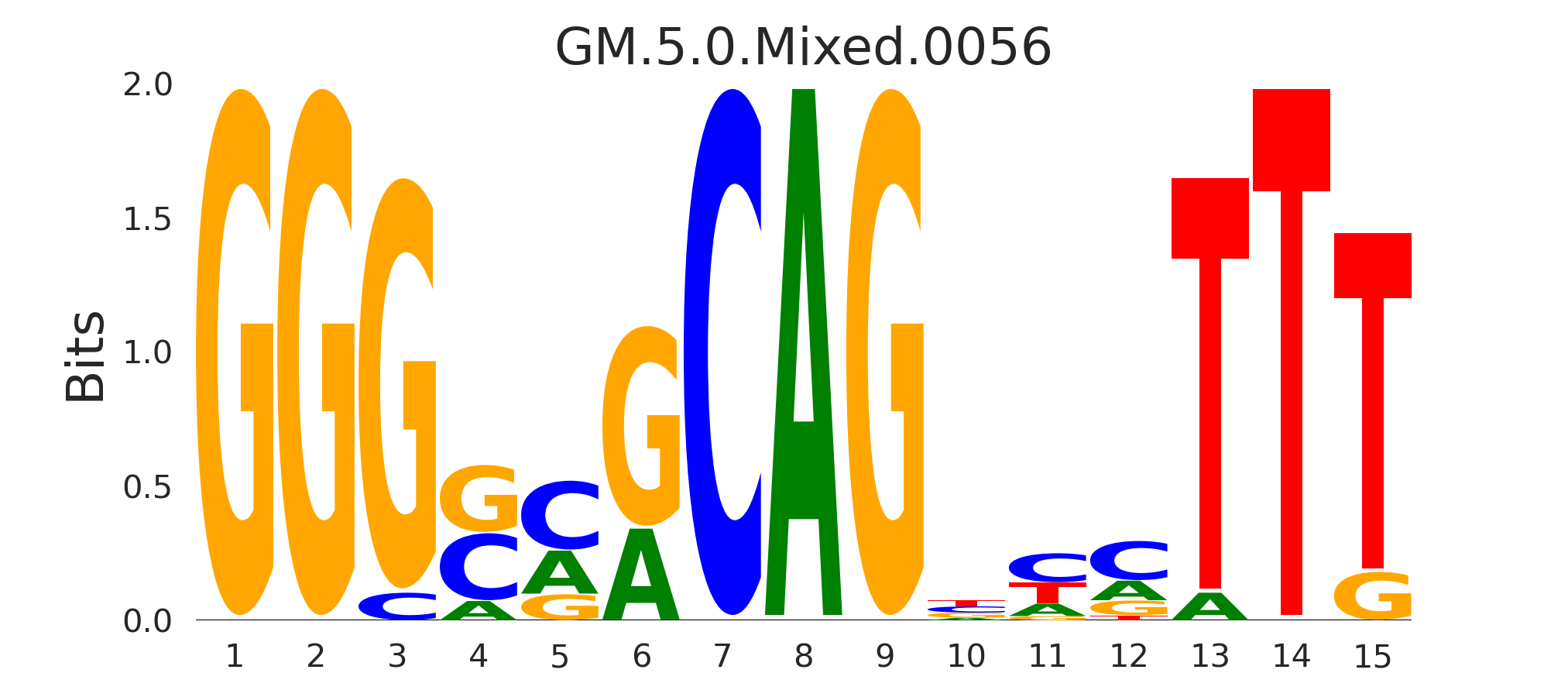
| GM.5.0.Mixed.0056 |
ZNF350 |
|
3.18 |
-1.64 |
-2.90 |
<1 |
0.03 |
-0.09 |
0.02 |
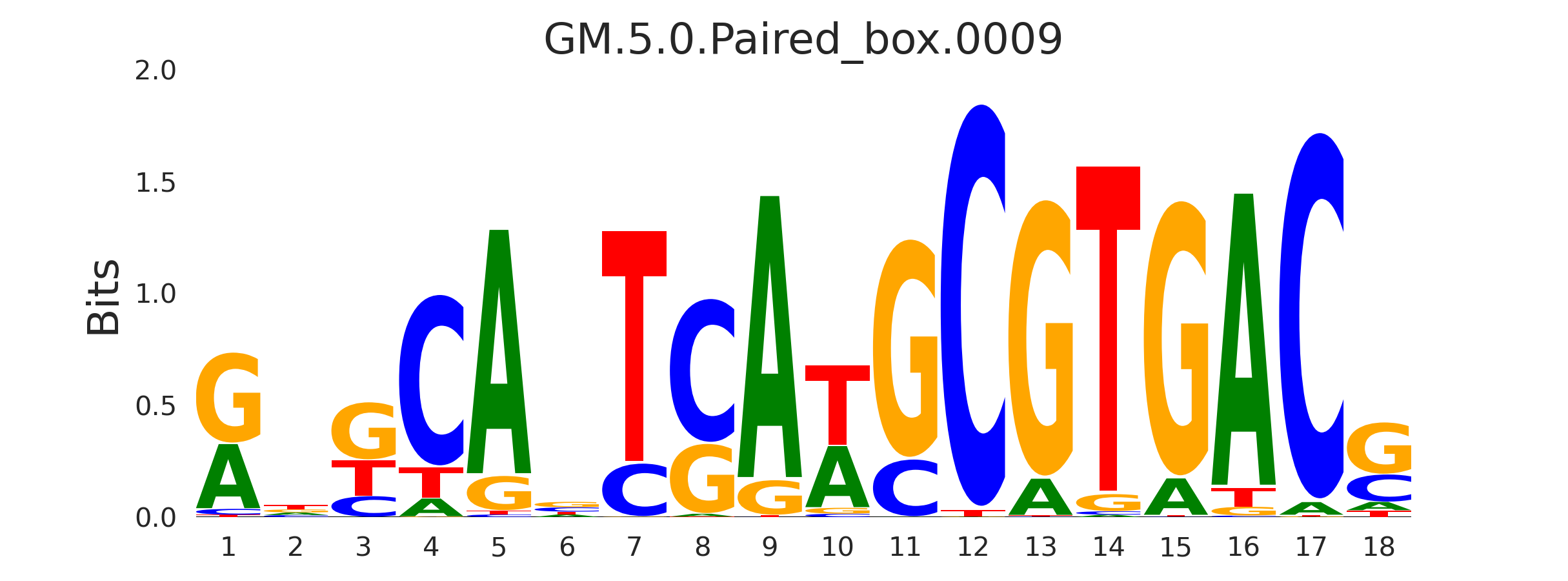
| GM.5.0.Paired_box.0009 |
PAX9,PAX1,PAX5,PAX2,PAX8 |
|
2.90 |
-0.71 |
-3.03 |
1 |
0.07 |
-0.01 |
-0.06 |
| GM.5.0.bZIP.0057 |
MAF |
|
-3.35 |
0.55 |
3.02 |
2 |
-0.02 |
0.02 |
0.01 |
| GM.5.0.bZIP.0059 |
NFE2L1,MAFG |
|
-3.28 |
-2.22 |
3.58 |
<1 |
-0.03 |
-0.04 |
0.05 |
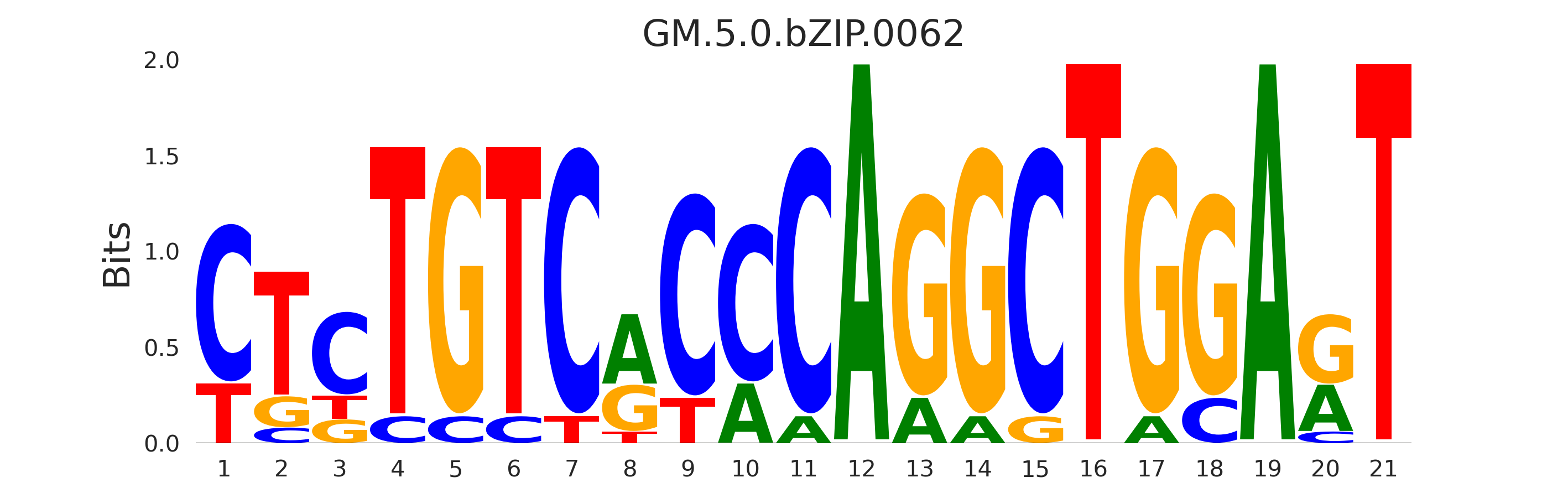
| GM.5.0.bZIP.0062 |
CEBPB |
|
3.54 |
1.14 |
-3.33 |
<1 |
0.05 |
0.03 |
-0.05 |
| GM.5.0.Unknown.0056 |
|
|
-4.52 |
-5.23 |
5.20 |
<1 |
-0.12 |
-0.06 |
0.14 |
| GM.5.0.Unknown.0058 |
CCNT2 |
|
3.80 |
-0.83 |
-3.84 |
<1 |
-0.06 |
-0.06 |
0.08 |
| GM.5.0.RFX.0009 |
RFX5 |
|
3.02 |
1.27 |
-2.96 |
<1 |
0.06 |
-0.06 |
-0.03 |
| GM.5.0.Unknown.0068 |
ZBTB14 |
|
2.97 |
1.58 |
-3.39 |
<1 |
0.20 |
-0.05 |
-0.16 |
| GM.5.0.GTF2I-like.0001 |
GTF2I |
|
-4.41 |
-2.99 |
4.66 |
<1 |
-0.11 |
-0.08 |
0.14 |
| GM.5.0.Nuclear_receptor.0087 |
THRB,THRA |
|
2.55 |
2.67 |
-3.36 |
<1 |
0.02 |
-0.02 |
-0.01 |
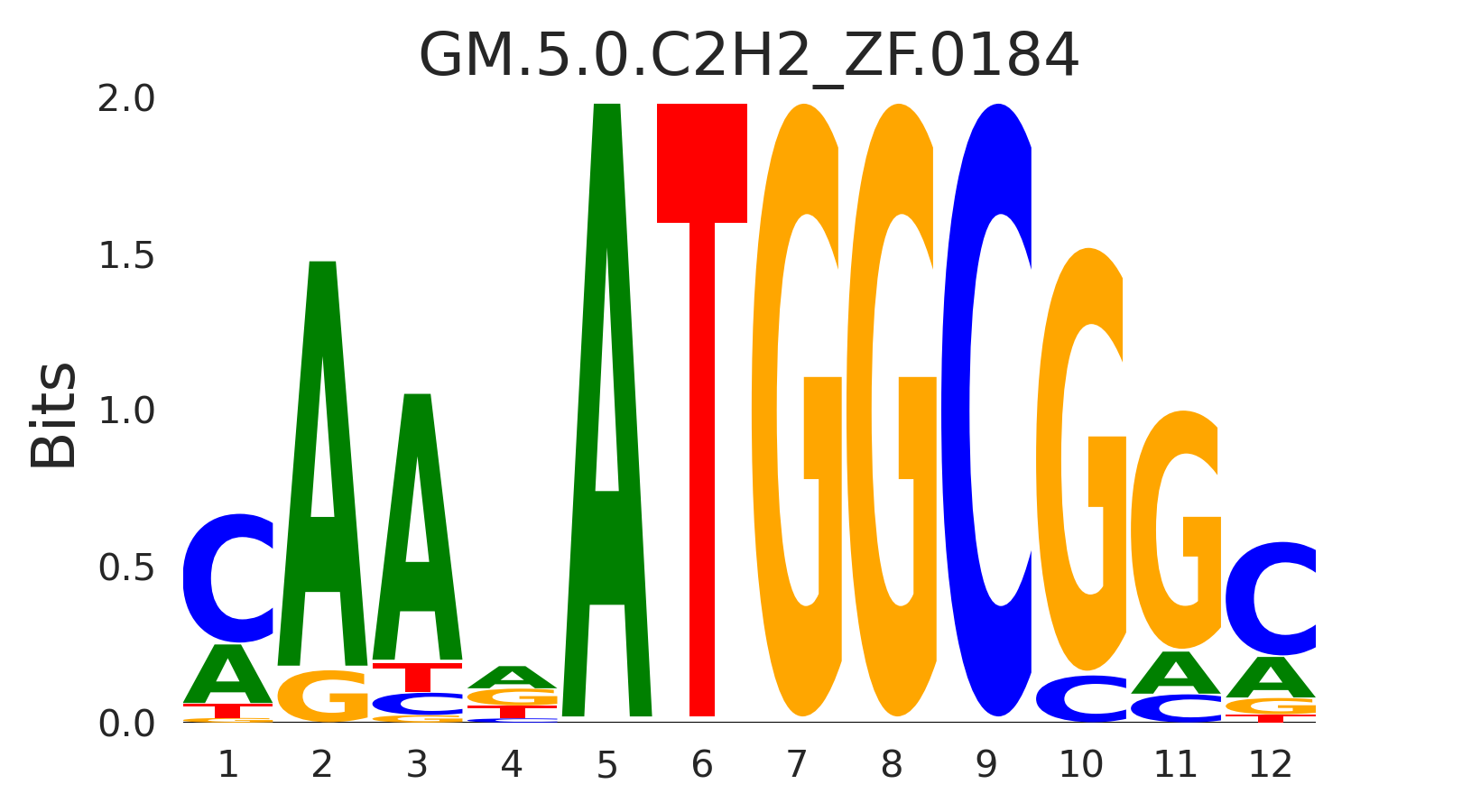
| GM.5.0.C2H2_ZF.0184 |
MBTPS2,YY1 |
|
-4.20 |
-3.24 |
4.21 |
1 |
-0.09 |
-0.12 |
0.14 |
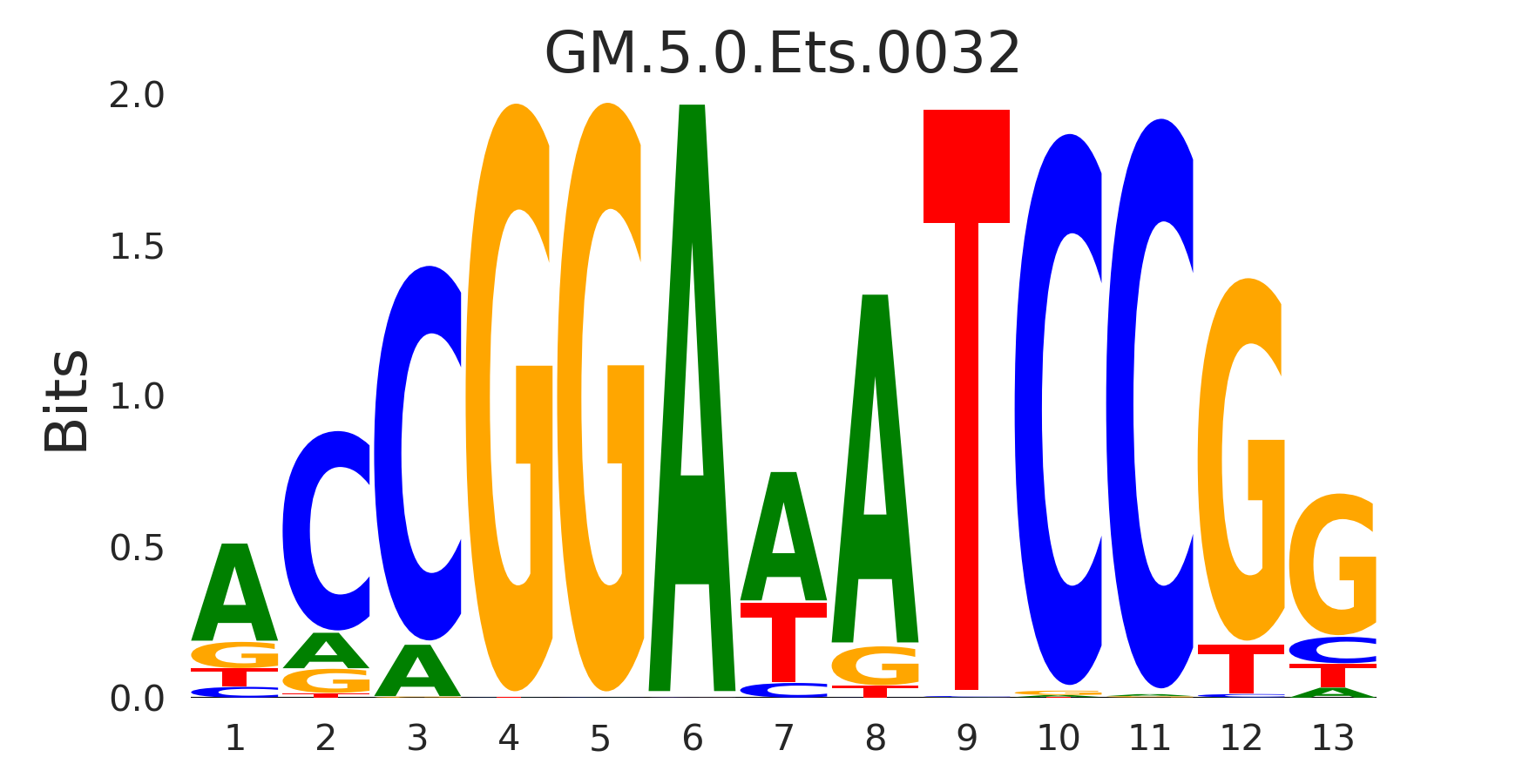
| GM.5.0.Ets.0032 |
FLI1,ERG,ERF,ENSMUSG00000044690,ETS2, (...) |
|
3.09 |
-1.32 |
-3.14 |
1 |
0.06 |
-0.09 |
-0.01 |
| GM.5.0.Unknown.0074 |
|
|
-3.63 |
-5.26 |
4.72 |
7 |
-0.22 |
-0.16 |
0.28 |
| GM.5.0.C2H2_ZF.0187 |
PATZ1 |
|
-3.23 |
1.84 |
2.99 |
4 |
-0.13 |
-0.05 |
0.14 |
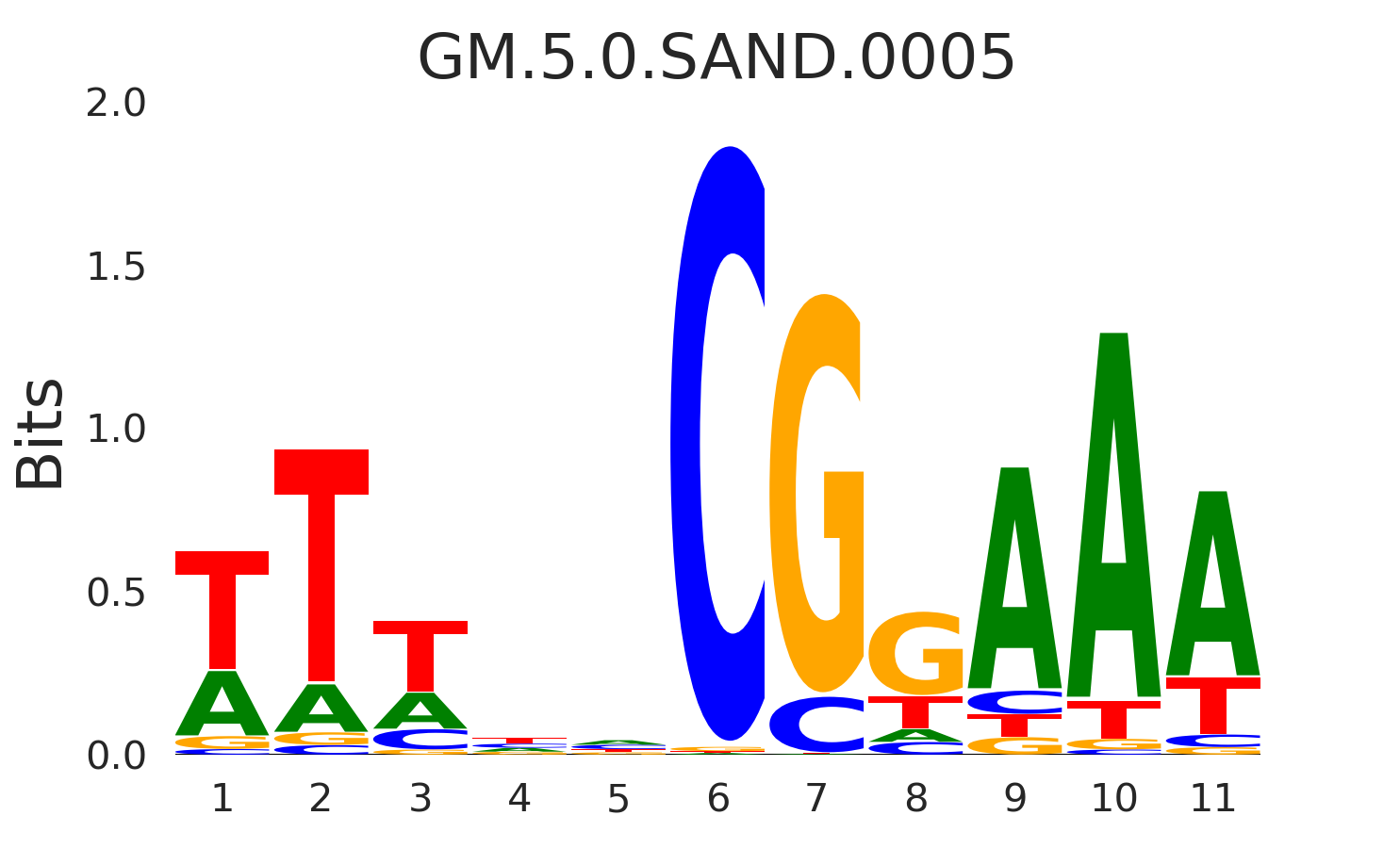
| GM.5.0.SAND.0005 |
SP100 |
|
3.22 |
-0.52 |
-3.40 |
2 |
0.13 |
-0.07 |
-0.08 |
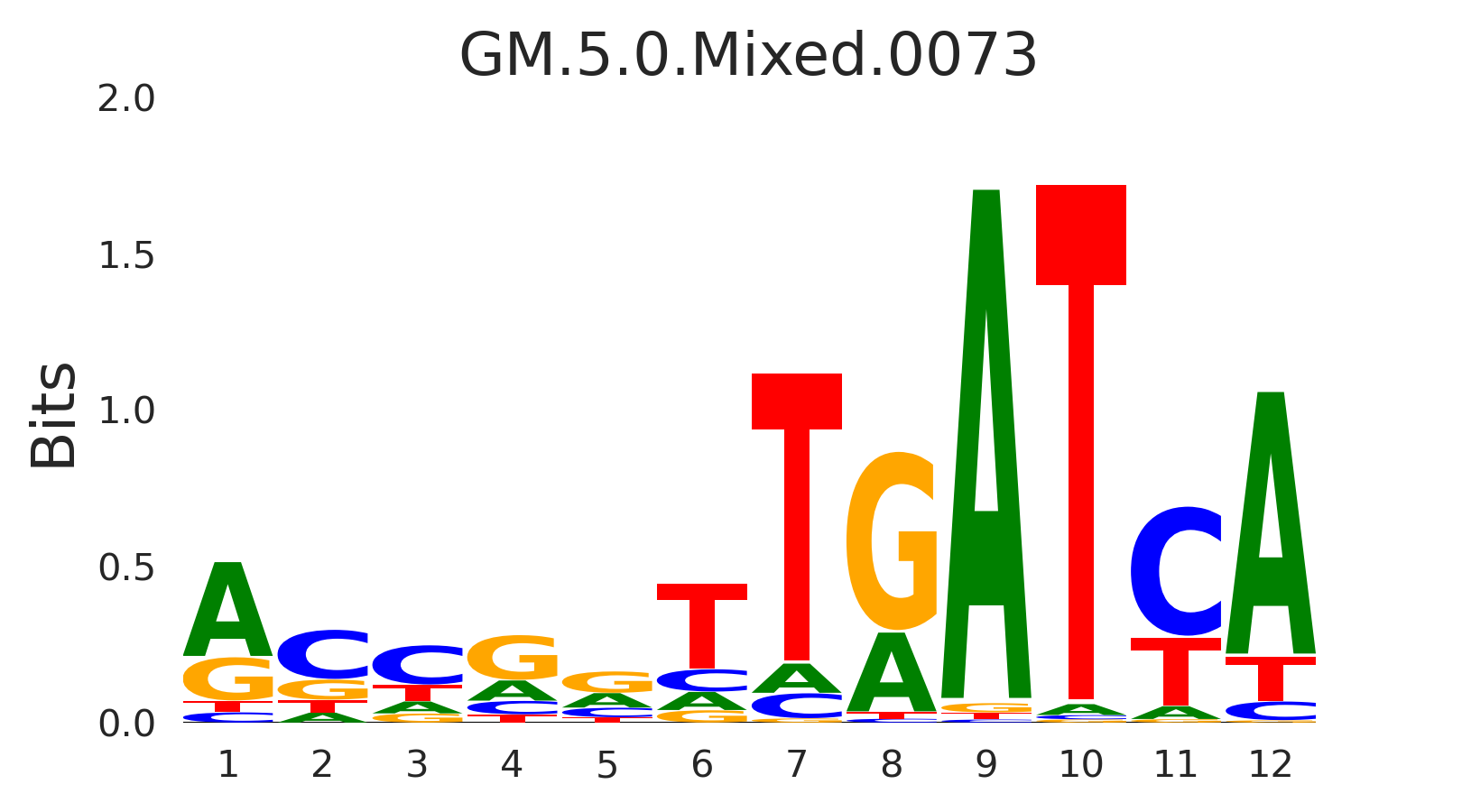
| GM.5.0.Mixed.0073 |
CUX1 |
|
2.60 |
-0.52 |
-3.05 |
1 |
0.06 |
-0.04 |
-0.04 |
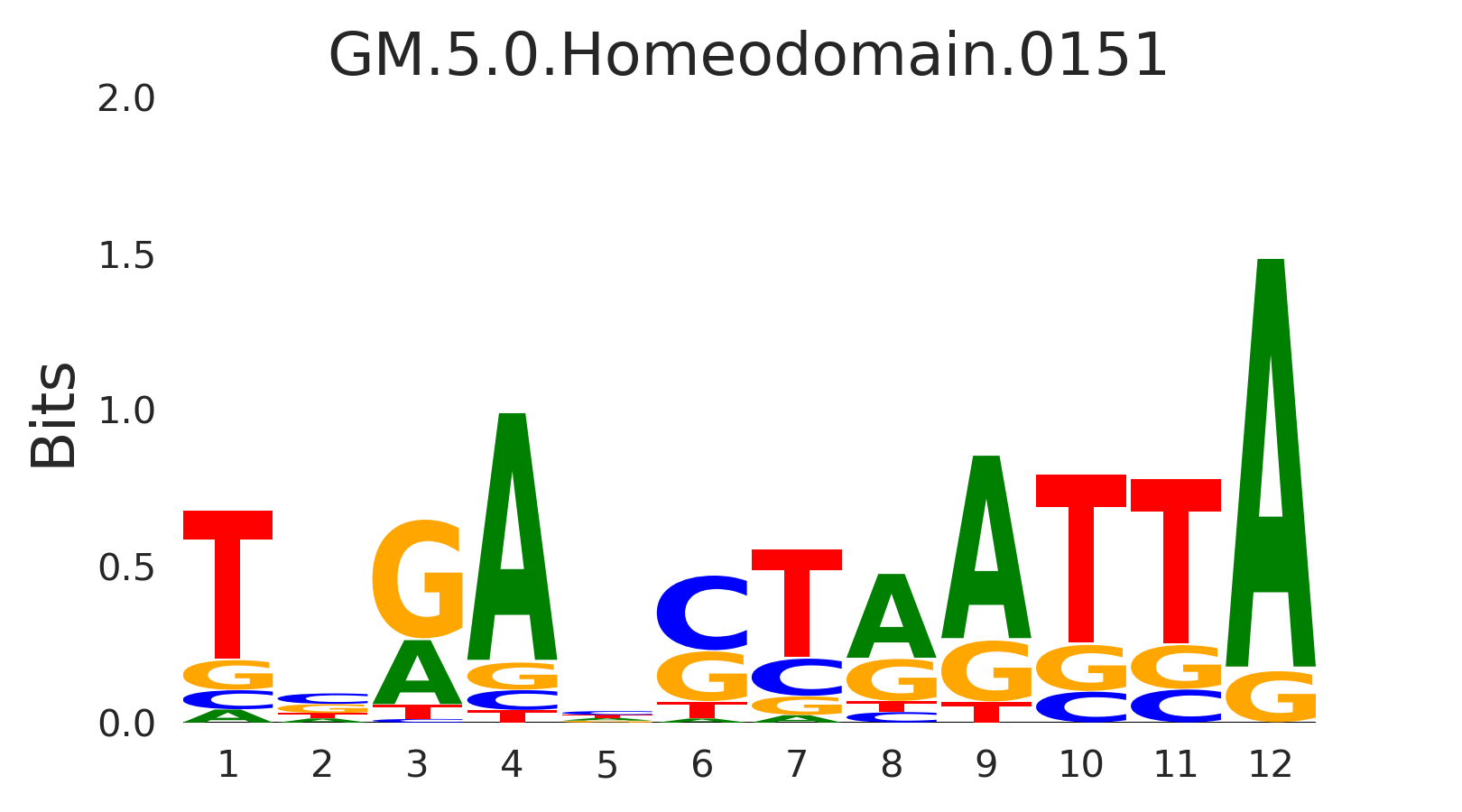
| GM.5.0.Homeodomain.0151 |
LBX2,LBX1 |
|
3.54 |
-0.76 |
-3.09 |
<1 |
0.06 |
0.02 |
-0.06 |
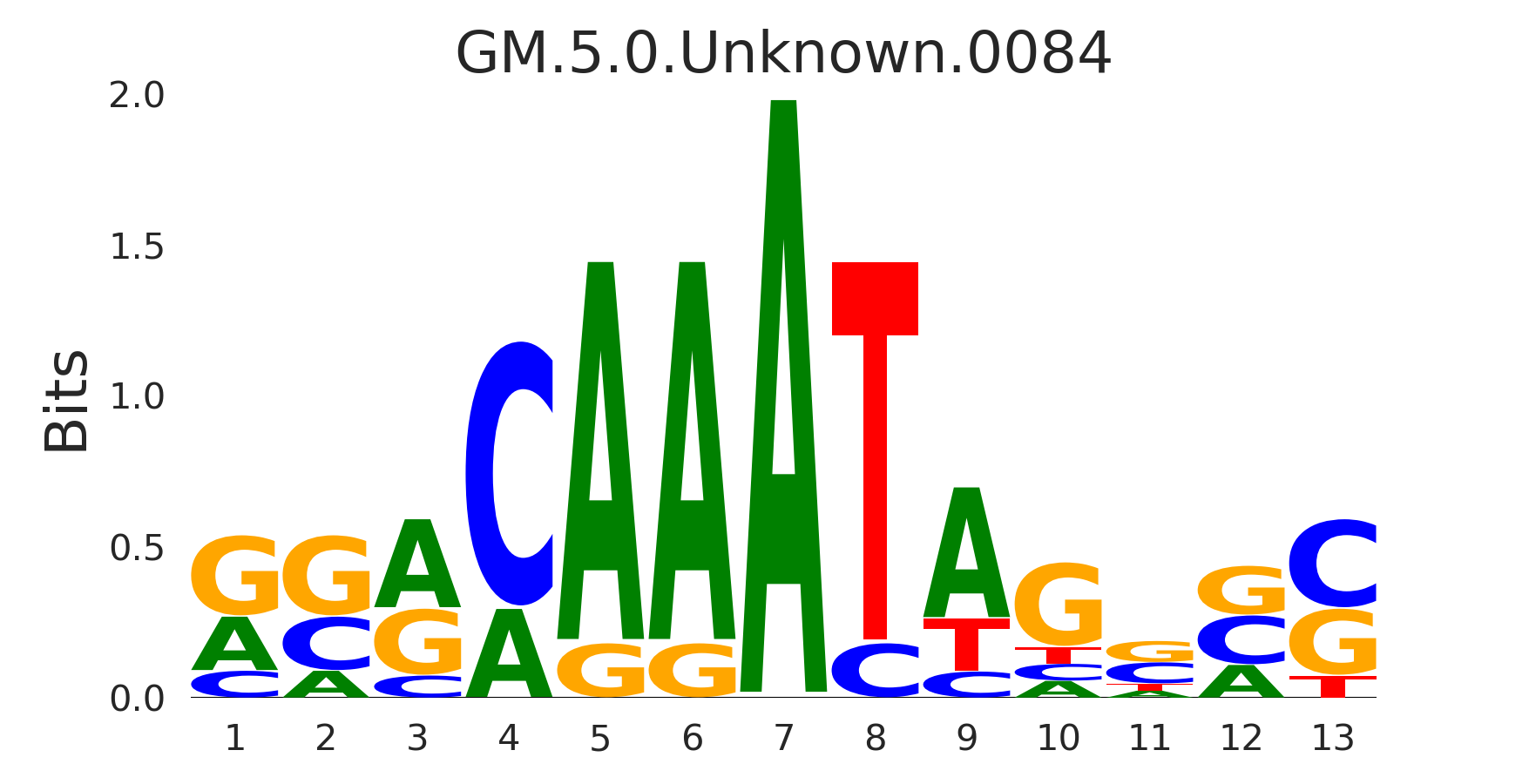
| GM.5.0.Unknown.0084 |
|
|
1.66 |
-4.31 |
1.55 |
2 |
0.01 |
-0.15 |
0.06 |
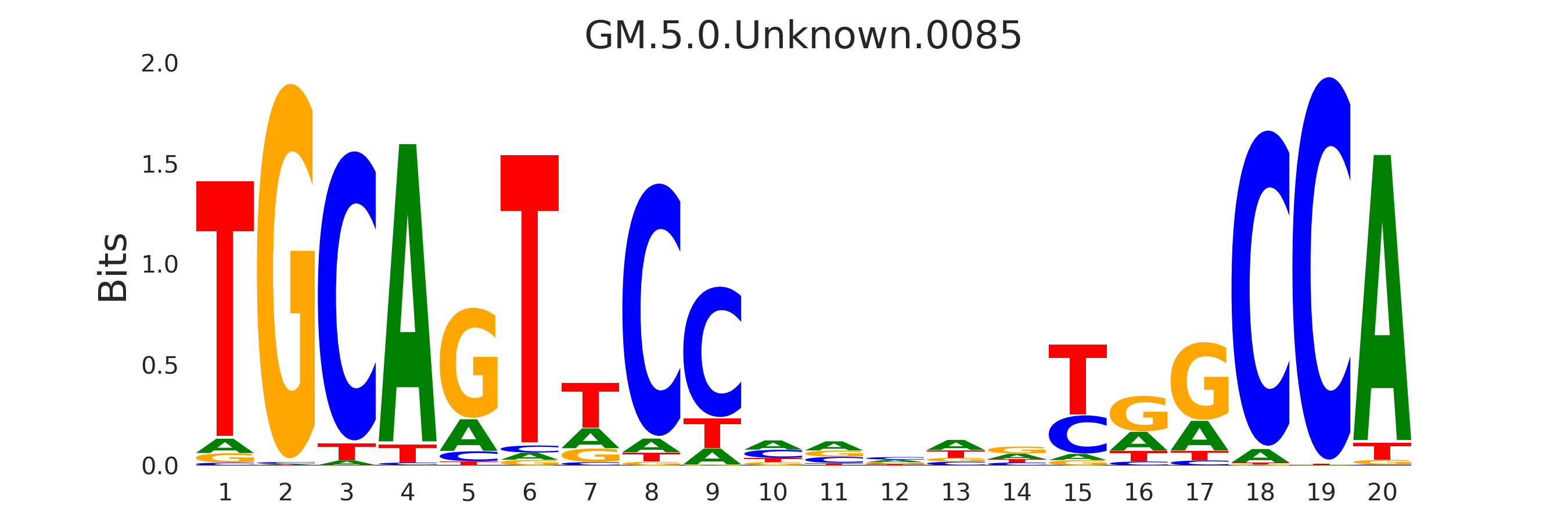
| GM.5.0.Unknown.0085 |
|
|
-3.24 |
-2.18 |
3.34 |
1 |
-0.10 |
-0.04 |
0.11 |
| GM.5.0.C2H2_ZF.0195 |
BCL11A |
|
-3.37 |
-5.42 |
4.65 |
5 |
-0.20 |
-0.18 |
0.27 |
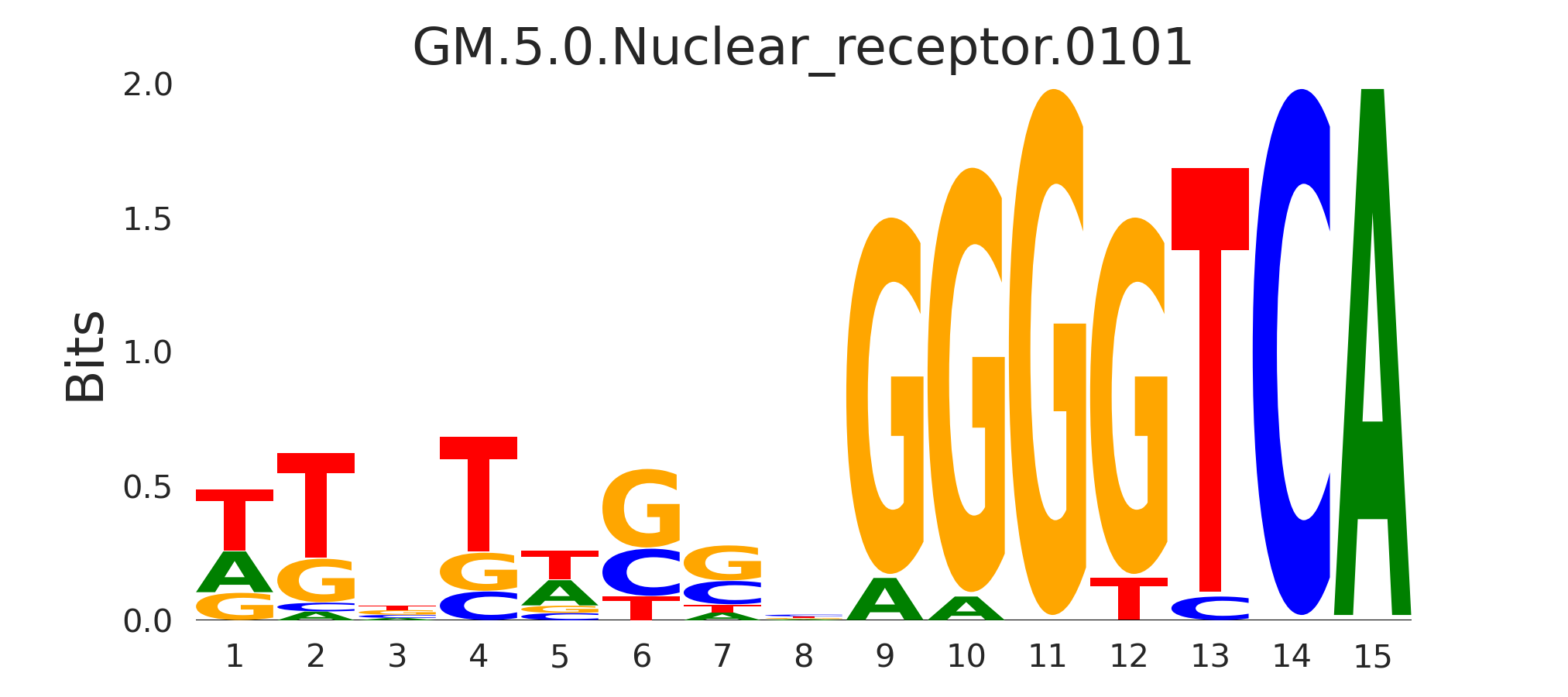
| GM.5.0.Nuclear_receptor.0101 |
HNF4G |
|
0.83 |
3.31 |
-1.76 |
<1 |
0.02 |
0.04 |
-0.03 |
| GM.5.0.Unknown.0091 |
|
|
4.66 |
3.51 |
-5.12 |
<1 |
0.19 |
-0.08 |
-0.13 |
| GM.5.0.Homeodomain.0158 |
ARX,ALX3,LHX2,LBX2,EN2, (...) |
|
0.95 |
3.26 |
-1.65 |
<1 |
0.06 |
-0.06 |
-0.02 |
| GM.5.0.MBD.0002 |
MECP2 |
|
3.40 |
1.74 |
-3.77 |
<1 |
0.16 |
-0.09 |
-0.10 |
| GM.5.0.Nuclear_receptor.0105 |
ESR1,RORB,ESR2,RORC |
|
3.08 |
-0.87 |
-1.60 |
<1 |
0.02 |
-0.04 |
-0.00 |
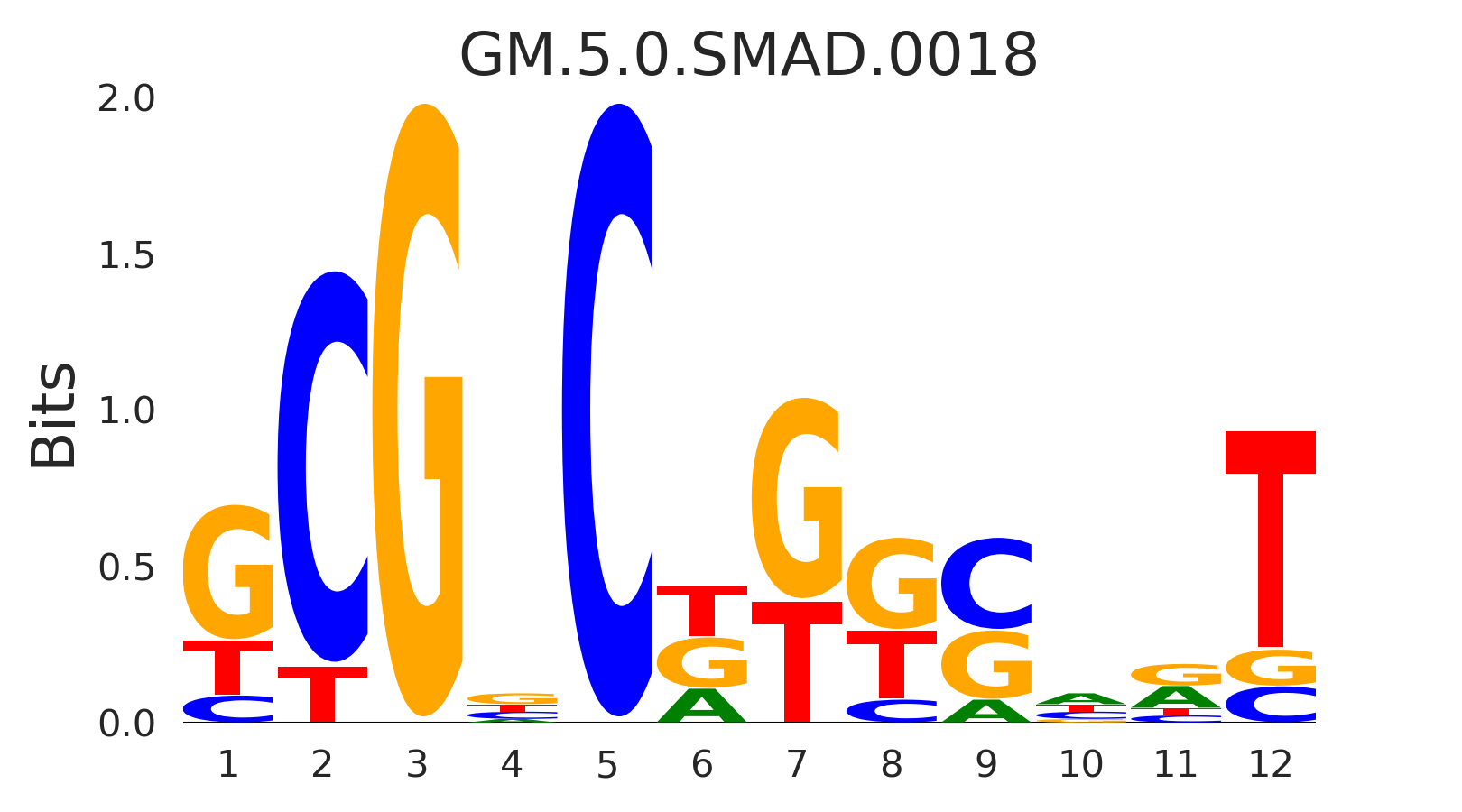
| GM.5.0.SMAD.0018 |
SMAD4 |
|
3.38 |
2.15 |
-3.66 |
2 |
0.11 |
-0.04 |
-0.08 |
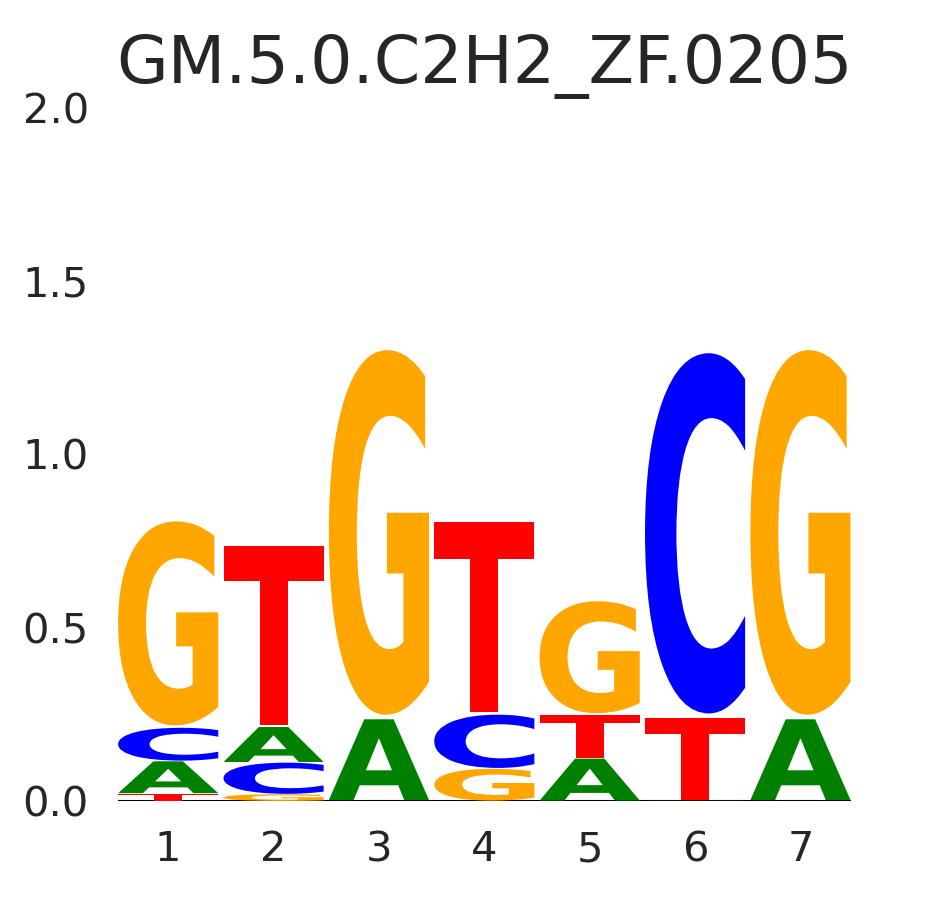
| GM.5.0.C2H2_ZF.0205 |
ZFP3 |
|
3.96 |
-2.80 |
-3.16 |
<1 |
0.13 |
-0.05 |
-0.10 |
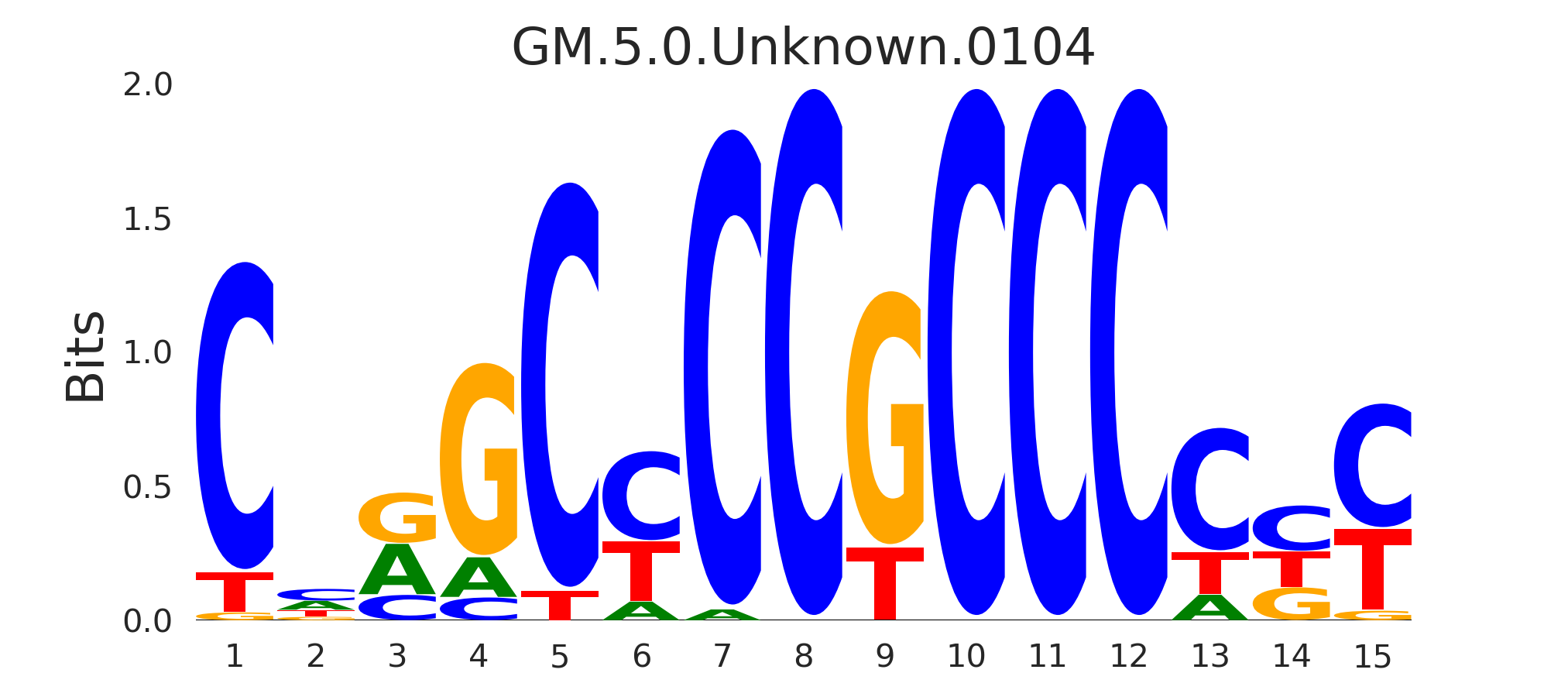
| GM.5.0.Unknown.0104 |
NRF1 |
|
-3.49 |
-0.39 |
3.44 |
<1 |
-0.08 |
-0.03 |
0.09 |
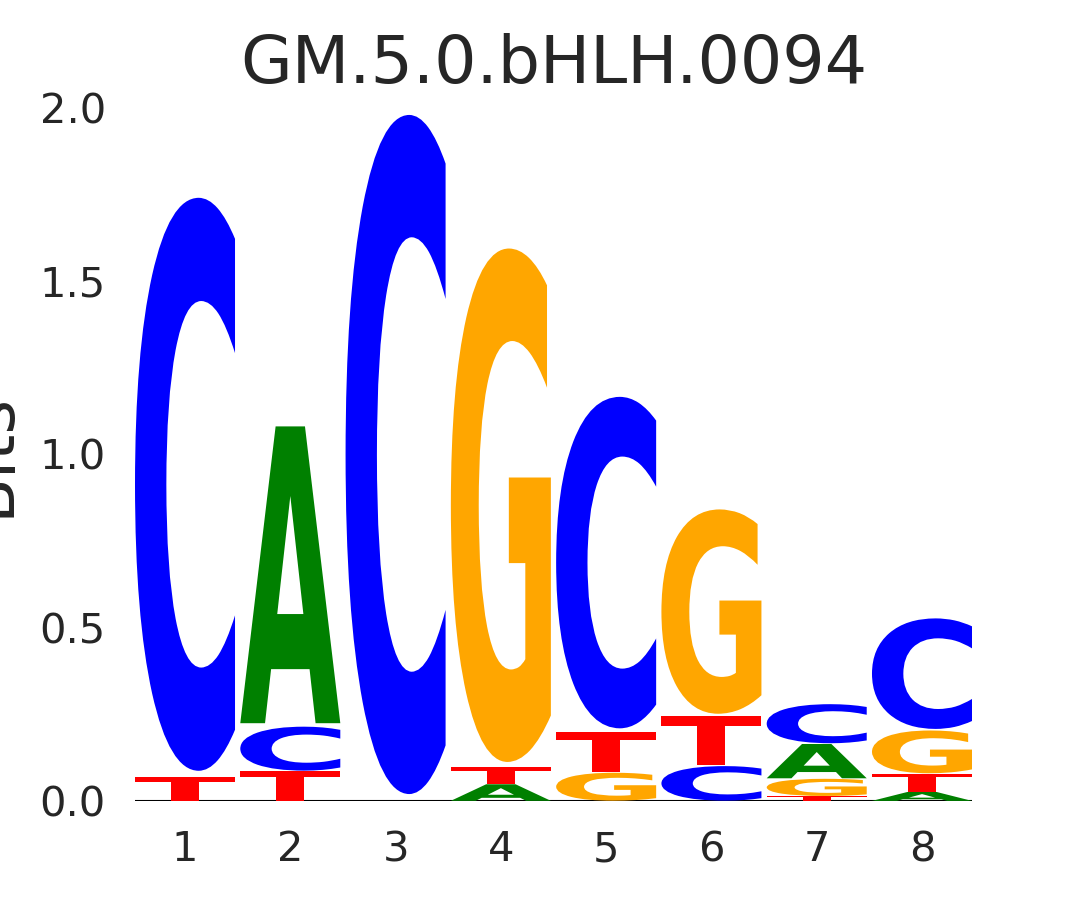
| GM.5.0.bHLH.0094 |
HES4 |
|
3.28 |
3.12 |
-3.81 |
<1 |
0.19 |
-0.03 |
-0.16 |
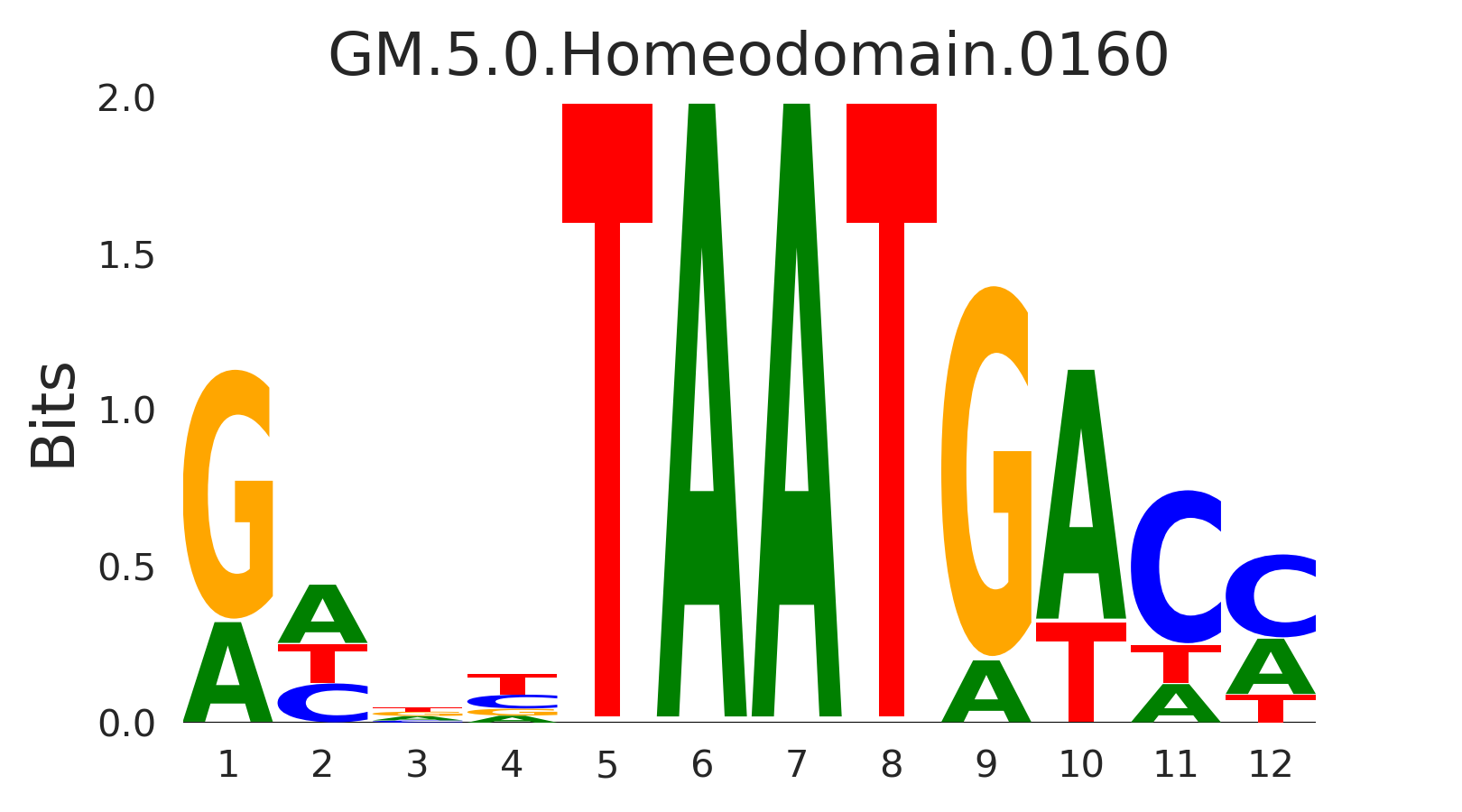
| GM.5.0.Homeodomain.0160 |
PDX1 |
|
1.24 |
3.10 |
-1.99 |
<1 |
0.04 |
-0.04 |
-0.02 |
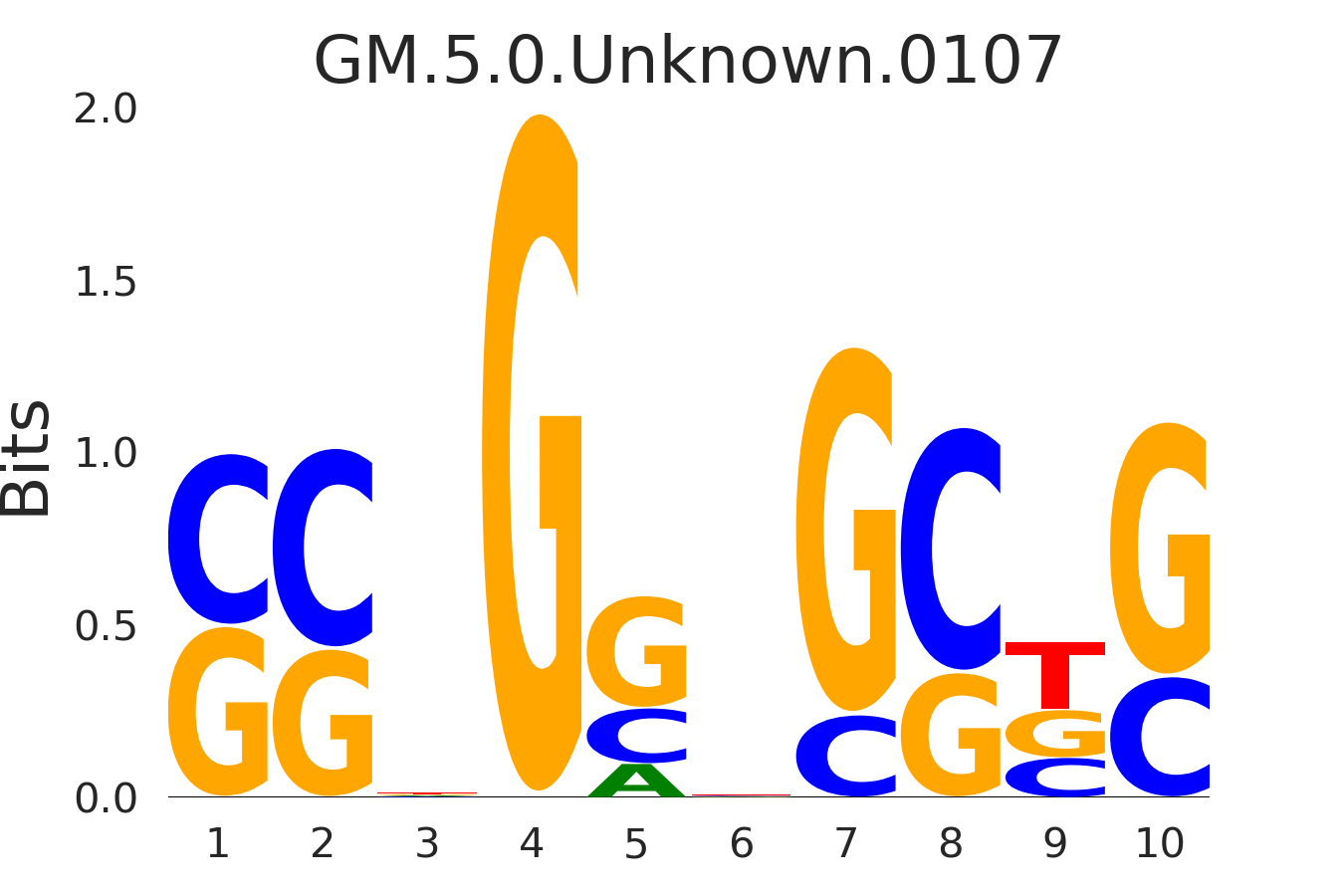
| GM.5.0.Unknown.0107 |
SIN3A |
|
1.90 |
-3.31 |
-0.77 |
<1 |
0.06 |
-0.14 |
0.01 |
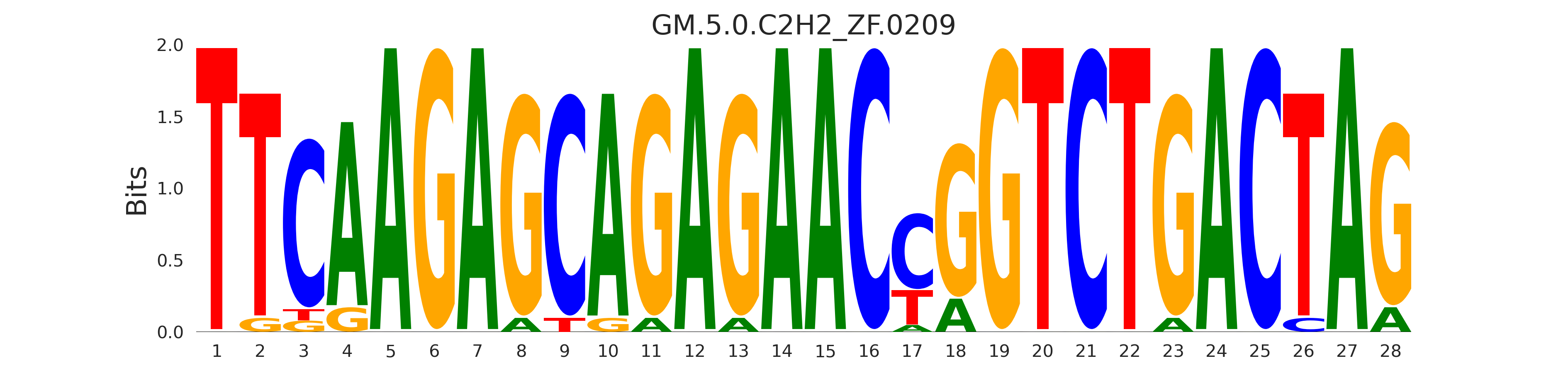
| GM.5.0.C2H2_ZF.0209 |
BCL11A |
|
-3.17 |
-2.95 |
3.98 |
7 |
-0.23 |
-0.14 |
0.28 |
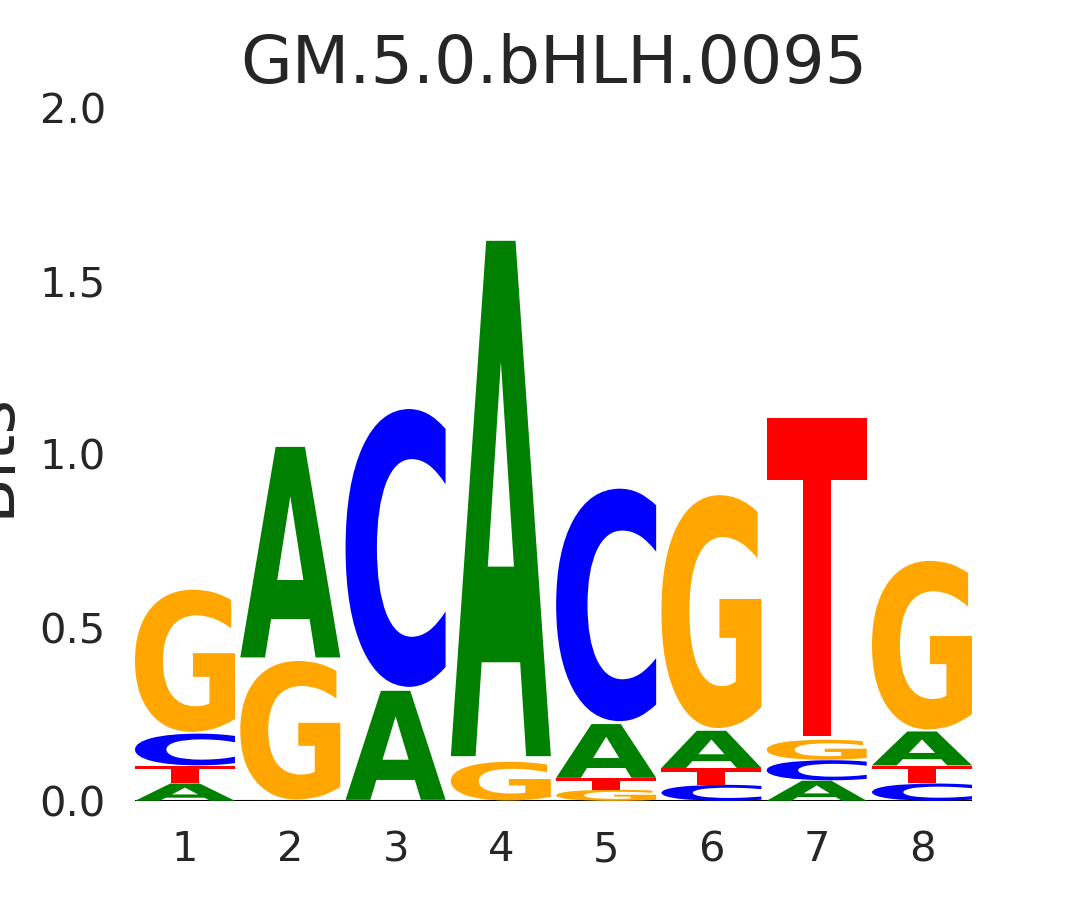
| GM.5.0.bHLH.0095 |
HES2,HES4 |
|
3.31 |
-1.24 |
-3.23 |
1 |
0.07 |
-0.04 |
-0.05 |
| GM.5.0.Unknown.0113 |
POLR3G,GTF3C2,POLR3A |
|
1.67 |
3.56 |
-2.18 |
<1 |
0.08 |
0.01 |
-0.07 |
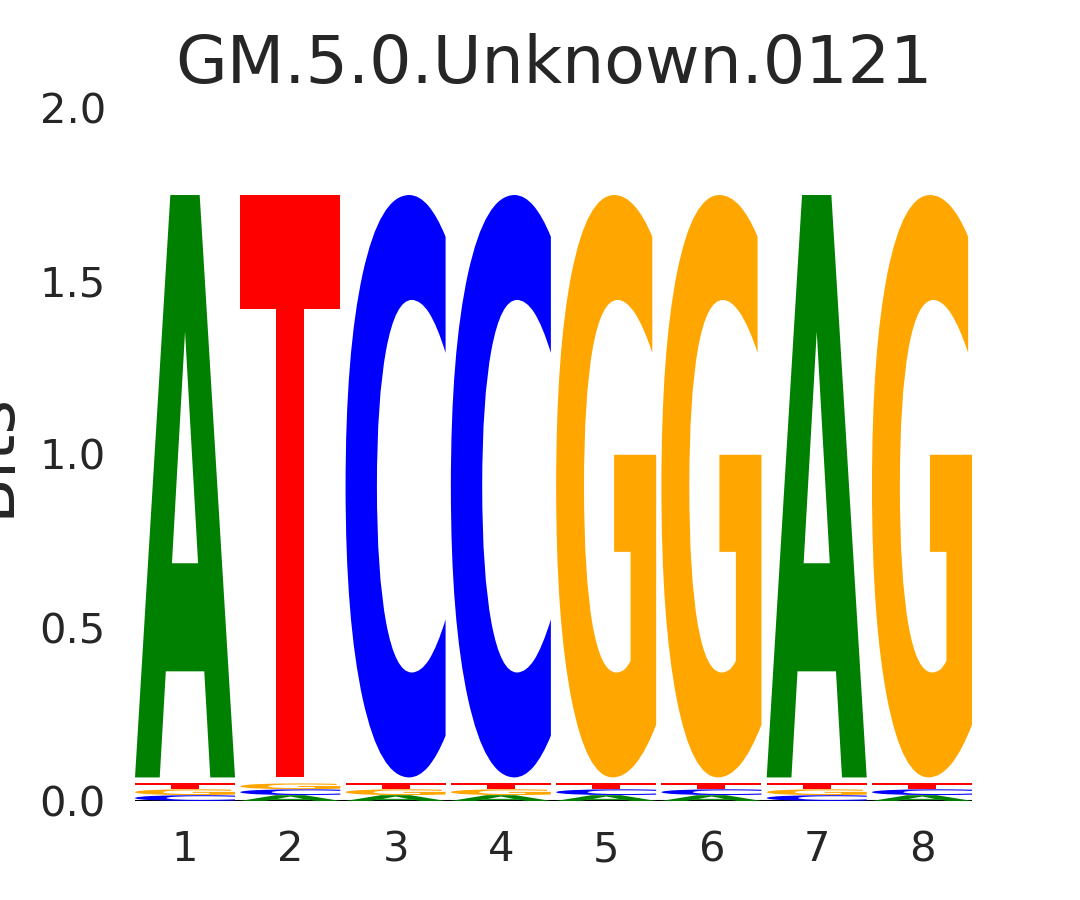
| GM.5.0.Unknown.0121 |
|
|
3.19 |
1.05 |
-2.72 |
22 |
0.08 |
-0.05 |
-0.05 |
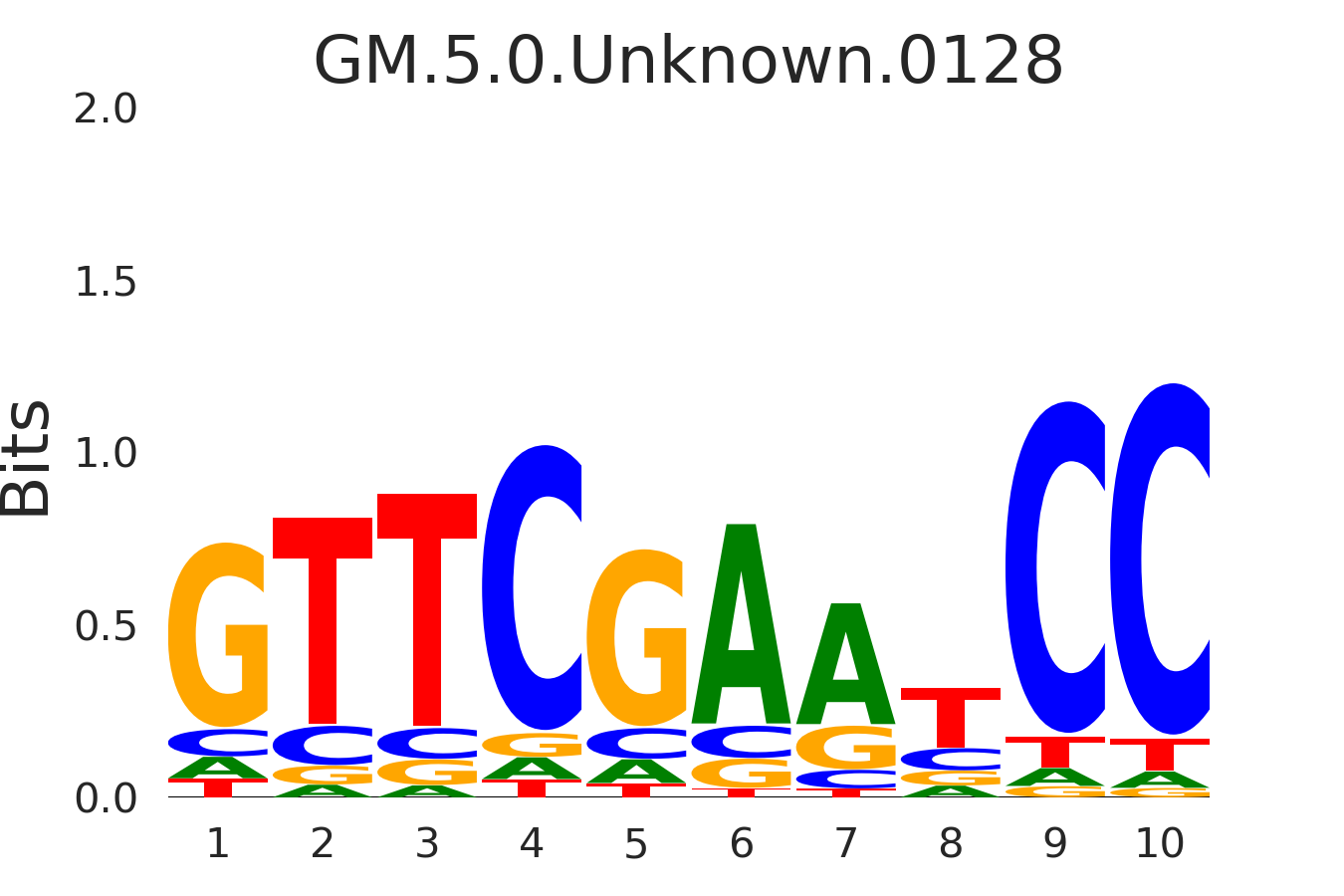
| GM.5.0.Unknown.0128 |
GTF3C2 |
|
4.40 |
-2.03 |
-4.17 |
2 |
0.12 |
0.04 |
-0.12 |
| GM.5.0.Unknown.0132 |
|
|
-3.22 |
-0.48 |
3.10 |
<1 |
0.00 |
-0.09 |
0.04 |
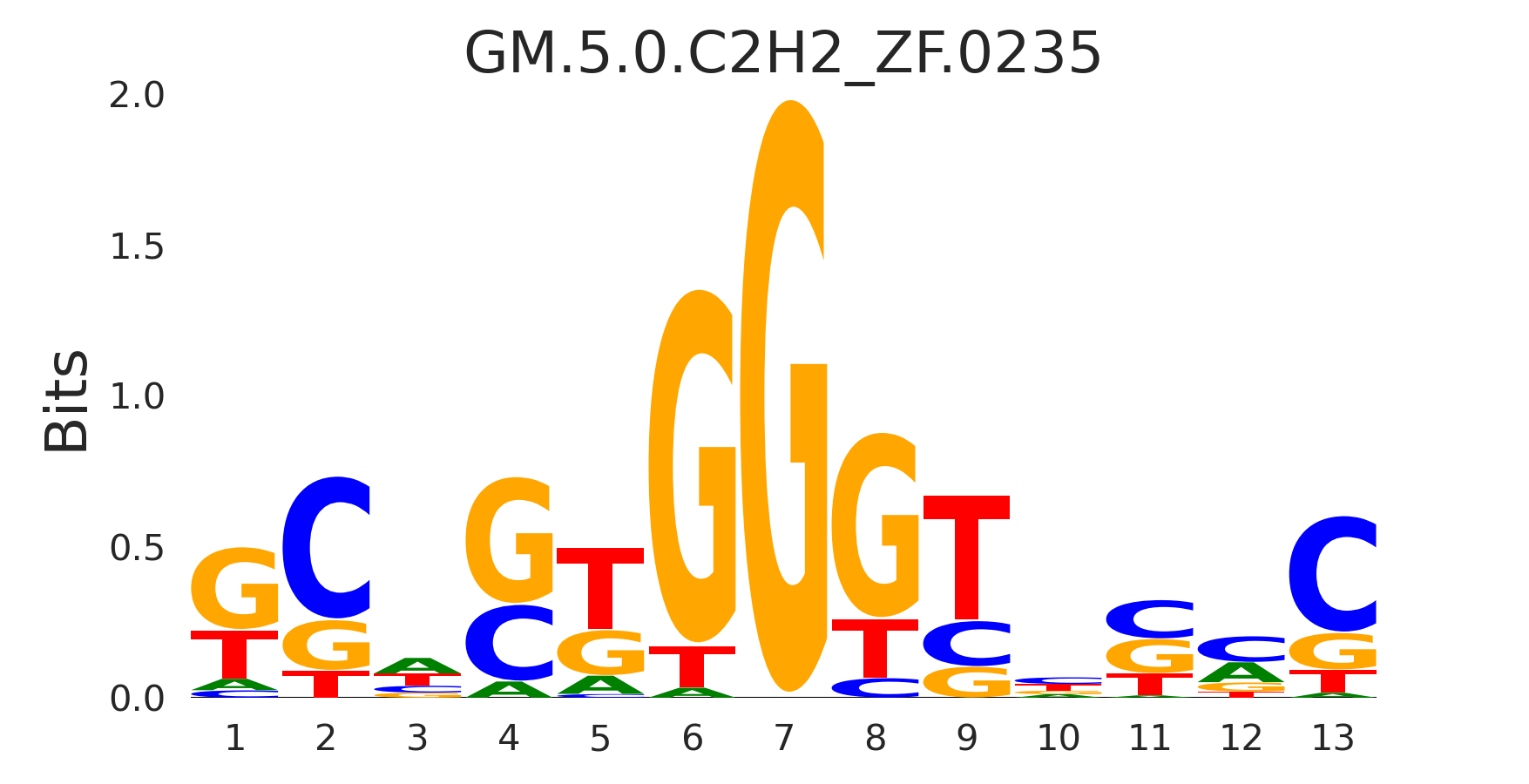
| GM.5.0.C2H2_ZF.0235 |
ZBTB7A,ZBTB7C |
|
3.62 |
2.81 |
-3.82 |
<1 |
0.01 |
-0.05 |
0.01 |
| GM.5.0.bZIP.0088 |
CEBPB |
|
-3.08 |
2.32 |
2.44 |
<1 |
-0.01 |
-0.02 |
0.02 |
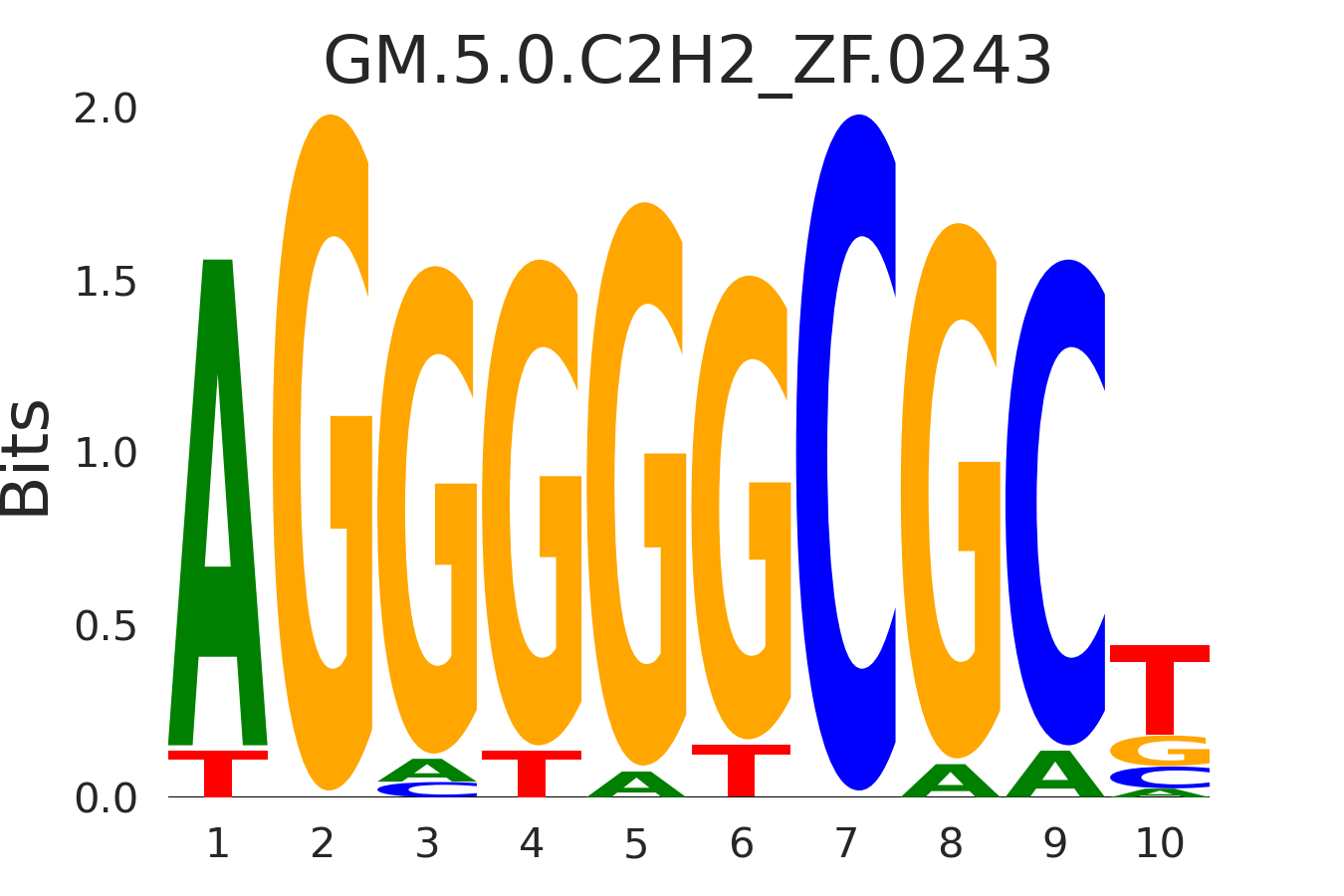
| GM.5.0.C2H2_ZF.0243 |
ZBTB7A |
|
-3.14 |
1.83 |
2.72 |
2 |
-0.00 |
-0.08 |
0.04 |
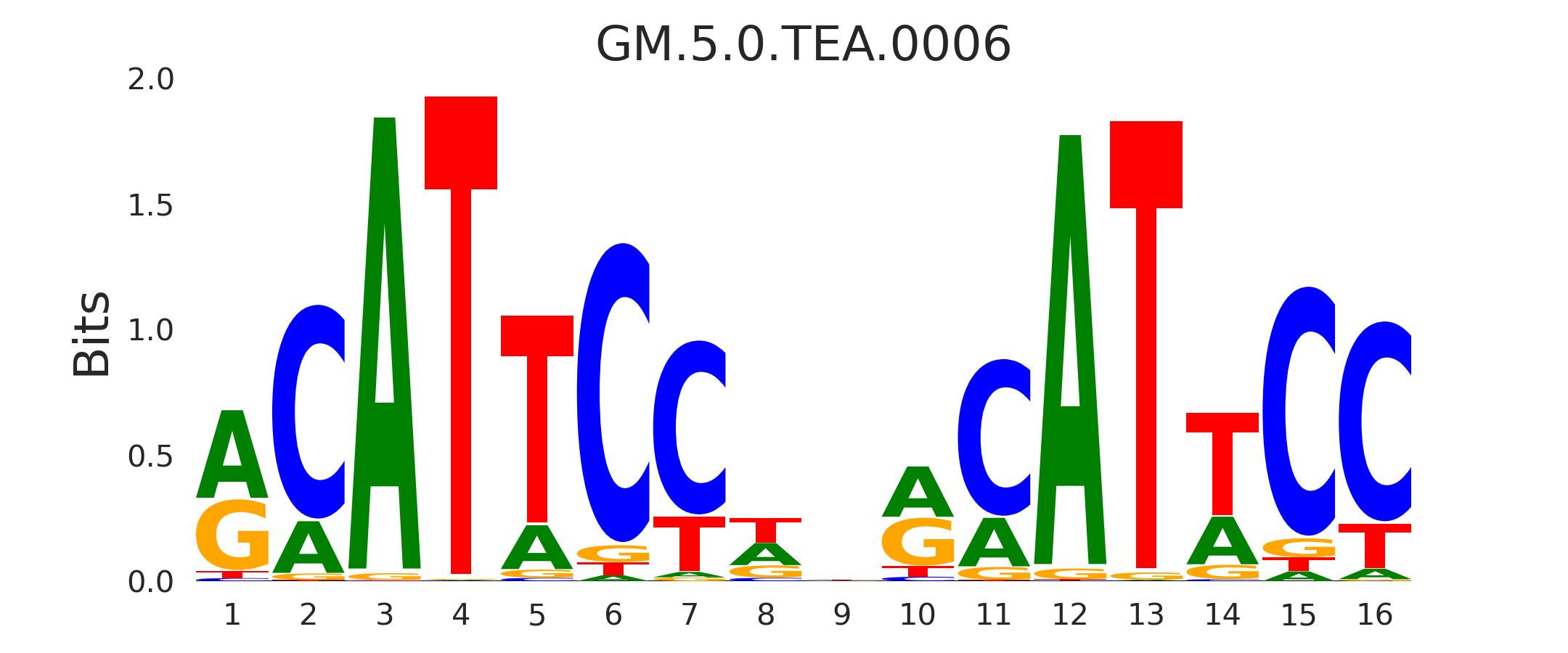
| GM.5.0.TEA.0006 |
TEAD3,TEAD1 |
|
-3.27 |
1.11 |
3.22 |
2 |
-0.08 |
0.01 |
0.07 |
| GM.5.0.Unknown.0157 |
HDAC2 |
|
0.88 |
-3.41 |
1.58 |
1 |
0.05 |
-0.09 |
0.00 |
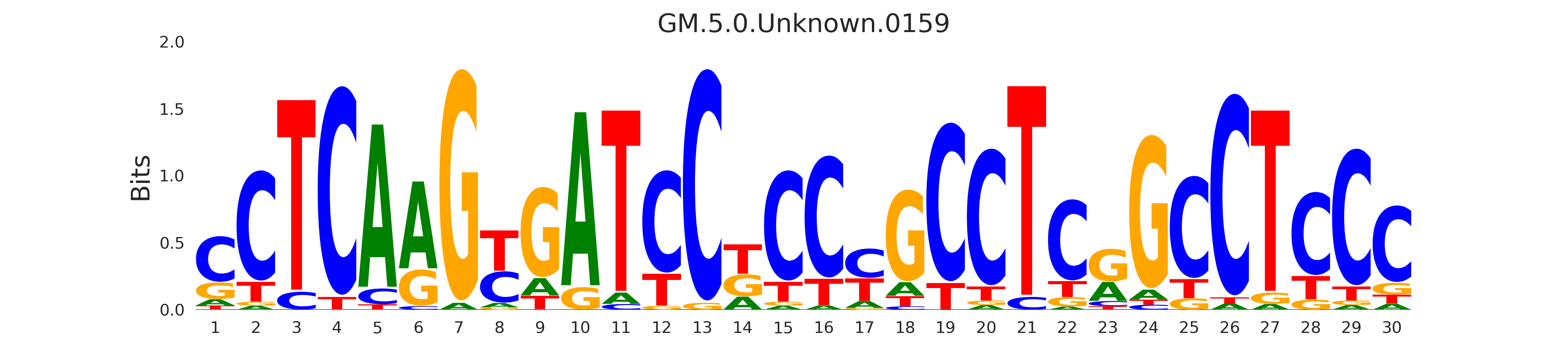
| GM.5.0.Unknown.0159 |
POLR3G,GTF3C2,POLR3A |
|
-3.22 |
2.85 |
2.35 |
<1 |
0.07 |
-0.00 |
-0.06 |
| GM.5.0.GATA.0026 |
GATA3 |
|
0.84 |
-3.03 |
1.72 |
<1 |
-0.05 |
-0.15 |
0.13 |
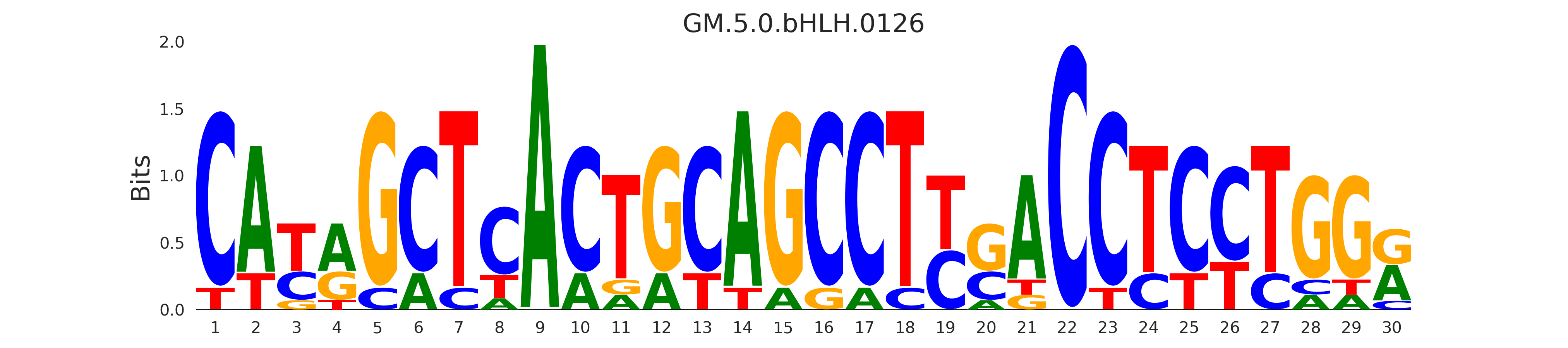
| GM.5.0.bHLH.0126 |
SREBF1 |
|
-1.61 |
3.17 |
-0.77 |
<1 |
0.01 |
0.02 |
-0.02 |
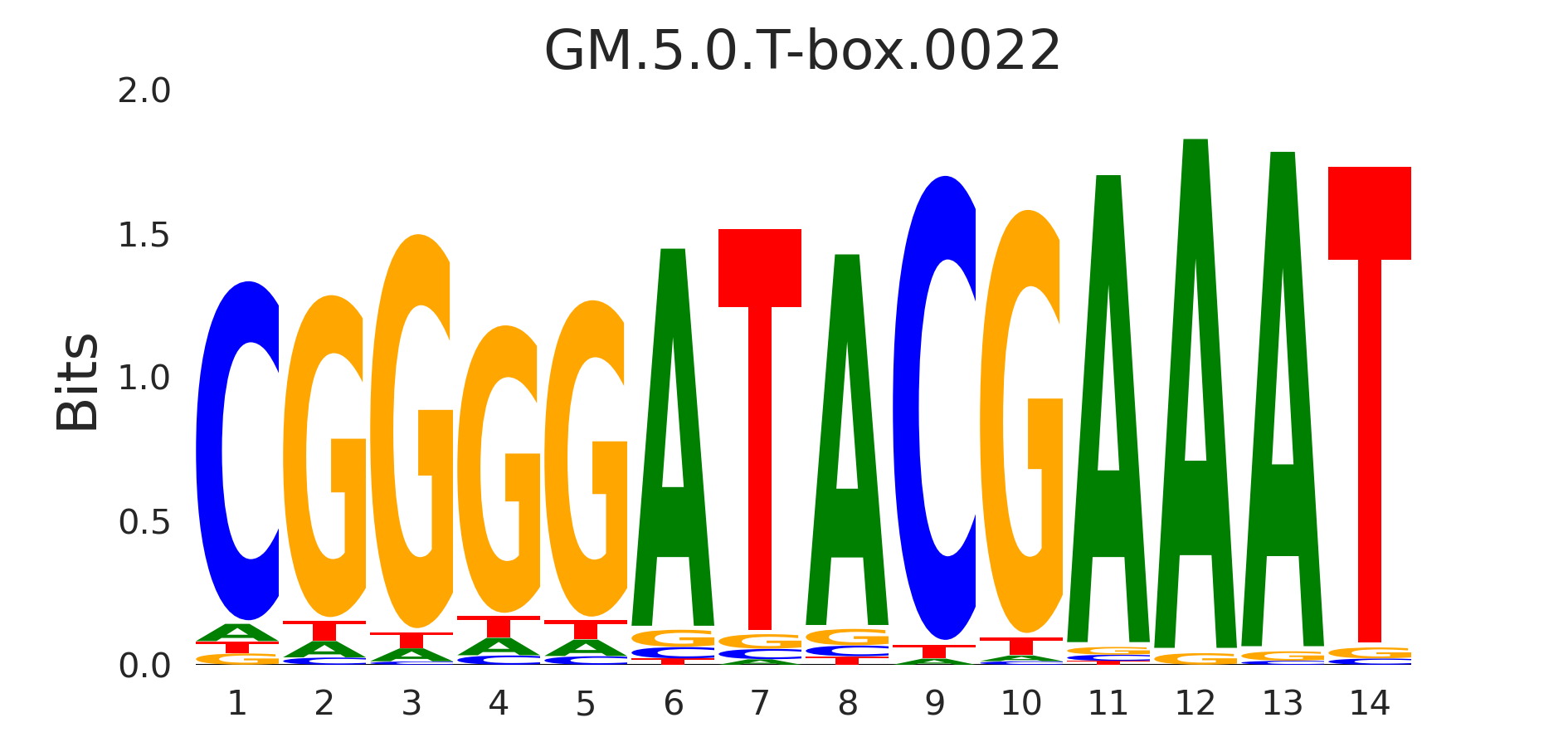
| GM.5.0.T-box.0022 |
EOMES,TBX21 |
|
4.07 |
-1.43 |
-4.01 |
1 |
0.11 |
-0.06 |
-0.07 |
| GM.5.0.C2H2_ZF.0258 |
YY1 |
|
3.24 |
-0.39 |
-3.28 |
<1 |
0.11 |
-0.10 |
-0.05 |
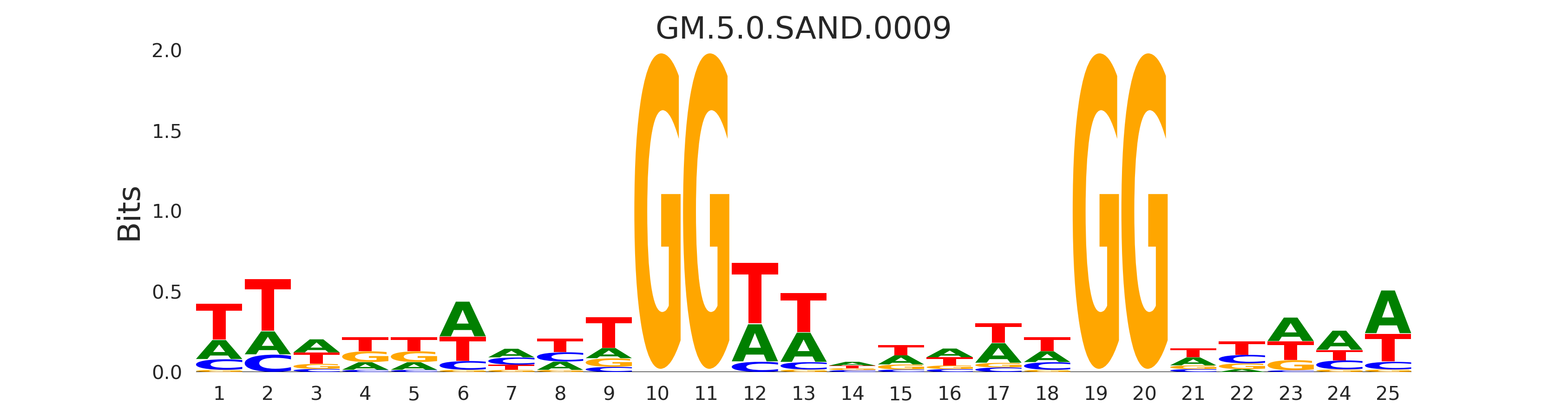
| GM.5.0.SAND.0009 |
AIRE |
|
0.93 |
3.01 |
-1.62 |
<1 |
0.04 |
0.01 |
-0.05 |
| GM.5.0.C2H2_ZF.0260 |
YY1 |
|
-3.11 |
2.55 |
2.63 |
1 |
-0.06 |
-0.08 |
0.10 |
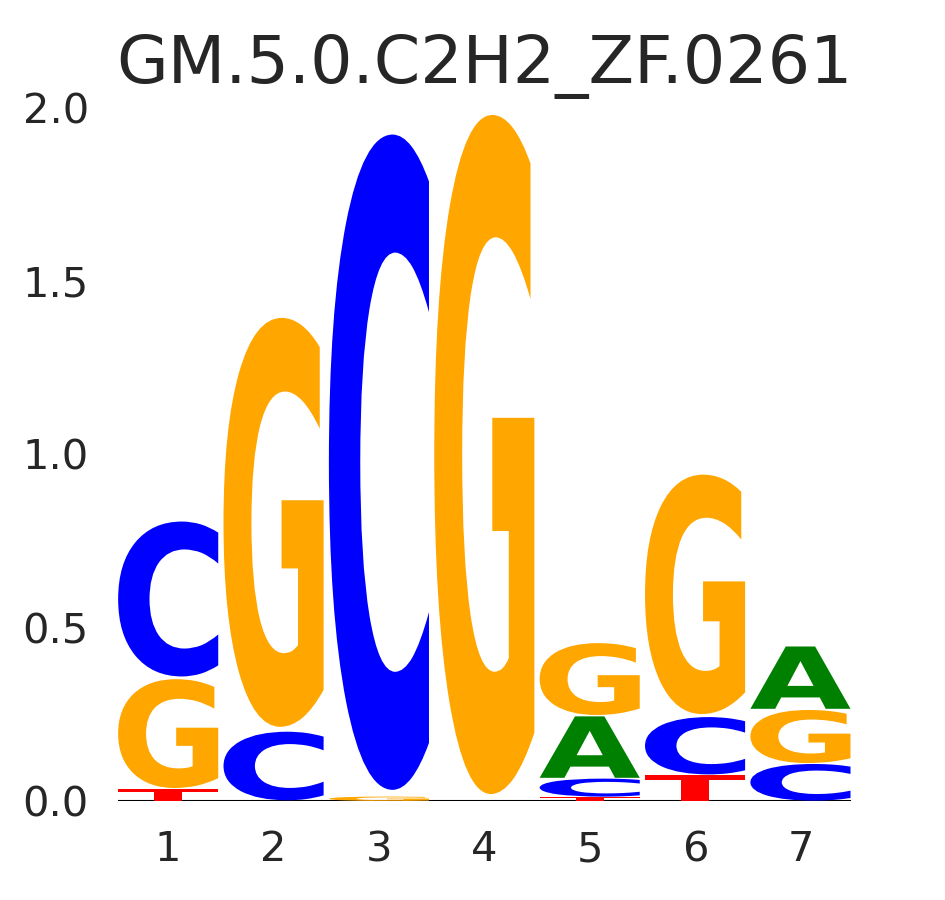
| GM.5.0.C2H2_ZF.0261 |
ZBTB33 |
|
2.24 |
3.14 |
-3.01 |
<1 |
0.17 |
-0.10 |
-0.11 |
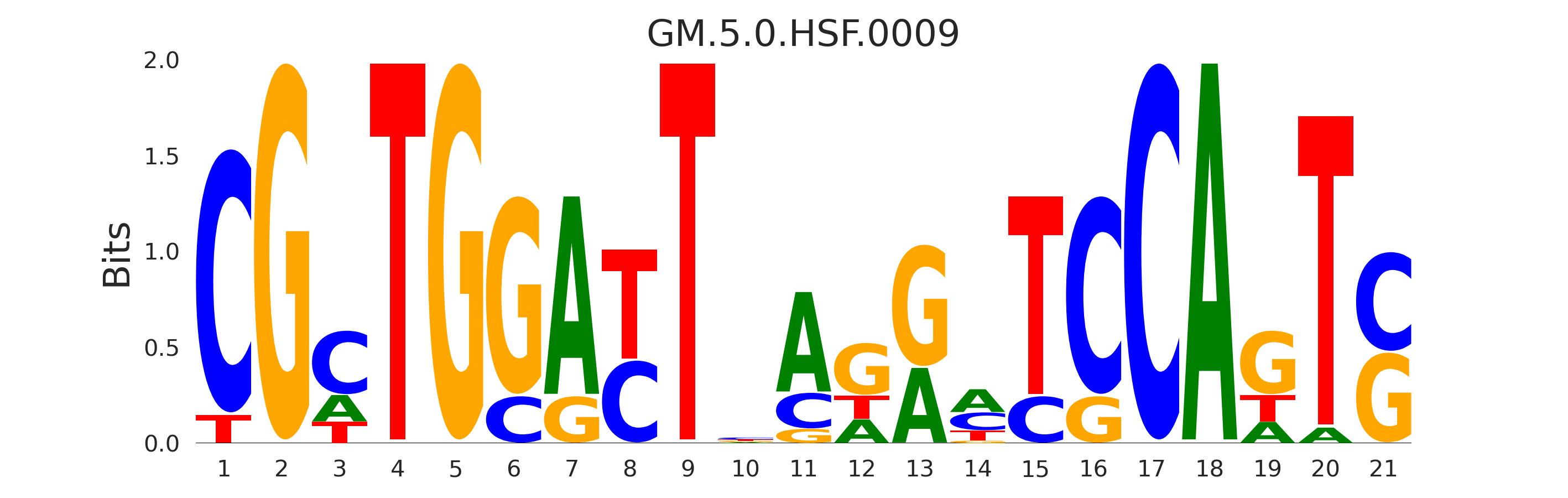
| GM.5.0.HSF.0009 |
HSF1 |
|
3.73 |
2.27 |
-3.90 |
1 |
0.00 |
0.03 |
-0.02 |
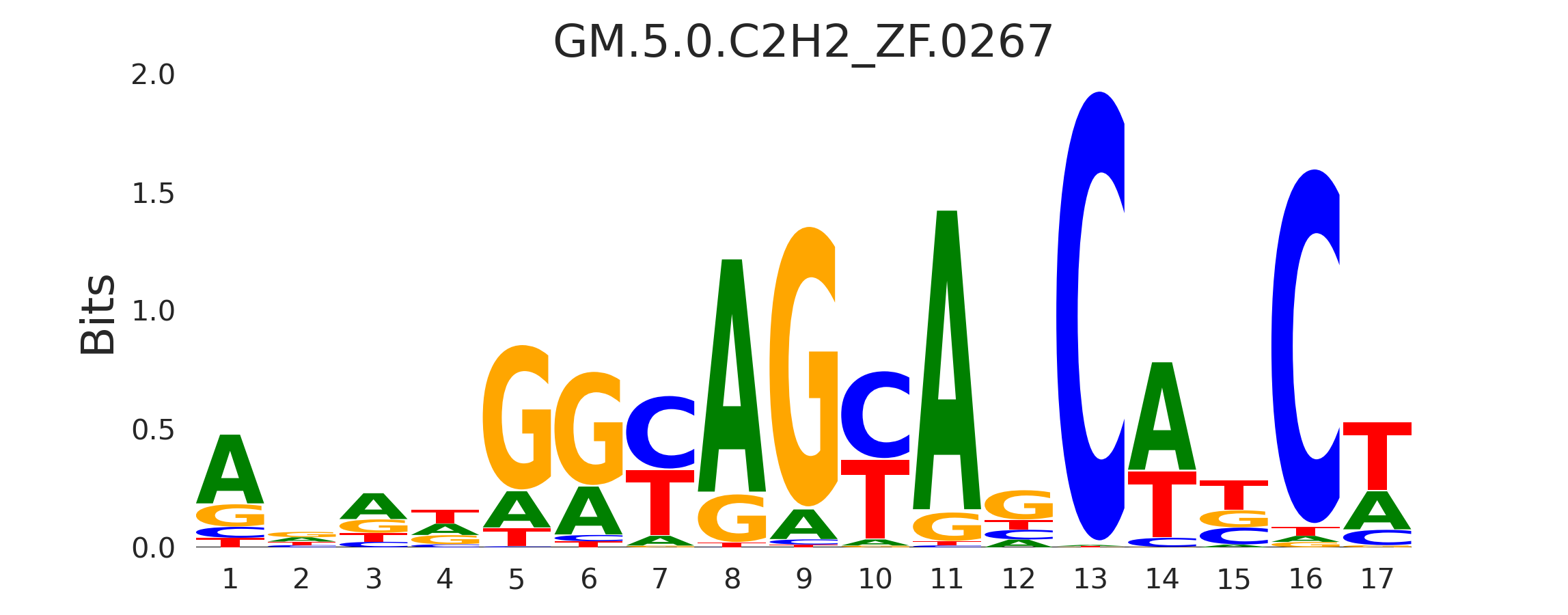
| GM.5.0.C2H2_ZF.0267 |
PRDM9 |
|
-2.17 |
3.57 |
0.62 |
<1 |
-0.01 |
0.03 |
-0.00 |
| GM.5.0.bHLH.0128 |
HES1,HES2,HES4 |
|
3.40 |
-1.32 |
-2.66 |
1 |
0.12 |
-0.04 |
-0.09 |
| GM.5.0.RFX.0015 |
ARID2,RFX8,RFX6,RFX5,RFX7 |
|
-3.29 |
-3.29 |
3.49 |
2 |
-0.03 |
-0.06 |
0.06 |
| GM.5.0.Nuclear_receptor.0129 |
AR |
|
0.03 |
4.06 |
-1.39 |
1 |
0.03 |
0.03 |
-0.04 |
| GM.5.0.Unknown.0167 |
ZBTB14 |
|
3.80 |
-2.73 |
-2.94 |
<1 |
0.21 |
-0.10 |
-0.14 |
| GM.5.0.C2H2_ZF.0275 |
ZSCAN10 |
|
-3.84 |
-0.79 |
3.71 |
<1 |
-0.04 |
-0.03 |
0.05 |
| GM.5.0.GATA.0029 |
GATA1,GATA2 |
|
-5.48 |
4.99 |
4.98 |
<1 |
-0.13 |
0.00 |
0.11 |
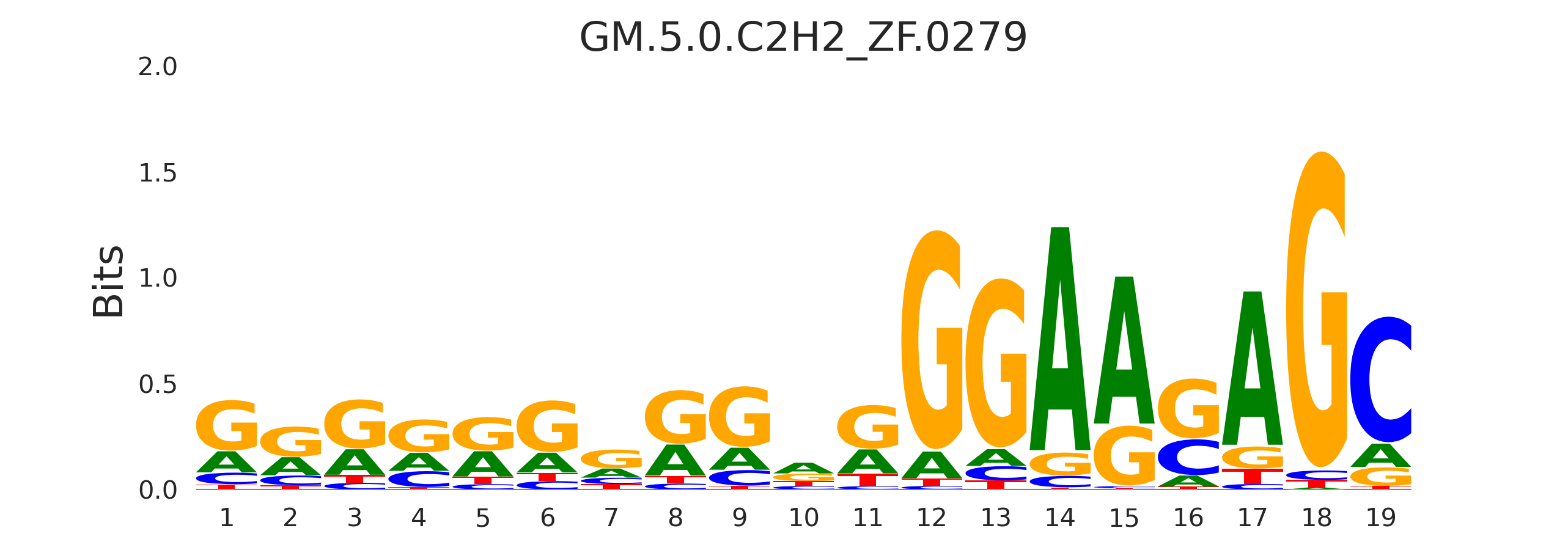
| GM.5.0.C2H2_ZF.0279 |
ZNF341 |
|
-2.69 |
-2.81 |
3.06 |
1 |
-0.06 |
-0.06 |
0.08 |
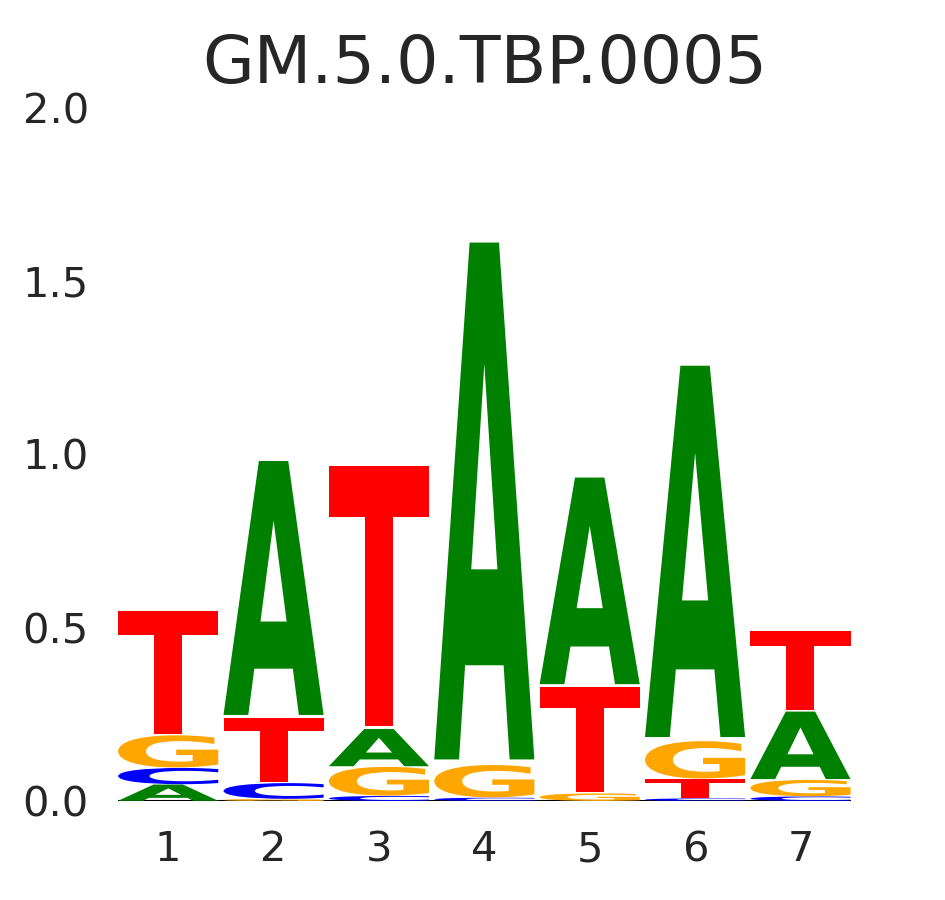
| GM.5.0.TBP.0005 |
TBPL2,TBP |
|
2.46 |
3.13 |
-2.46 |
<1 |
0.11 |
-0.09 |
-0.06 |
| GM.5.0.C2H2_ZF.0288 |
SP6,SP9,SP7,SP5,SP3, (...) |
|
-4.91 |
-1.38 |
4.91 |
<1 |
-0.16 |
-0.05 |
0.17 |
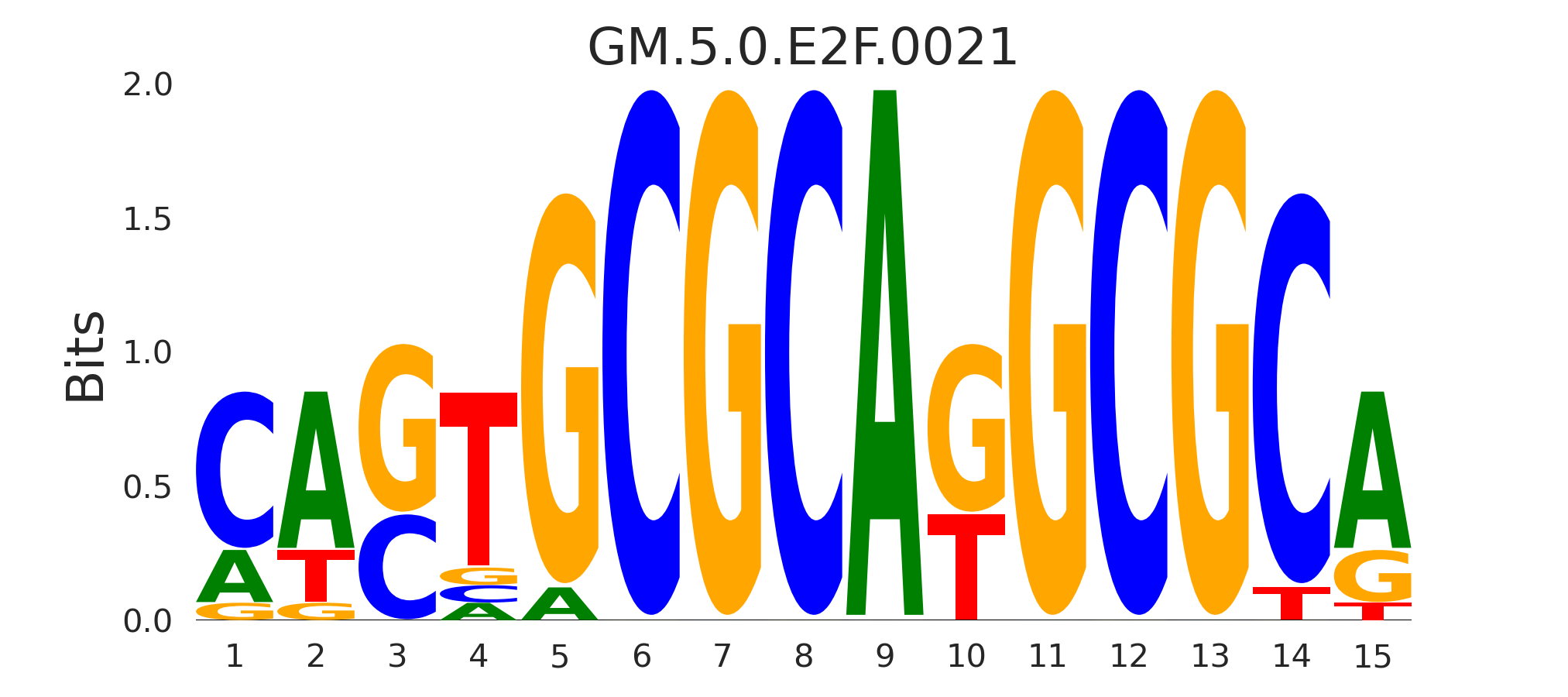
| GM.5.0.E2F.0021 |
E2F1 |
|
-3.16 |
-3.72 |
3.90 |
1 |
0.05 |
-0.09 |
0.00 |
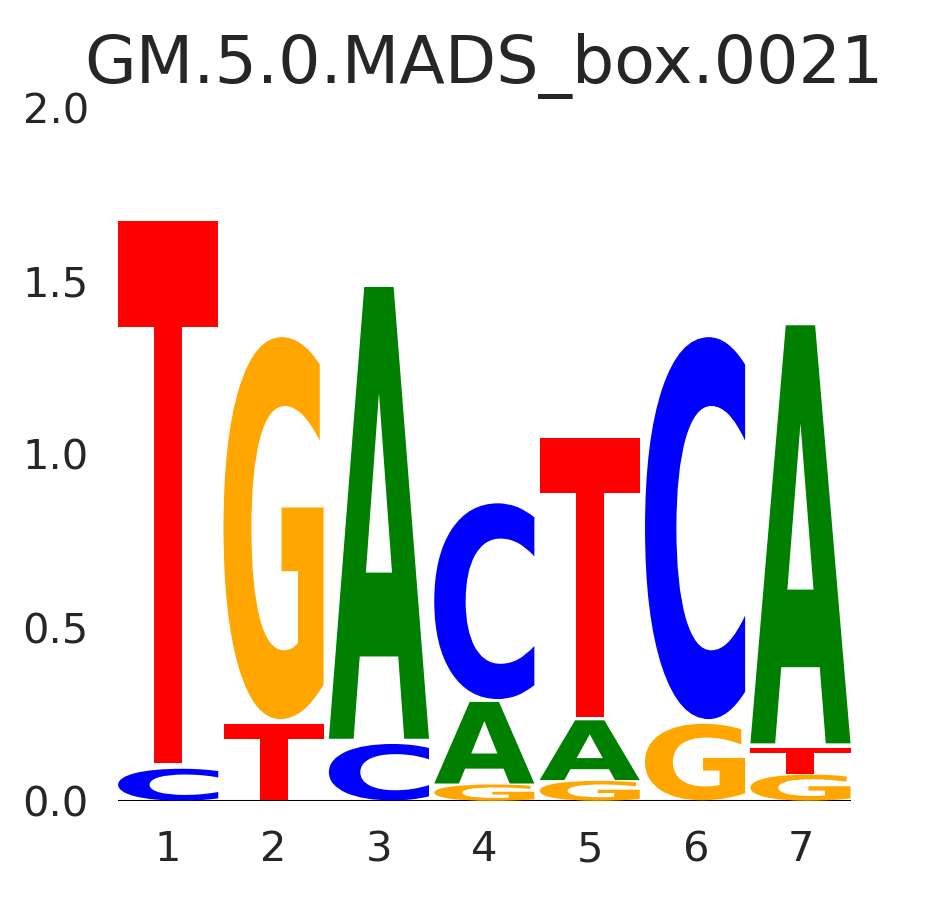
| GM.5.0.MADS_box.0021 |
AP1 |
|
-2.96 |
3.54 |
1.84 |
<1 |
-0.02 |
-0.00 |
0.02 |
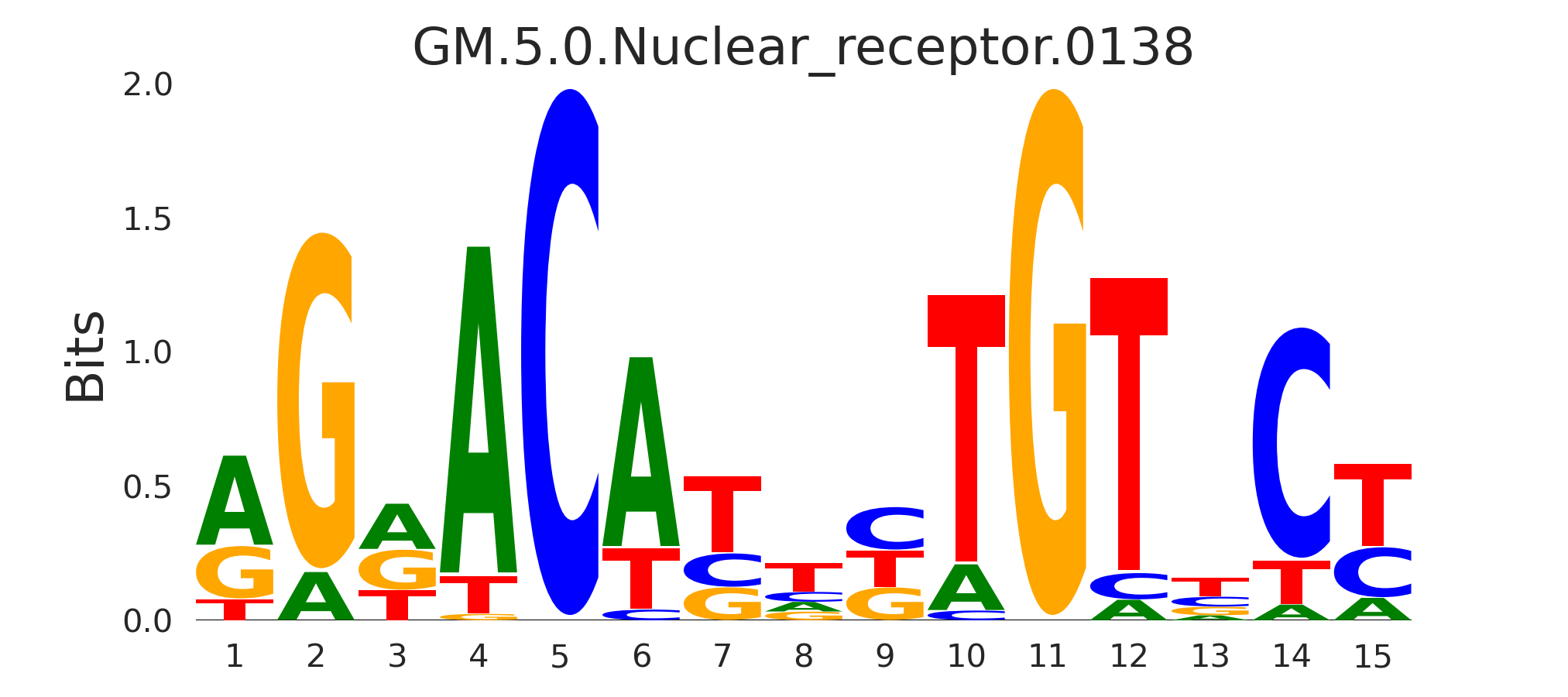
| GM.5.0.Nuclear_receptor.0138 |
NR3C1,NR3C2,PGR |
|
3.03 |
-0.98 |
-2.23 |
<1 |
0.05 |
-0.05 |
-0.02 |
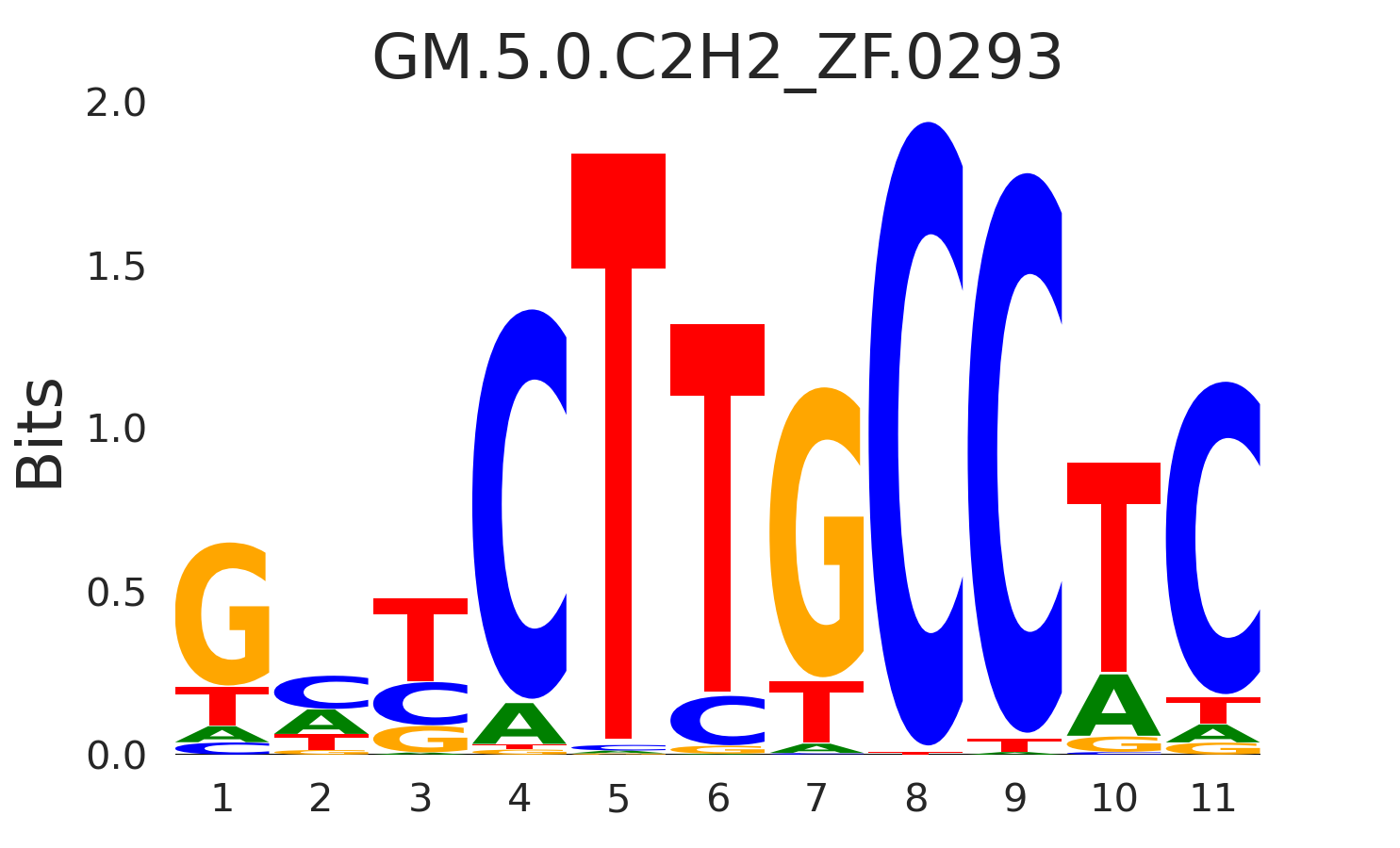
| GM.5.0.C2H2_ZF.0293 |
ZNF257 |
|
2.57 |
2.19 |
-3.16 |
<1 |
0.06 |
-0.02 |
-0.04 |
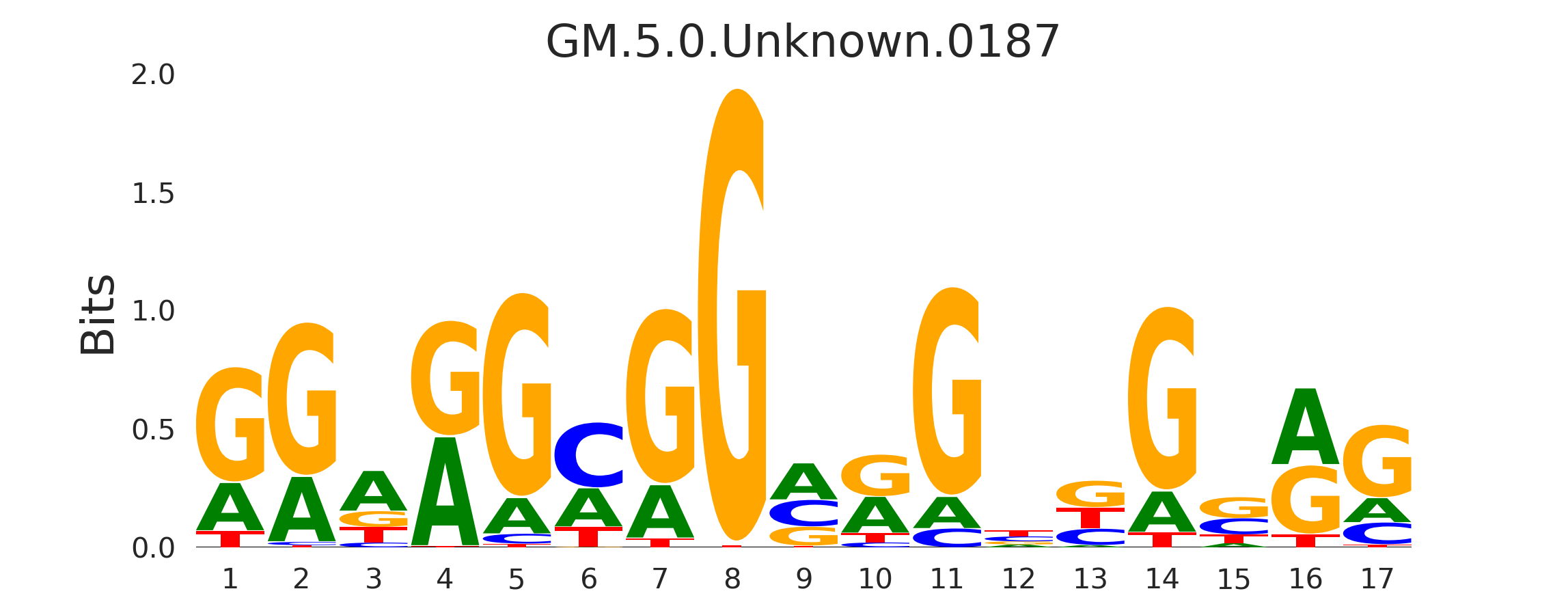
| GM.5.0.Unknown.0187 |
|
|
3.13 |
1.01 |
-3.01 |
<1 |
0.09 |
-0.02 |
-0.08 |
| GM.5.0.Ets.0046 |
SPDEF |
|
3.27 |
4.53 |
-4.22 |
<1 |
0.13 |
0.02 |
-0.13 |
| GM.5.0.C2H2_ZF.0300 |
E4F1 |
|
-3.35 |
1.57 |
2.76 |
1 |
-0.01 |
0.01 |
0.00 |
| GM.5.0.C2H2_ZF.0303 |
SP5,SP9,SP3,SP8 |
|
3.45 |
-1.59 |
-3.45 |
<1 |
0.14 |
-0.03 |
-0.11 |
| GM.5.0.Nuclear_receptor.0146 |
ESR1,ESR2 |
|
3.15 |
2.56 |
-3.41 |
<1 |
0.04 |
-0.03 |
-0.02 |
| GM.5.0.C2H2_ZF.0314 |
KLF5,KLF7,KLF3,KLF2,KLF6 |
|
-3.53 |
3.79 |
2.91 |
<1 |
-0.12 |
-0.03 |
0.13 |
| GM.5.0.RFX.0018 |
RFX3,RFX2,RFX5,RFX6,RFX4, (...) |
|
-3.58 |
1.78 |
3.30 |
2 |
-0.02 |
-0.04 |
0.04 |